Kaarten: Biochemie en moleculaire biologie: Kennisclip Antibiotica | Quizlet
1/17
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
18 Terms
Wat zijn antibiotica en waar werken ze tegen?
- geneesmiddelen dat bacteriële infecties bestrijdt
- werkt niet tegen virussen
Wat zijn de 2 manieren waarop antibiotica kan werken?
bactericide (doodt de bacterie)
bacteriostatica (remt de groei van de bacterie)
Wat zijn de 3 werkingsmechanismes van antibiotica? (geef telkens voorbeeld)
1) inhibitie van microbiale celwand synthese (peniciline)
2) inhibitie van DNA synthese functie (quinolone = blokkeert DNA gyrase (soort topo-isomerase))
3) Inhibitie van proteïne synthese (chloramphenicol, erythomycine, streptomycine, tetracycline)

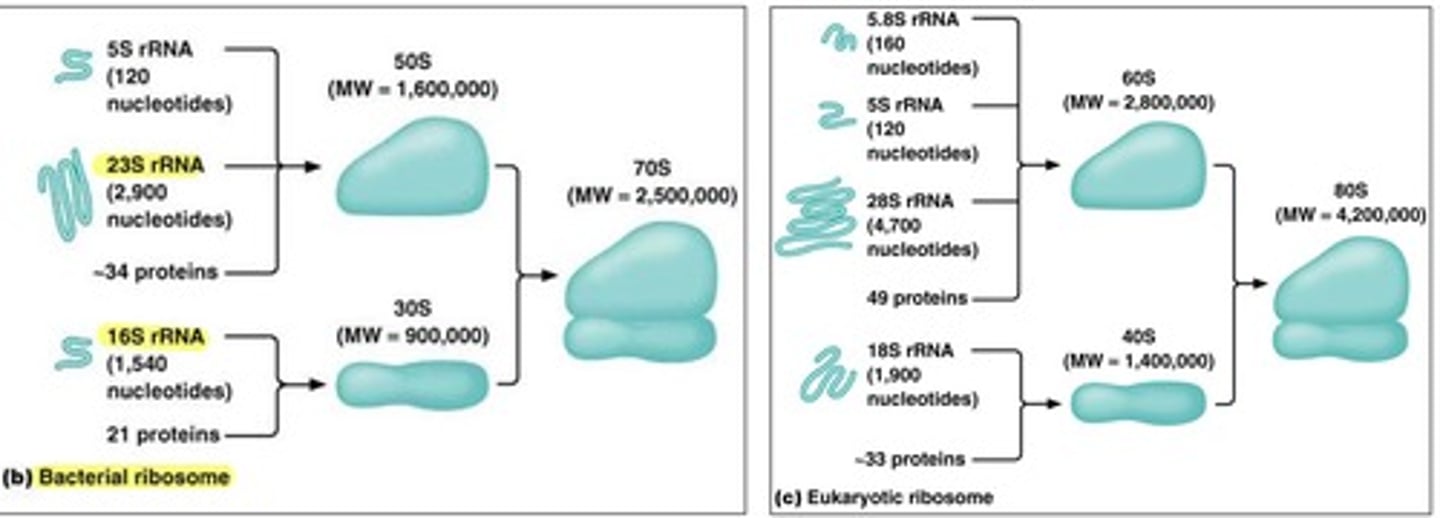
Hoe gaat streptomycine werken?
-bindt 16S rRNA van 30S (16S gaat samen met Shine-Delgarno hybridiseren zodat transcriptie kan beginnen)
= geen initiatie van transcriptie OF verkeerde vorming van initiatiecomplex waardoor het foute eiwit wordt gevormd
Hoe werkt Tetracycline?
- bindt A-plaats (amino-acyl) van 30S
= tRNA's kunnen niet binden dus de 1e stap van de prokaryotische translatie wordt geblokkeerd
Hoe werkt chloramphenicol?
- bindt aan de 23S (peptidyltransferase = katalyseren van de peptidebinding) rRNA van 50S
= geen peptidyltransferase dus de 2e stap van de elongatie in de prokaryotische translatie wordt geblokkeerd
Hoe werkt Erythromycine?
- bindt 23S rRNa van 50S
= geen translocatie van het ribosoom (het ribosoom kan geen plaats opschuiven waardoor de A-plaats bezet blijft en de eiwitsynthese stopt) de 3e stap van de prokaryotische translatie is geblokkeerd
Samenvattende tekening van de vorige antibiotica

Hoe komt het dat de translatie van de mens niet in het gedrang komt na het nemen van antibiotica?
De antibiotica werkt op rRNA's dat enkel aanwezig zijn in prokaryoten (23S, 16S) hierdoor gaan ze geen effect hebben op de eukaryotische cellen.
Wat is de definitie van een ziekenhuisbacterie?
Bacteriën dat resistent zijn tegen (bijna) alle antibiotica dat nu bestaan
Welke factoren dragen bij tot antibiotica resistentie?
- mutaties
- uitwisseling van genetisch materiaal tussen bacteriën
- overmatig en ondoordacht gebruik van antibiotica
Hoe gaan deze mutaties bijdragen tot antibiotica resistentie?
- Bacteriën delen om 20 min
- Ze maken 1 fout/ 10^9
= na 10u: 2^30 bacterien en 10^6 mutaties
Hoe gaan uitwisselingen van genetisch materiaal bijdragen tot antibiotica resistentie in bacteriën?
Bacteriën gaan aan uitwisseling doen ahv 3 mechanismes:
1) conjugatie
2) transformatie
3) transductie
(Transposons en plasmide zorgen voor makkelijke overdracht van resistentie genen)
Dit proces wordt gevoed door onderdoordacht en overmatig gebruik van antibiotica
Mechanisme antibiotica resistentie van streptomycine
Mutatie in 16S rRNa waardoor de bindingsplaats verandert en streptomycine niet meer kan binden
Mechanisme antibiotica resistentie van Tetracycline
RPPs (ribosomal protection proteïns) gelijkend op EF-Ty/EF-G -, ook GTPase
Ze gaan binden op de A-plaats waardoor Tetracycline gaan loskomen. Hierna gaat GTP hydrolyseren waarna ze zelf loskomen en translatie verder kan gaan
Het RPP gen ligt in transposons waardoor deze makkelijk wordt verspreid
Mechanisme antibiotica resistentie van Chloramphenicol
cat-gen: chloramphenicol acetyltransferase
Gaat 2 acetylgroepen binden op chloramphenicol waardoor deze niet meer kan binden op het 23S rRNa
Het cat-gen ligt op plasmide (gemakkelijk te verspreiden)
2 Mechanismes antibiotica resistentie van Erythromycine
1) mutatie in 23S rRNa waardoor Erythromycine niet kan binden
2) methylering van 23S door methylase
het methylase gen ligt in transposons

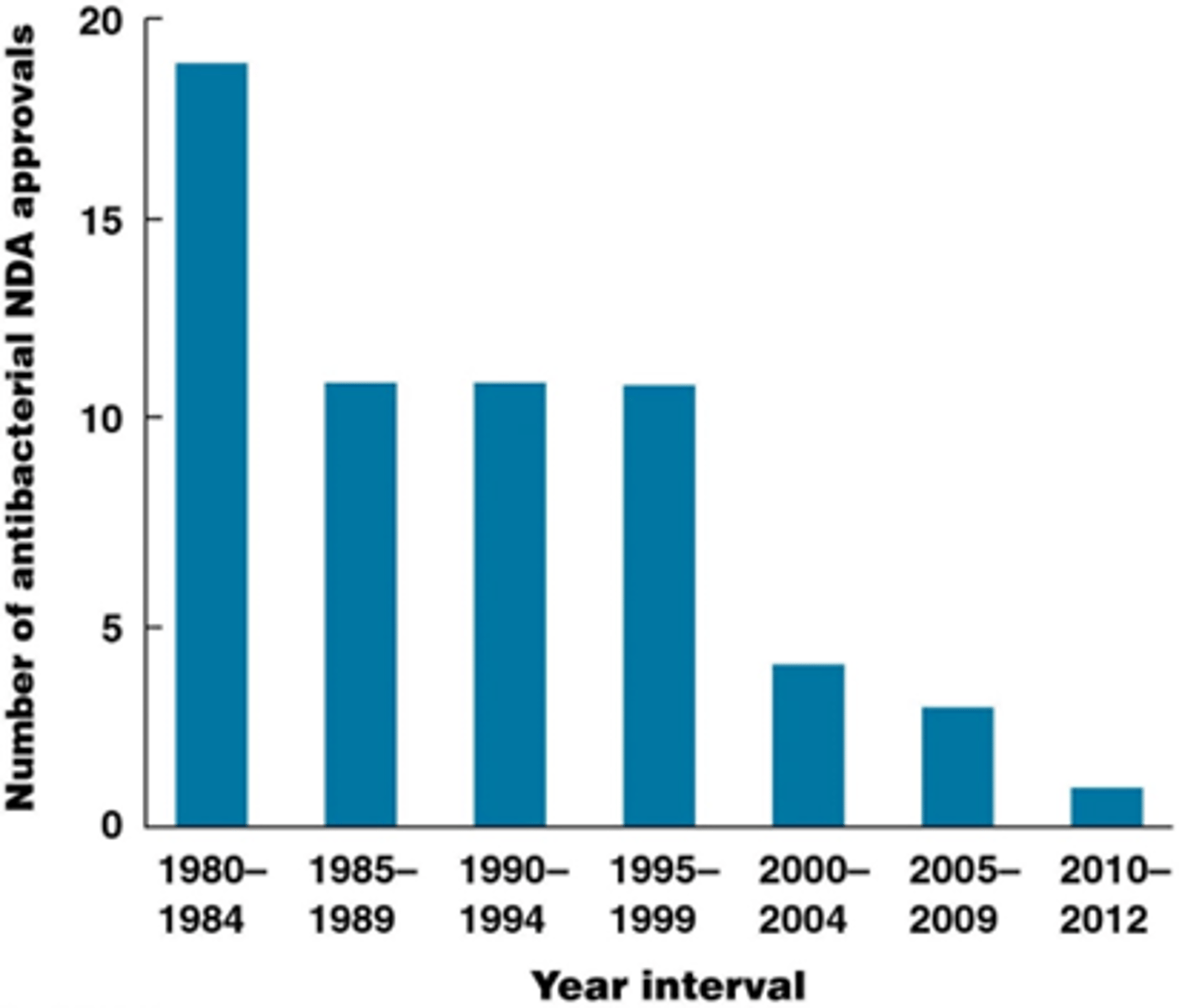
Wat is het besluit na deze kennisclip
Er is grote nood aan nieuwe klasse van antibiotica dat via een ander mechanisme werkt. Er is onderzoek aan KUL rond aggregatie van bacteriale proteïne