CM3 - Technologie de l’ADN recombinant : étapes et outils
1/23
Earn XP
Description and Tags
Partie 1 : Production de protéines recombinantes
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No study sessions yet.
24 Terms
I. II. La technologie de l’ ADN recombinant
Qu’est-ce que la technologie de l’ADN recombinant ?
Définir : ADN recombinant
Définir : transgène
Définir : protéine recombinante
Quelles sont les étapes de production d’une protéine recombinante ?
Combien de protéines recombinantes sont autorisées sur le marché en Europe ?
Quelle a été la première protéine recombinante mise sur le marché ?
C’est quoi l’ingénierie des protéines ?
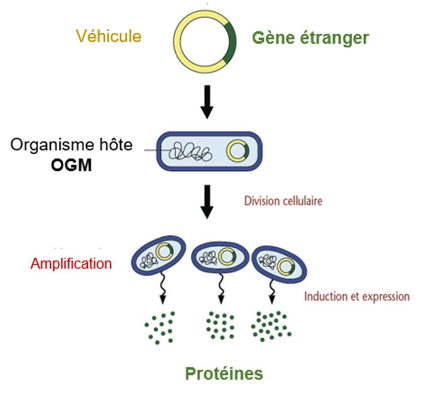
Technologie de l’ADN recombinant
Technique de laboratoire consistant à combiner de l’ADN de différentes origines et à l’introduire dans une cellule hôte afin de modifier génétiquement cette cellule et exprimer un gène d’intérêt.
ADN recombinant
Molécule d’ADN artificielle, formée in vitro, par association de séquences génétiques non naturellement associées.
Transgène
Gène étranger introduit dans le génome d’un organisme vivant.
Protéine recombinante
Protéine produite par une cellule hôte ayant reçu et exprimé un ADN recombinant.
Étapes de production d’une protéine recombinante
Obtenir l’ADN codant la protéine
Insérer le gène dans une cellule hôte
Production de la protéine par la cellule
Extraction et purification de la protéine
Nombre de protéines recombinantes autorisées en Europe
➡ Plus de 170 protéines recombinantes.
Première protéine recombinante mise sur le marché
➡ Insuline humaine recombinante (1982).
Ingénierie des protéines
Discipline visant à modifier la structure d’une protéine pour améliorer ou créer de nouvelles fonctions (activité, stabilité, efficacité).
II. Outils du génie génétique : E. coli
Quelle est la bactérie modèle et pourquoi?
Escherichia coli : une bactérie modèle
• bactérie intestinale de l’être humain et des
animaux à sang chaud
• en général non pathogène
• découverte en 1885, ADN séquencé en 1997
• génome facilement mainpulable :
plasmides/transfert de gènes
• culture aisée (temps de division 20 min à 37°C)
• usines de production de plasmides ou de protéines
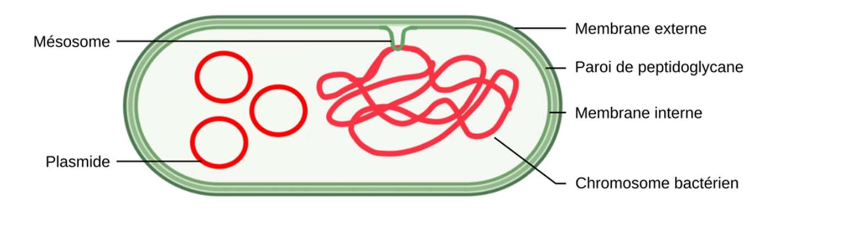
II. Outils du génie génétique : les plasmides
Définir : vecteur
Définir : plasmide
Quelles sont les caractéristiques générales des plasmides ?
Qu’est-ce qu’une carte plasmidique ?
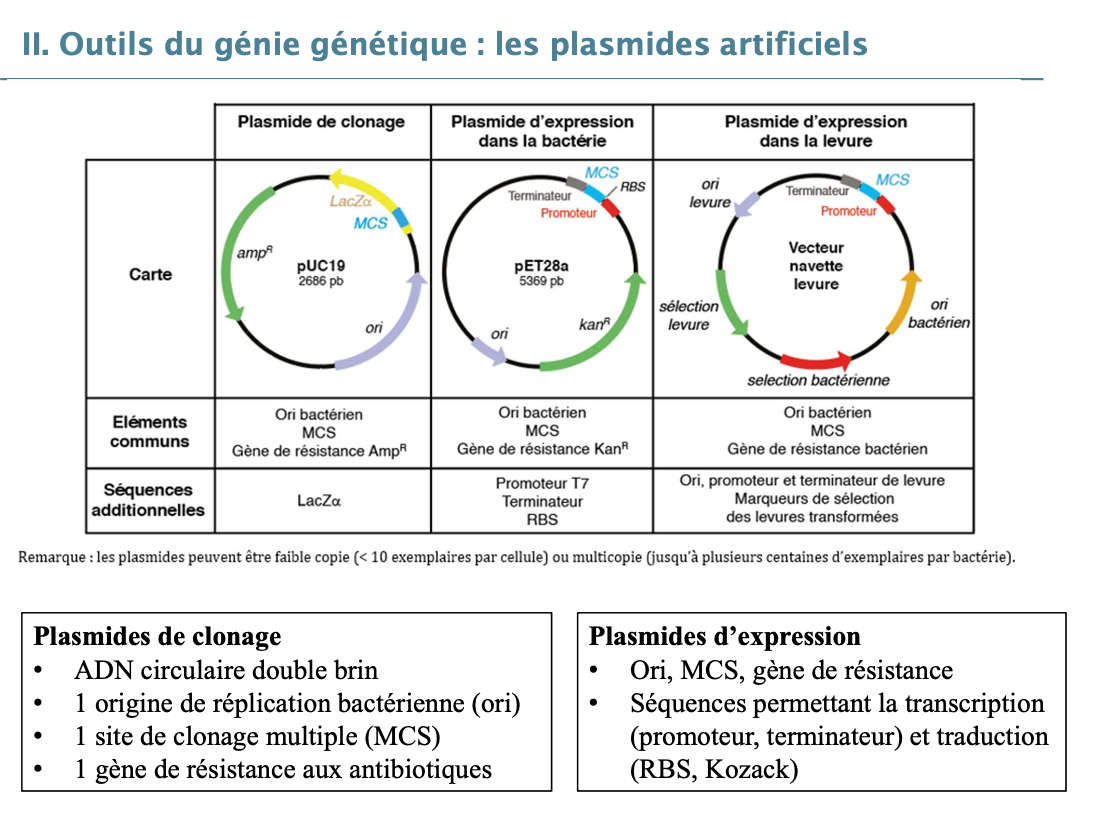
Quels sont les deux types de plasmides artificiels ?
Quelles sont les différences entre les plasmides d’expression en bactérie et en levure ?
1. Vecteur
Structure biologique naturelle ou artificielle capable de véhiculer un ADN étranger dans une cellule.
2. Plasmide
Molécule d’ADN extracromosomique, généralement circulaire double brin, capable de réplication autonome.
3. Caractéristiques des plasmides
ADN extracromosomique
Double brin circulaire fermé
Auto-réplicatif (1 origine de réplication)
Souvent isolé sous forme superenroulée
Confère un avantage sélectif (ex : résistance aux antibiotiques)
4. Carte plasmidique
Représentation schématique d’un plasmide indiquant :
Les sites de restriction
Les gènes d’intérêt
L’origine de réplication (ori)
Le sens de transcription et de réplication
5. Deux types de plasmides artificiels
Plasmides de clonage
Plasmides d’expression
6. Différences plasmides d’expression bactérie / levure
En bactérie :
Ori bactérien
Promoteur bactérien (ex : T7)
RBS
Gène de résistance aux antibiotiques
En levure :
Ori bactérien + ori levure (vecteur navette)
Promoteur et terminateur de levure
Marqueurs de sélection levure + bactérie

II. Outils du génie génétique : les enzymes
Définir : polymérase
Définir : nucléase
Définir : ligase
• Des enzymes qui copient les acides nucléiques (polymérases)
• Des enzymes qui coupent les acides nuléiques (nucléases)
• Des enzymes qui collent deux fragments d’acides nucléiques (ligases)
II. Outils du génie génétique : les polymérases
Comment fonctionnent les ADN polymérases in vivo ?
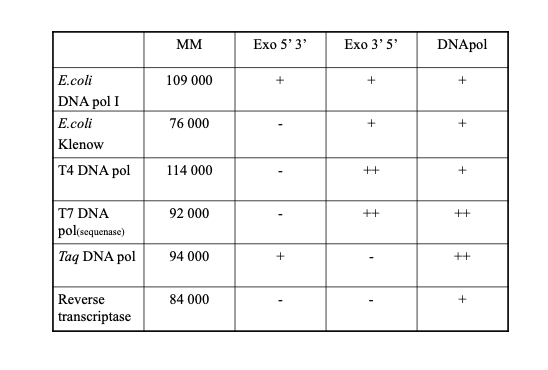
Combien y a-t-il d’ADN polymérases chez E. coli ?
Différences de rôle des ADN polymérases in vivo vs in vitro ?
Pourquoi les dNTP sont-ils essentiels ?
Quelles sont les caractéristiques de la polymérase utilisée pour la PCR ?
Donner les différents types de polymérases et leur fonction.
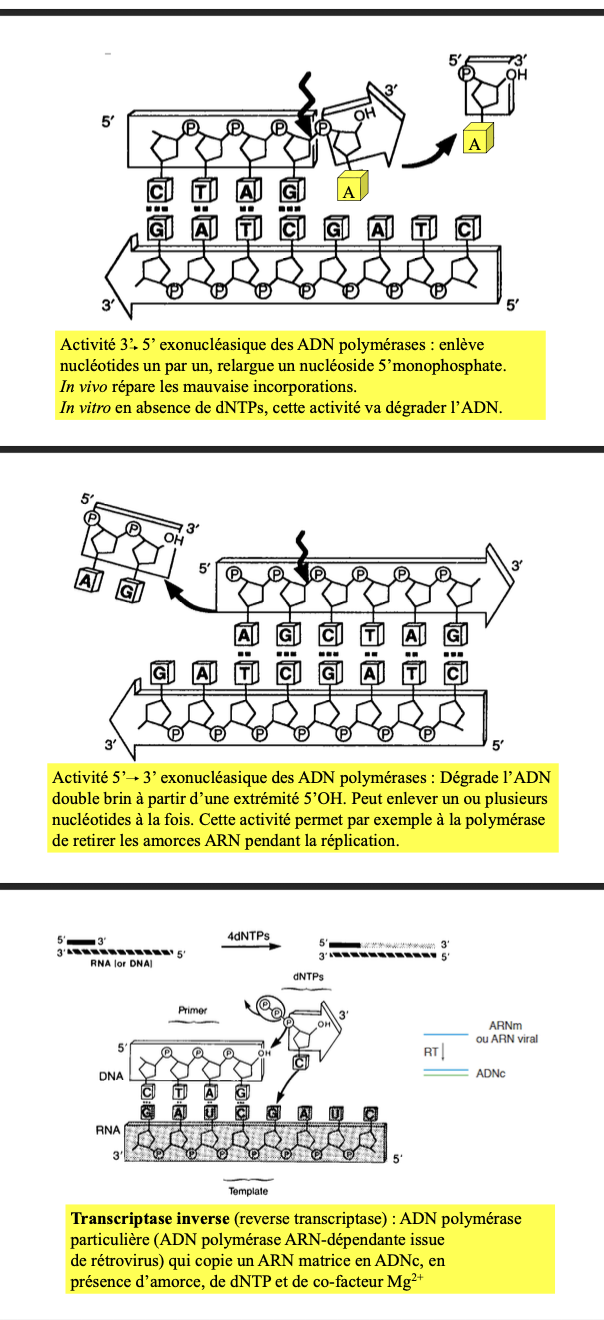
10. Fonctionnement des ADN polymérases in vivo
Utilisent des dNTP
Nécessitent une amorce ARN (synthétisée par la primase)
Synthèse 5’ → 3’
Activité exonucléasique pour la correction des erreurs
11. ADN polymérases chez E. coli
➡ 3 ADN polymérases :
ADN pol I
ADN pol II
ADN pol III
12. Rôle in vivo vs in vitro
In vivo :
ADN pol III : réplication de l’ADN
ADN pol I et II : correction / réparation
In vitro :
Les 3 peuvent synthétiser de l’ADN
13. Rôle des dNTP
Substrats nécessaires à la synthèse de l’ADN
Leur absence entraîne une activité exonucléasique dégradant l’ADN
14. Caractéristiques de la polymérase de PCR
Stable à 95 °C
Active à 72 °C
Résiste aux cycles thermiques
Ex : Taq polymérase
15. Types de polymérases et fonctions
ADN polymérases : synthèse d’ADN
Transcriptase inverse : ARN → ADNc
Terminal transférase : ajout de nucléotides en 3’
Taq polymérase : amplification PCR
T7 ADN polymérase : séquençage (faible fidélité)

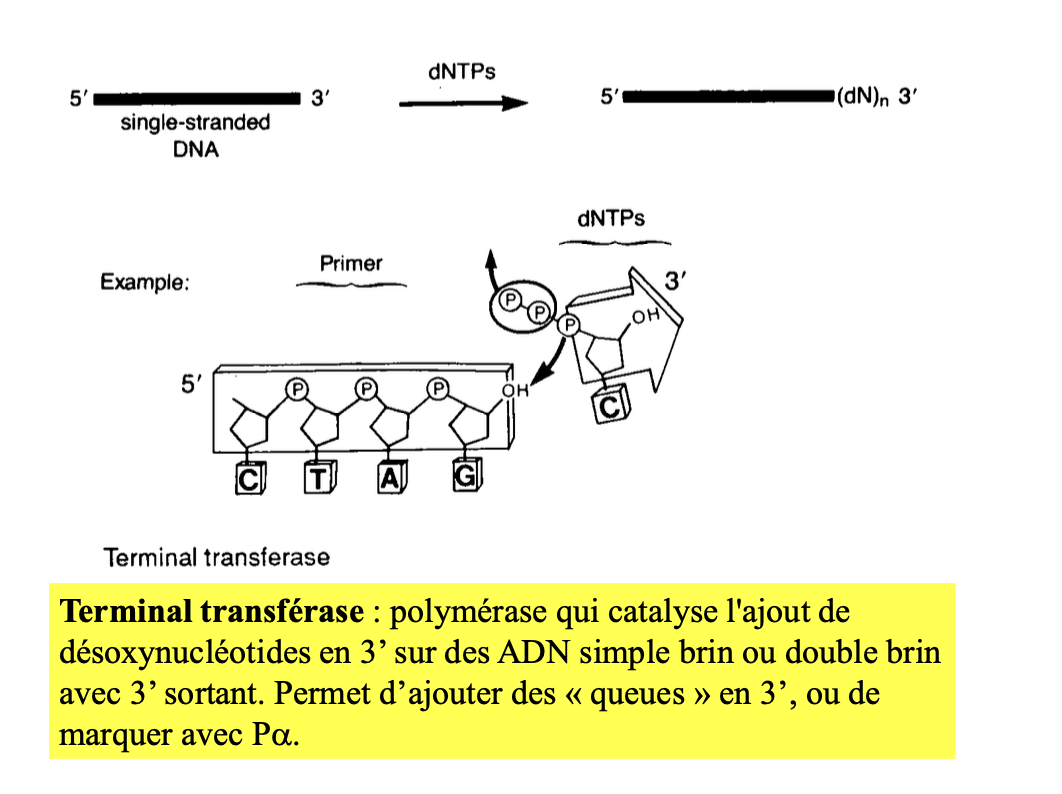

II. Outils du génie génétique : les ligases
Définir : ADN ligase
Sur quels types d’extrémités l’ADN ligase peut-elle agir ?
Quelle est la différence entre ligases utilisant l’ATP et celles utilisant le NAD ?
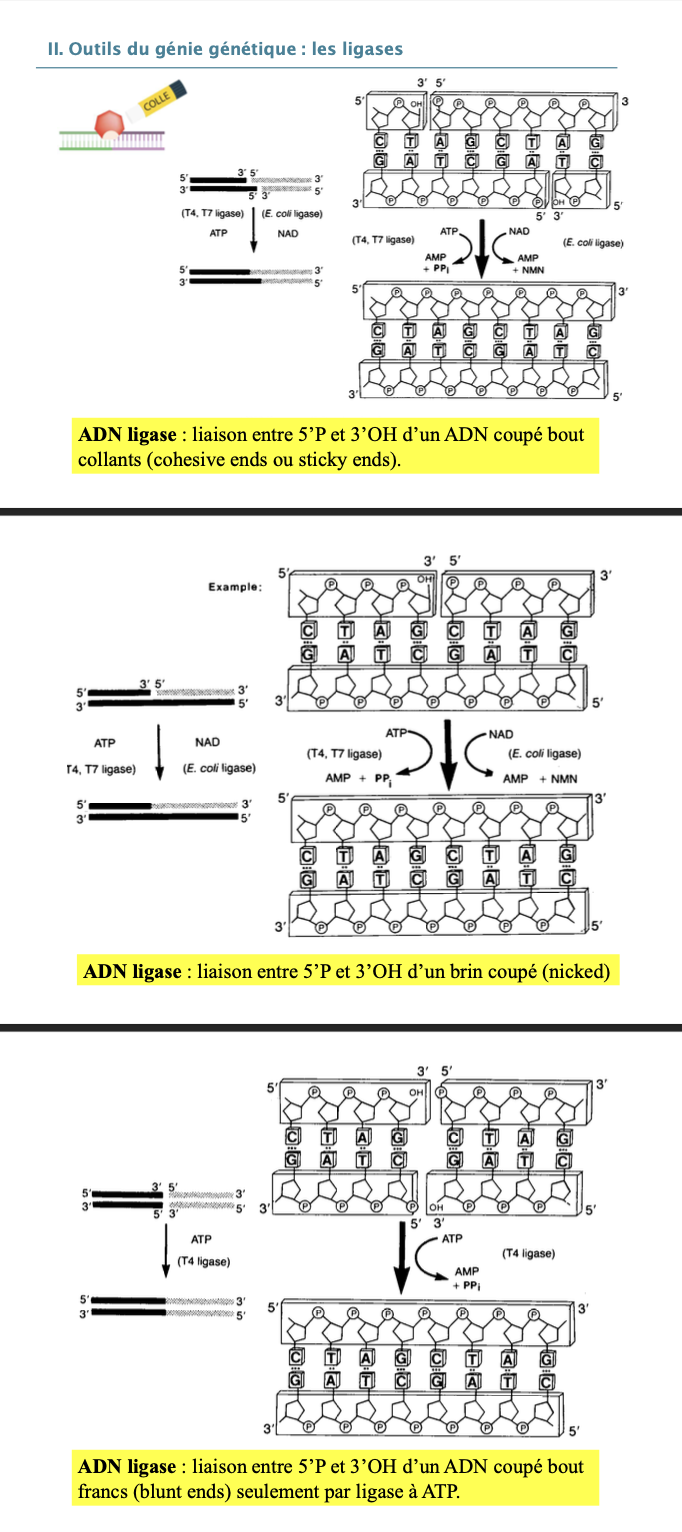
1. ADN ligase
Enzyme qui catalyse la formation d’une liaison phosphodiester entre le 5’ phosphate et le 3’ OH de fragments d’ADN.
2. Types d’extrémités reconnues par l’ADN ligase
Bouts cohésifs (sticky ends)
Bouts francs (blunt ends)
Brèches sur un brin (nicks)
3. Différence ligase ATP / NAD
Ligases T4 et T7 : utilisent ATP
Ligase d’E. coli : utilise NAD
➡ Les bouts francs ne peuvent être ligaturés que par une ligase ATP-dépendante
II. Outils du génie génétique : les nucléases
Définir : nucléase
Quelle est la différence entre endonucléase et exonucléase ?
Quel est le rôle de l’exonucléase VII ?
Quel est le rôle de l’exonucléase III ?
Quel est le rôle des exonucléases λ et T7 ?
Quel est le rôle de la RNase H ?
Donner le rôle des nucléases suivantes :
a. RNase A
b. RNase T1
c. DNase I
d. Nucléase Bal31
e. Nucléase S1
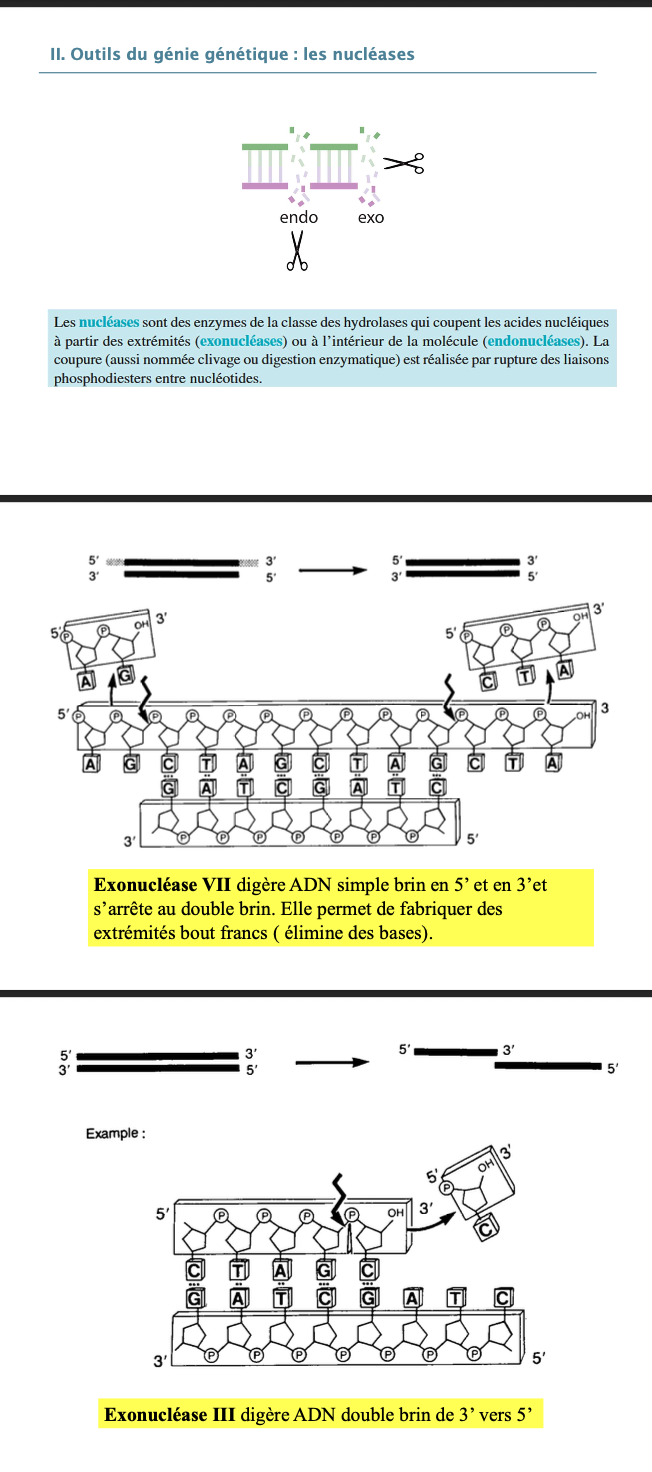

4. Nucléase
Enzyme de la classe des hydrolases qui coupe les acides nucléiques en rompant les liaisons phosphodiester.
5. Endonucléase vs exonucléase
Endonucléase : coupe à l’intérieur de la molécule
Exonucléase : coupe à partir des extrémités (5’ ou 3’)
6. Exonucléase VII
Digère l’ADN simple brin en 5’ et 3’
S’arrête au double brin
Permet de créer des bouts francs
7. Exonucléase III
Digère l’ADN double brin de 3’ vers 5’
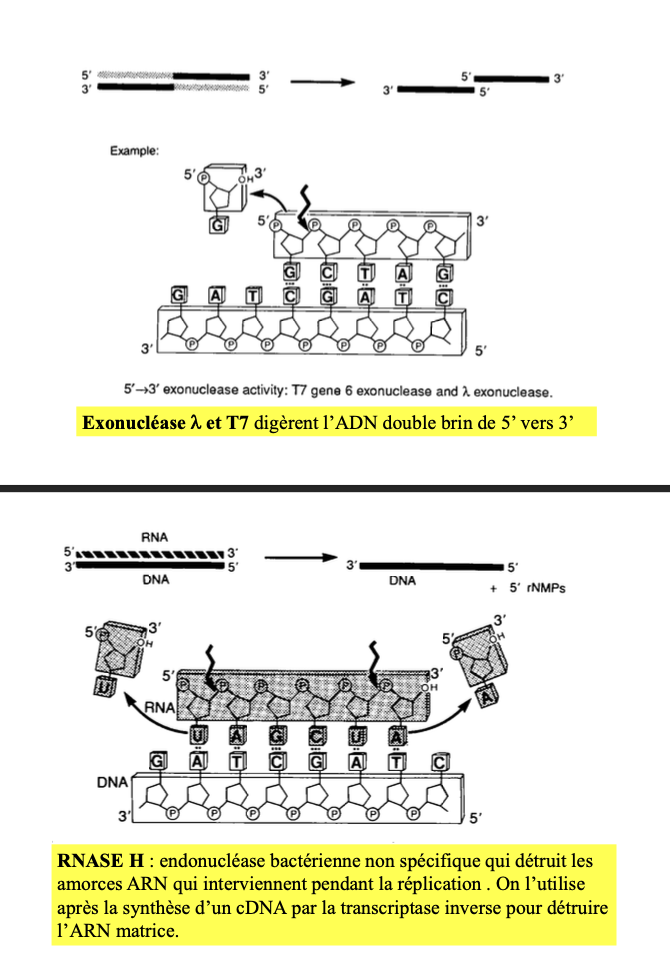
8. Exonucléases λ et T7
Digèrent l’ADN double brin de 5’ vers 3’
9. RNase H
Endonucléase détruisant les amorces ARN
Utilisée après synthèse d’ADNc pour éliminer l’ARN matrice
10. Autres nucléases
a. RNase A
Digère l’ARN simple brin
Hydrolyse entre P et C5’
b. RNase T1
Hydrolyse l’ARN en aval des G
c. DNase I
Endonucléase de l’ADN simple et double brin
Coupe de manière aléatoire
Utilisée pour créer des brèches
d. Nucléase Bal31
Exonucléase mange les extrémités 3’
Crée des brins 5’ sortants
Digère ensuite l’ADN simple brin
Peut tout digérer
e. Nucléase S1
Spécifique des simples brins ADN ou ARN
Utilisée pour créer des bouts francs
II. Outils du génie génétique : enzymes de restriction
Définir : enzyme de restriction
En quoi consiste le système restriction–modification bactérien ?
Pourquoi l’ADN bactérien n’est-il pas digéré par ses propres enzymes de restriction ?
Quelles séquences sont reconnues par les enzymes de restriction ?
Pourquoi la plupart des sites de restriction sont-ils palindromiques ?
Qu’est-ce qu’un fragment de restriction ?
Quels sont les trois types d’extrémités générées par les enzymes de restriction ?
1. Enzyme de restriction
Endonucléase qui coupe les deux brins d’ADN après reconnaissance d’une séquence spécifique (site de restriction).
2. Système restriction–modification
Système de défense bactérien contre les bactériophages associant :
enzymes de restriction (digestion ADN étranger)
enzymes de méthylation (protection ADN bactérien)
3. Protection de l’ADN bactérien
Les bases des sites de restriction sont méthylées, empêchant la coupure.
4. Séquences reconnues
Séquences spécifiques de 4, 6 ou parfois 8 nucléotides.
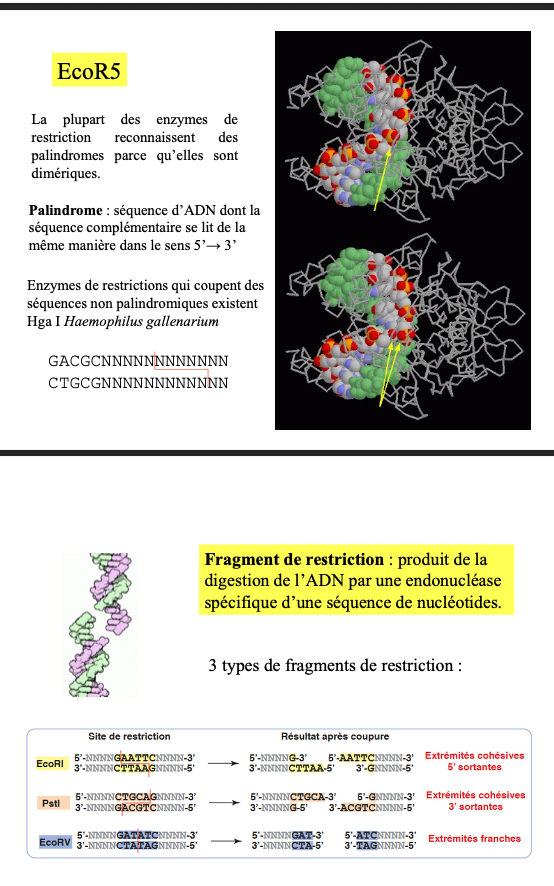
5. Séquences palindromiques
Les enzymes de restriction sont dimériques et reconnaissent des séquences lues identiquement en 5’→3’ sur les deux brins.
6. Fragment de restriction
Fragment d’ADN obtenu après digestion par une enzyme de restriction.
7. Types d’extrémités
Extrémités cohésives 5’ sortantes
Extrémités cohésives 3’ sortantes
Extrémités franches

II. Outils du génie génétique : kinases et phosphatases
Quel est le rôle général des kinases et des phosphatases ?
Quel est le rôle de la T4 polynucléotide kinase ?
Quel est le rôle de la phosphatase alcaline ?
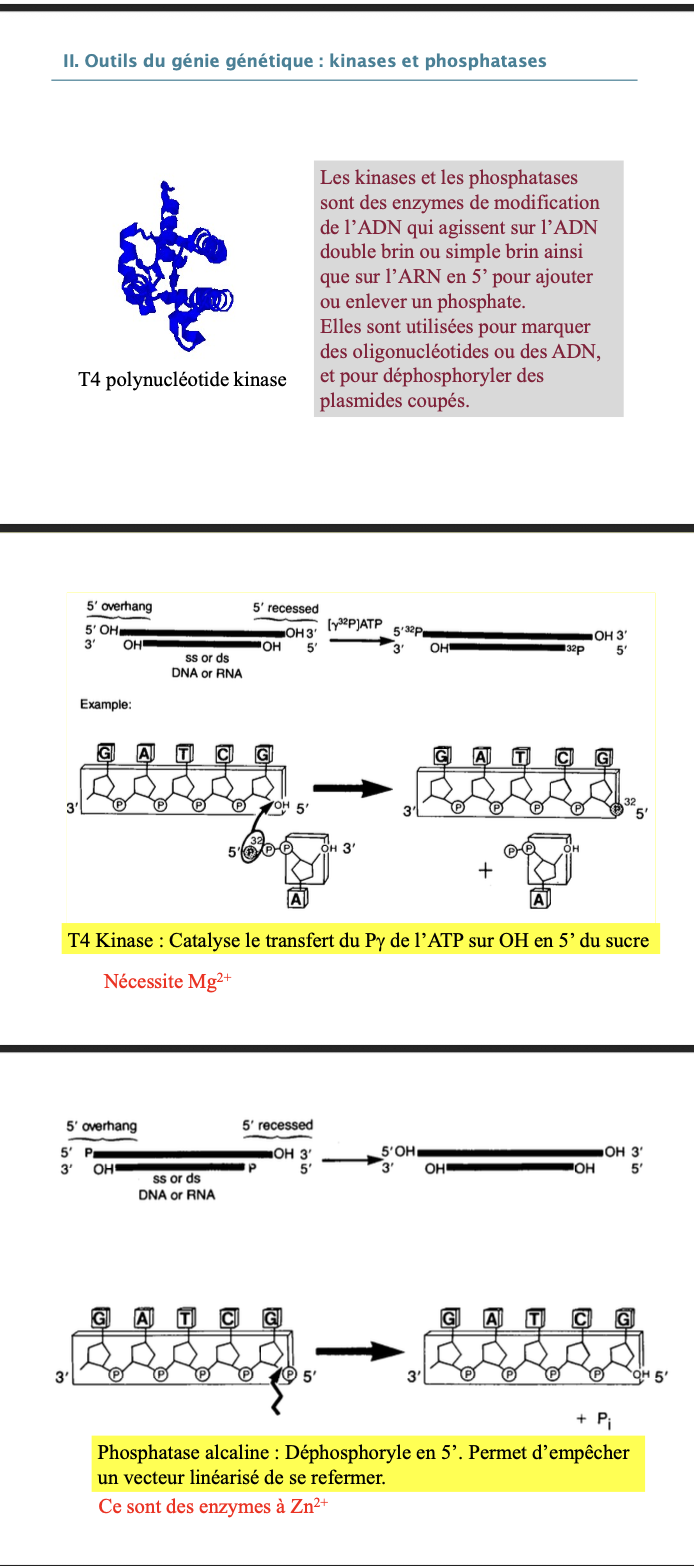
8. Rôle kinases / phosphatases
Enzymes modifiant l’ADN ou l’ARN en ajoutant ou retirant un phosphate en 5’.
9. T4 polynucléotide kinase
Transfère le phosphate γ de l’ATP sur le OH en 5’ des acides nucléiques.
➡ Nécessite Mg²⁺
10. Phosphatase alcaline
Déphosphoryle les extrémités 5’.
➡ Empêche la religation d’un vecteur.
➡ Enzyme Zn²⁺ dépendante
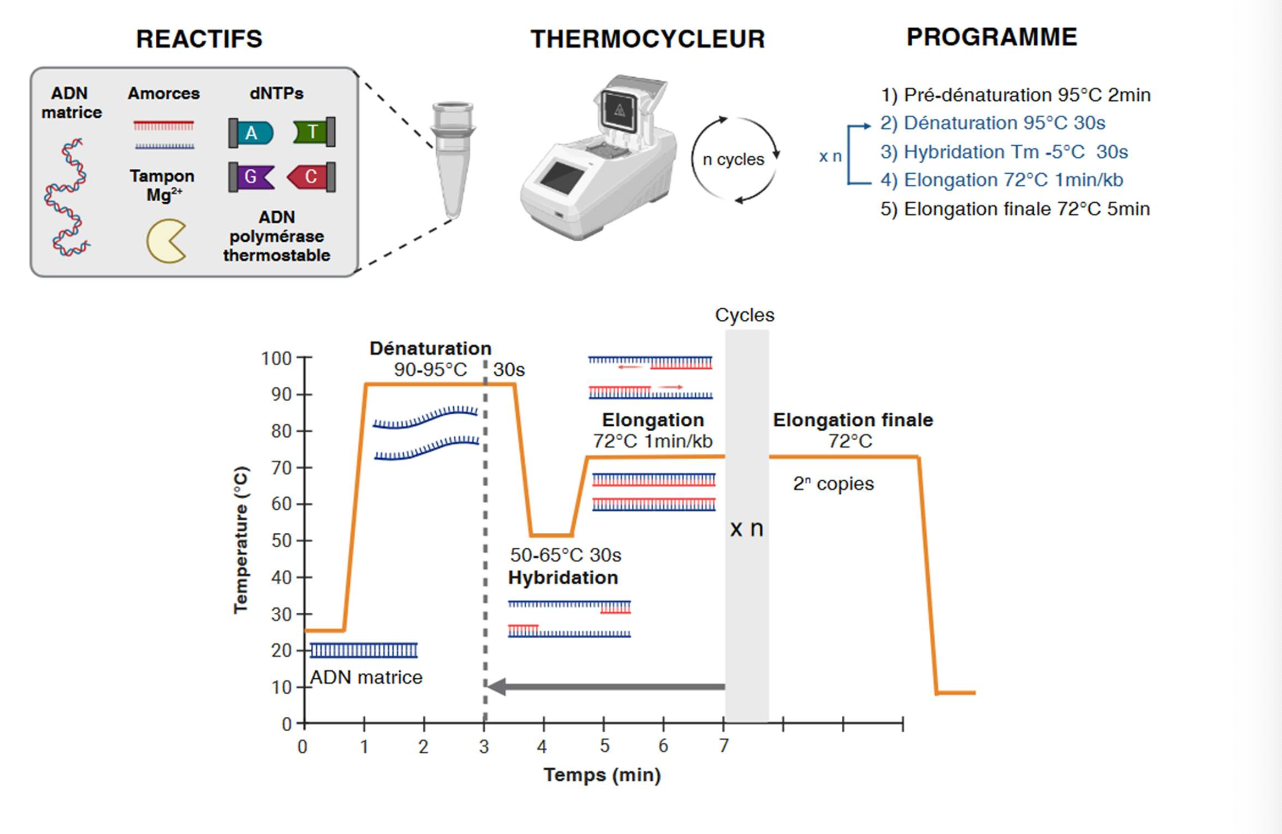
III. Techniques de biologie moléculaire : PCR
Définir : PCR
Définir : ADN matrice
Définir : amorces de PCR
Définir : amplicon
Quel est le rôle des amorces dans la PCR ?
Quelles sont les caractéristiques générales des amorces de PCR ?
Pourquoi utilise-t-on une polymérase thermorésistante ?
Quelles sont les caractéristiques de la Taq polymérase ?
Quelles sont les caractéristiques de la Pfu polymérase ?
Pourquoi utiliser des mélanges Taq + Pfu ?
Quelle est la particularité de la KOD polymérase ?
Pourquoi l’ajout d’un A en 3’ par la Taq est-il utile en clonage ?
11. PCR
Technique d’amplification in vitro d’un ADN cible à l’aide d’une polymérase, d’amorces et de nucléotides.
12. ADN matrice
ADN source contenant la séquence cible à amplifier.
13. Amorces de PCR
Courts oligonucléotides ADN simple brin servant de point de départ à la synthèse.
14. Amplicon
Fragment d’ADN délimité par les amorces, produit de l’amplification PCR.
15. Rôle des amorces
S’hybrident aux extrémités 3’
Délimitent la région amplifiée
16. Caractéristiques des amorces
Longueur : 15–30 nucléotides
Tm proches (±5 °C)
%GC : 40–60 %
Pas de dimères ni hairpins
17. Polymérase thermorésistante
Résiste aux cycles de dénaturation à haute température.
18. Taq polymérase
Rapide
Peu fidèle (1 erreur / 1000 pb)
Pas d’activité exo 3’→5’
Ajoute un A en 3’
Fragments ≤ 6 kb
19. Pfu polymérase
Lente
Très fidèle (1 erreur / 10 000 pb)
Activité exonucléasique
Produit des bouts francs
20. Mélanges Taq + Pfu
Compromis entre vitesse et fidélité, bouts francs.
21. KOD polymérase
Très rapide
Fragments longs (<21 kb)
Bouts francs
22. Intérêt du A en 3’
Permet le clonage TA dans des vecteurs portant un T en 5’.
III. Techniques de biologie moléculaire : RT-PCR
Définir : RT-PCR
Quelles sont les deux étapes de la RT-PCR ?
23. RT-PCR
Technique permettant de faire une PCR à partir d’ARN.
24. Étapes RT-PCR
Transcription inverse (ARN → ADNc)
PCR classique

III. Techniques : introduction d’ADN exogène dans les cellules
Définir : transformation
Définir : transfection
Définir : transduction
En quoi consiste la transformation chimique CaCl₂ / choc thermique ?
En quoi consiste l’électroporation ?
25. Transformation
Introduction d’ADN ou ARN exogène dans des cellules bactériennes ou végétales.
26. Transfection
Introduction d’acides nucléiques dans des cellules eucaryotes animales.
27. Transduction
Transfert d’ADN exogène via un vecteur viral.
28. Transformation CaCl₂ / choc thermique
Ca²⁺ neutralise les charges → choc thermique → nanopores transitoires.
29. Électroporation
Champ électrique créant des pores membranaires réversibles.
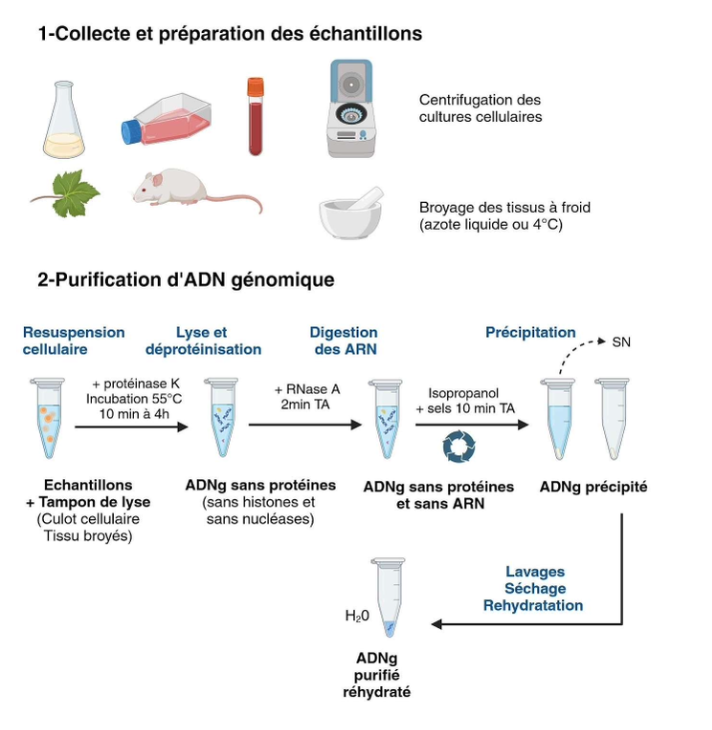
III. Techniques de purification des acides nucléiques : ADN génomique
Quelles sont les grandes étapes de purification de l’ADN génomique ?
Quel est le rôle de la protéinase K ?
Quel est le rôle de la RNase A ?
Pourquoi utilise-t-on l’isopropanol lors de la purification ?
30. Étapes purification ADN génomique
Lyse cellulaire
Déprotéinisation
Digestion ARN
Précipitation
Lavage / réhydratation
31. Protéinase K
Dégrade les protéines et nucléases.
32. RNase A
Dégrade les ARN.
33. Isopropanol
Permet la précipitation de l’ADN.
III. Techniques : purification d’acides nucléiques sur support solide
Quelles sont les grandes étapes d’une purification sur support solide ?
Sur quoi repose la chromatographie sur phase de silice ?
Sur quoi repose la chromatographie échangeuse d’ions ?
Sur quoi repose la chromatographie d’affinité ?
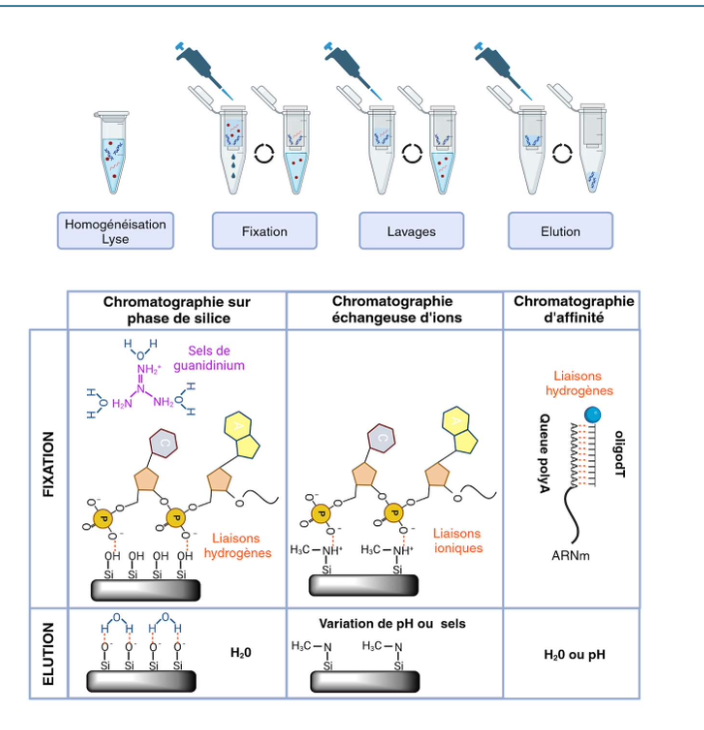
1. Étapes purification sur support solide
Lyse / homogénéisation
Fixation des acides nucléiques
Lavages
Élution
2. Chromatographie sur silice
Fixation de l’ADN via liaisons hydrogène en présence de sels chaotropiques (guanidinium), élution à l’eau.
3. Chromatographie échangeuse d’ions
Interaction par liaisons ioniques, élution par variation de pH ou de force ionique.
4. Chromatographie d’affinité
Fixation spécifique via interaction ligand–cible, élution par eau ou changement de pH.
III. Techniques de purification d’acides nucléiques : Miniprep
Quel est le principe général de la miniprep plasmidique ?
Quelles sont les étapes clés de la miniprep ?
Quel est le rôle du SDS et du NaOH ?
Quel est le rôle de la neutralisation acide (acétate de potassium) ?
Pourquoi l’ADN plasmidique reste-t-il en solution après neutralisation ?
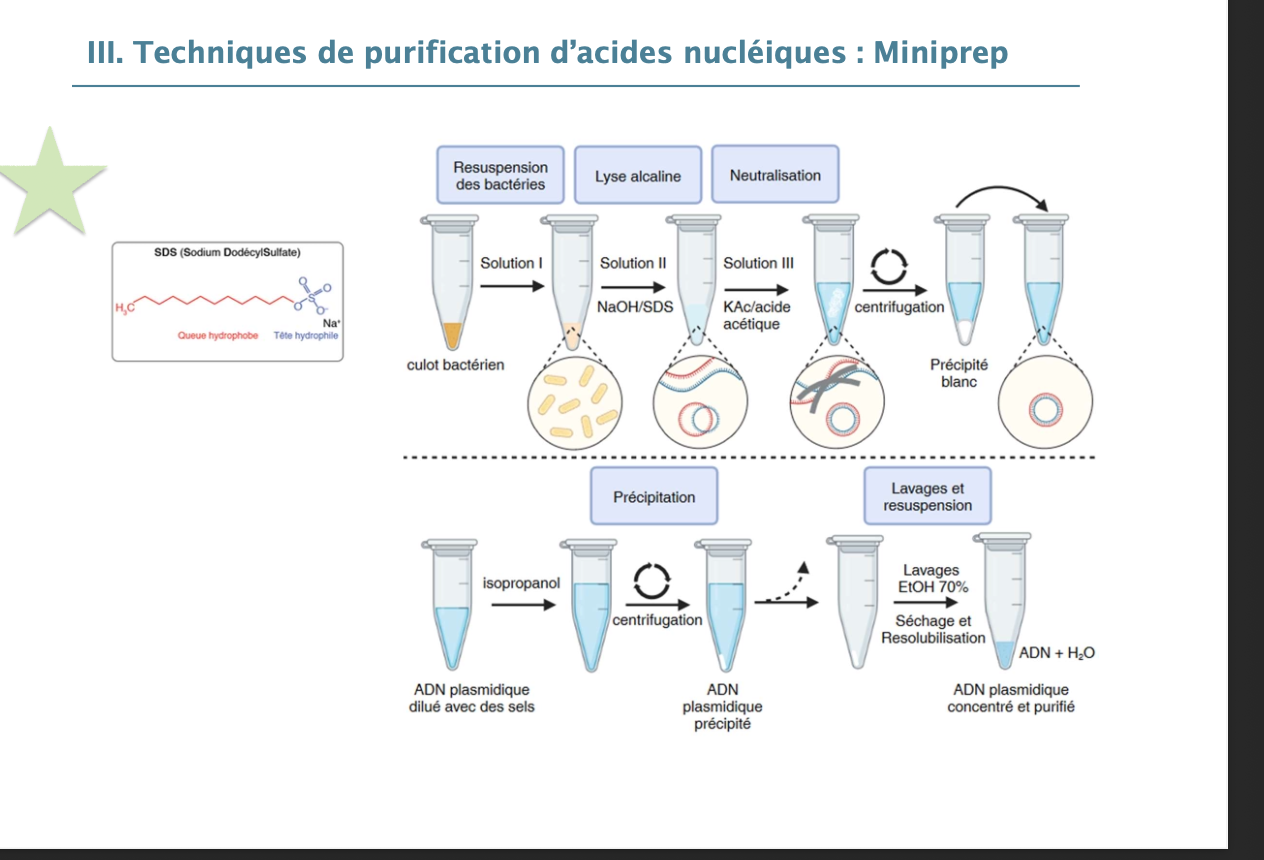
5. Principe de la miniprep
Isolation rapide de l’ADN plasmidique à partir de bactéries par lyse alcaline sélective.
6. Étapes de la miniprep
Resuspension
Lyse alcaline
Neutralisation
Centrifugation
Précipitation
Lavages
Resuspension
7. Rôle SDS / NaOH
SDS : lyse des membranes
NaOH : dénaturation ADN et protéines
8. Rôle neutralisation acide
Précipite ADN génomique, protéines et membranes sous forme de culot.
9. ADN plasmidique en solution
ADN circulaire superenroulé renature correctement après neutralisation.
III. Dosage, pureté et intégrité des acides nucléiques
Définir : absorbance (A) ou densité optique (DO)
À quelle longueur d’onde absorbent les acides nucléiques ?
À quelle longueur d’onde absorbent les protéines ?
Comment calculer la concentration en ADN ou ARN à partir de A260 ?
À quoi servent les rapports A260/A280 et A260/A230 ?
10. Absorbance (DO)
Mesure de la capacité d’une solution à absorber la lumière à une longueur d’onde donnée.
11. Absorption des acides nucléiques
➡ 260 nm
12. Absorption des protéines
➡ 280 nm
13. Calcul concentration
ADN double brin : C = A260 × 50 µg/mL
ADN simple brin : C = A260 × 33 µg/mL
ARN : C = A260 × 40 µg/mL
14. Rapports de pureté
A260/A280 : contamination protéique
A260/A230 : contamination organique (phénol, sels)
IV. Étapes globales du clonage moléculaire
Quelles sont les grandes étapes d’un projet de clonage ?
Étapes globales clonage
Préparer l’ADN cible
Stratégies de clonage
Montage d’expression
Optimisation production
Ingénierie génétique
CRISPR/Cas9


IV. Étape 1 : Préparer l’ADN cible
Quelles sont les étapes de préparation de l’ADN génomique procaryote ?
Quel est le rôle du lysozyme ?
Quel est le rôle de la protéinase K ?
Quel est le rôle de la RNase A ?
16. Préparation ADN génomique procaryote
Culture bactérienne
Lyse (lysozyme)
Protéinase K
RNase A
Extraction phénol/CHCl₃
Précipitation EtOH
17. Rôle du lysozyme
Digère la paroi bactérienne.
18. Rôle protéinase K
Dégrade les protéines et nucléases.
19. Rôle RNase A
Dégrade les ARN.
IV. Étape 1 : Préparation du cDNA eucaryote
Comment est synthétisé le cDNA à partir du mRNA ?
Pourquoi faut-il que le gène soit exprimé dans l’échantillon de départ ?
22. Synthèse du cDNA
Transcriptase inverse
Amorçage par oligo-dT
Destruction du mRNA
Synthèse du brin complémentaire ou PCR
23. Expression préalable du gène
Le mRNA n’est présent que si le gène est exprimé.
IV. Étape 1 : Synthèse de gène
En quoi consiste la synthèse chimique d’un gène ?
24. Synthèse de gène
Assemblage chimique d’oligonucléotides par cycles successifs.
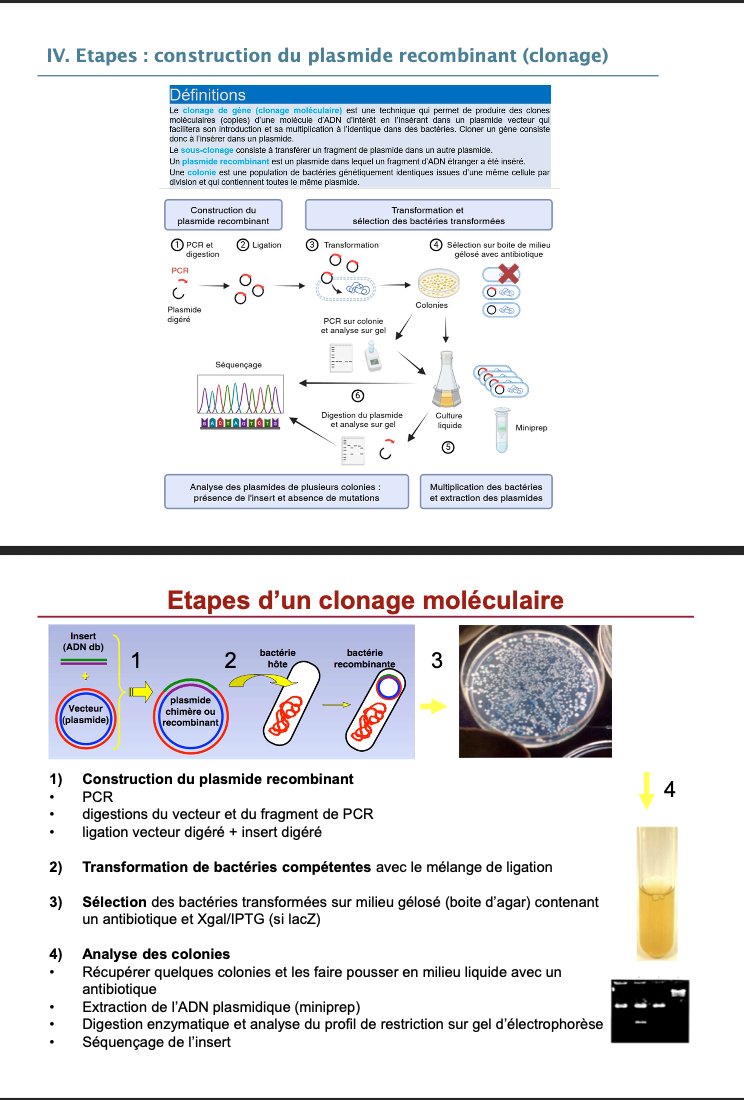
IV. Étape 2 : Construction du plasmide recombinant (clonage)
Définir : clonage moléculaire
Qu’est-ce qu’un plasmide recombinant ?
Qu’est-ce qu’une colonie bactérienne clonale ?
Quelles sont les étapes d’un clonage moléculaire classique ?
25. Clonage moléculaire
Insertion d’un fragment d’ADN d’intérêt dans un vecteur plasmidique pour amplification.
26. Plasmide recombinant
Plasmide contenant un ADN étranger inséré.
27. Colonie clonale
Population de bactéries génétiquement identiques issues d’une seule cellule transformée.
28. Étapes du clonage moléculaire
Construction plasmide recombinant
Transformation bactérienne
Sélection sur antibiotique
Analyse des colonies (miniprep, digestion, séquençage)