2.4 Protein tertiary and quaternary structure analysis
1/11
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No study sessions yet.
12 Terms
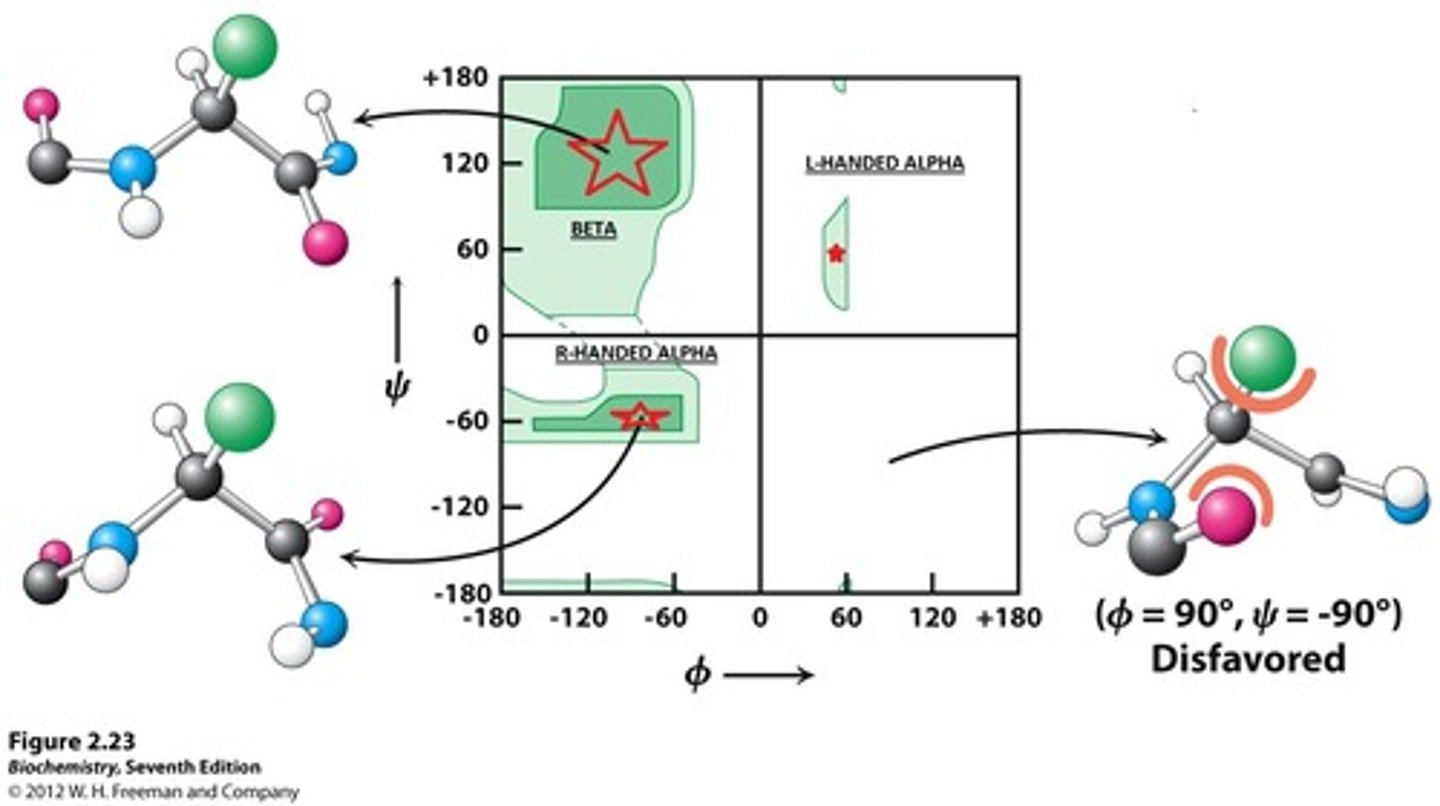
Ramachadran plot
Winkeln:
Φ: C-N-Cα-C Ramachandran plot
Ψ: N-C-Cα-N Ramachandran plot
ω: peptide bond 180 (trans), 0 (cis)
Strukturbiologie ermöglicht: (5)
•Verständnis von Aufbau und Funktionen von Proteinen und Multiproteinkomplexen
•Erkenntnisse zu Faltung und evolutionären Verwandtschaftsbeziehungen
•mechanistische Einblicke in Enzymreaktionen
•strukturbasierte Modifikation von Enzymen für biotechnologische Zwecke
•strukturbasierte Medikamentenentwicklung/Suche nach Hemmstoffen
Methoden zur Strukturbestimmung: (3)
-Nuclear Magnetic Resonance (NMR) spectroscopy
-X-ray crystallograhy
-Cryo-Electron Microscopy (Cryo-EM)
NMR spectroscopy
Nuclear Magnetic Resonance
für 1H, 13C(nicht 12C!), 15N, 19F, 31P
Atome besitzen ein Spin, Magnetfelder richten die Spinne der Atome aus (parallel/antiparallel zum magn.Feld)
Transmitter berechnet die benötige E
supraleitende (keine Wärme entsteht, durch Kühlen mit flüßigen He und N_2) Spulle generiert ein sehr starkes Magnetfeld
kl. Org. Moleküle, kl. Proteine in Lösung (Deuterium 2H Wasser)
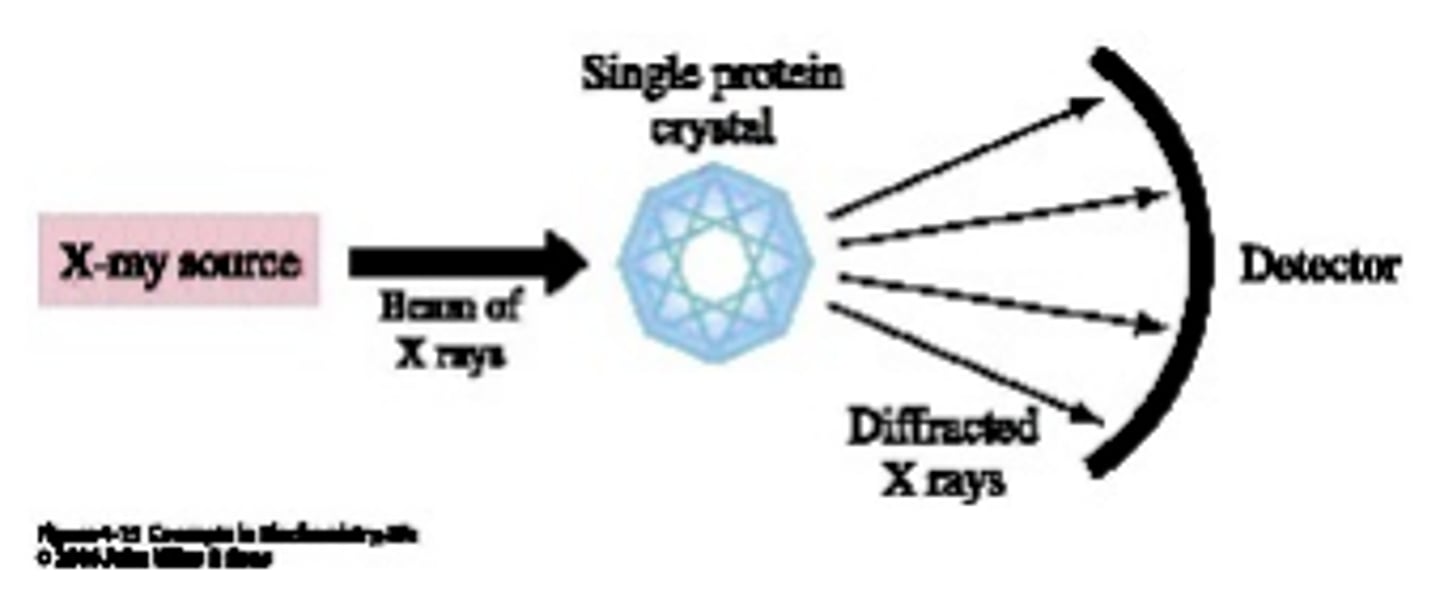
X-Ray crystallography
Krystal wird aus einer Röntgenquelle bestrahlt, die mit einem Detektor delektiert werden (0,5-2Å)
->Beugungsbild (Intensität der Röntgenstrahlung, weil es keine Linsen gibt, um die Strahlungen zu binden und ein Bild des Proteins zu generieren).
->daraus die Struktur ausrechnen
kl., mitt., gr. Moleküle (nicht alle Proteine lassen sich kristallisieren)
*Struktur im Krystall sind identisch zur Struktur in einer Lösung.
*Wegen Röntgenstrahlung muss man den auf -170°C kühlen (H2O kristallisiert sich und deswegen die Protein geht kaputt) → Proteine in Frostschutzmitteln zuerst eintauchen
Cryo-EM
Cryo-Electron Microscopy
Lichtquelle, magnetische Linsen, Probenhalter, Detektor, fl. Ethan zum Kühlen
→ direktes Bild
→ einzelne Bilder von Proteine in aller Orientierungen auf einem Computer zu einer 3D-Struktur zusammenbinden
mittelgroße-große Proteine
tertiäre und quaternäre Struktur von Proteinen (2) DB
-PDB
-CATH
(+Dali server
+Foldseek (vergleichen)
+PISA server)
PDB
the RCSB Protein DB (1971)
speichert Biopolymerstrukturen (DNA; RNA; Proteine; Protein-DNA-Komplexe usw.)
Zeigt:
die Abbildung, Code, Tittle, Literatur - Link zu anderen DB (z.B. PubMed), Link zu UniPlotDB für die Sequenzinformation
Datenformat: PDB, mmCIF (macromolecular crastallographic information file)
PDB Dateiformat
•Atomkoordinaten
•Chemische Verbindungen zwischen den Atomen
•Sekundärstruktur
•Experimentelle Daten: Methoden zur Strukturbestimmung
•Zusätzliche Metadaten: Angaben zur Quelle des Moleküls (z. B. Organismus), experimentellen Bedingungen und mehr.
CATH
Protein Structure Classification DB
C: Class (α/β/α+β)
A: Architecture
T: Topology
H: Homologous Superfamily
Dali Server
Liste von ähnliche oder verwandte Proteinstrukturen aus PDB
Structural similarity is measured by Z-Score:
>2: Significant similarities, usually similar folds
>n/10-4 (with n = number of AS of query protein): strong match
PISA server
(Proteins, Interfaces, Structures and Assemblies)
berechnet ob ein Protein ein Monomer oder Multimer ist;
Zeigt die Interaktionsfläche von Molekülen im Krystall (wenn groß → Multimer)