Synthese des proteines
1/122
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
123 Terms
Quelle est la difference au niveau des etapes de la traduction dans sa globalite entre les procaryotes et eucaryotes ?
Chez les procaryotes, la traduction se fait pratiquement instantanément apres la transcription alors que chez les eucaryotes il faut que l’ARNm quitte le noyau etc.
Que synthetise un brin d’ARNm ?
Un seul polypeptide
Dans quel sens se fait la traduction de l’ARNm ?
5’ → 3’
A quoi sert la partie 5’ UTR ?
Pour la stabilite de l’ARNm et l’efficacite de la traduction
A quoi sert la 3’ UTR ?
Contient des regions regulatrices qui influencent l’expression des genes au niveau post transcriptionnel
Combien de triplets de nucleotides possible pour code un aa ?
61, car 3 codons stop qui ne codent pas
Quels sont trois codons stop ?
UAA, UAG, UGA
Quelle est la structure secondaire de l’ARNt ?
4 helices et 3 boucles (attention pas proteine)
Quelles sont les 3 boucles et a quoi servent elles ?
Boucle D : interagit avec l’aminoacyl-ARNt synthetase
Boucle T : se fixe au ribosome
Boucle de l’anticodon : se fixe au codon sur l’ARNm
Ou se lie l’acide amine sur l’ARNt
Sur le site de liaison en 3’ de l’ARNt
Que peut on dire sur la sequence des bases sur les boucles de l’ARNt ?
Il y a desfois des bases modifiees (genre ionisine).
Combien de bases modifiees par boucle ?
boucle D = 2
boucle de l’anticodon = 1
boucle T = 3
Combien de nucleotides par boucle ?
80
Quelle est la 4 eme boucle ?
Boucle variable gamma
A quoi sert l’aminoacyl-ARNt synthetase ?
Lie le bon ARNt au bon acide amine
Combien d’aminoacyl-ARNt synthetase y a t il ?
Il y en a 20
Combien d’erreurs de la aminoacyl-ARNt synthetase ?
Une erreur tous les 1E5
Comment y a t il une frequence d’erreur si basse ?
Car il y a une activite de correction d’epreuves
Comment l’aa-ARNt synt. reconnait l’ARNt ?
Elle se lie a deux regions de l’ARNt pour le reconnaitre :
La souche acceptatrice (partie qui se lie a l’aa en 3’)
Une region specifique
Soit la boucle anticodon
Soit une autre region caracteristique comme la boucle D
Comment l’ARNt reconnait l’acide amine ?
Suivant la taille et les groupements chimiques reactifs
Que peuvent avoir les groupement qui facilitent la reconnaissane par l’ARNt synthetase ?
Par exemple la threonine a un groupe OH et NH3+ qui lui permet de fixer un zinc → reconnaissance par l’ARNt synthetase
Comment se forme la liaison entre un ARNt et l’acide amine ?
ATP hydrolyse et perd 2 phosphates
AMP se lie au C du groupement carboxyle, ce qui fait perdre le OH du C. Cela forme un aminoacyl-adenylate
Transfert de cette molecule sur l’ARNt en formant une liaison ester → perte du AMP sur l’acide amine
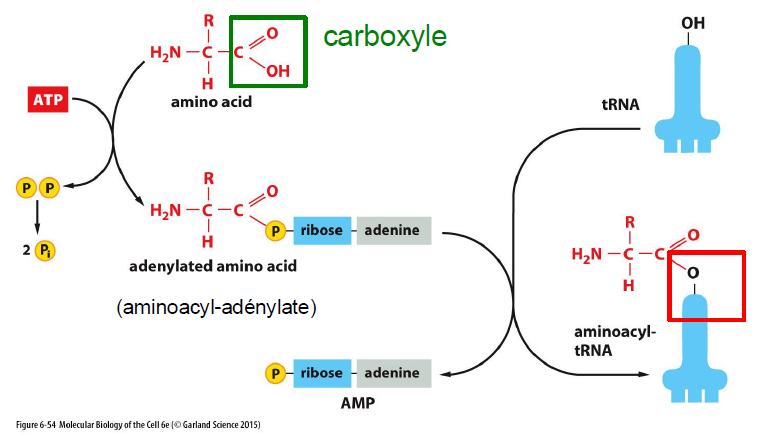

D ou vient l’energie pour former la liaison ARNt - acide amine ?
De l’hydrolyse de l’ATP
Dans une cellule (dis si procaryote ou eucaryote) y a combien de ribosomes ainsi que la masse totale par rapport a la cellule ?
Cellule procaryote (car eucaryotes sont trop varies) il y a 20 000 ribosomes, faisant 25% du poids cellulaire
Ou peuvent etre localise les ribosomes e t quel est leur role en fonction de leur localisation ?
Dans le cytoplasme qui a pour role de generer des proteines qui restent a l interieur de la cellule
Associe a un RE pour generer des proteines soit membranaires soit secretees, soit pour endosomes/lysosomes/RE/Golgi
De quoi est compose un ribosome chez un procaryote ?
La grande unite est formee de
2 molecules d’ARNr : 23S, 5S
31 proteines → L1 - L31
La petite unite est formee de :
une molecule d’ARNr : 16S
21 proteines → S1 - S21
De quoi est forme un ribosome chez un eucaryote ?
La grande unite est formee de
3 molecules d’ARNr : 28S et 5.8S lies, et 5S
50 proteines → L1 - L50
La petite unite est formee de :
une molecule d’ARNr : 18S
33 proteines → S1 - S33
Quel est le taux de sedimentation des ribosomes de eucaryotes ?
80 S
Quel est le taux de sedimentation des ribosomes de procaryotes ?
70 S
A quoi servent les ARNr chez les ribosomes ?
forment le squelette du ribosome
role enzymatique → ribozyme
A quoi sert le ribosome ?
A lie l’ARNm avec les ARNt et formation du polypeptide
A quoi servent les grands ARNr dans les ribosomes ? Quelle est le nom de son activite ?
Fonction catalytique dans les liaisons polypeptidiques. C est une activite peptidyl-transferase
Comment est un ribosome au repos ?
La grande unite et la petite unite sont separees
Comment sont separees les deux unites d’un ribosome au repos ?
Grace aux facteurs :
eIF6 qui s’attache a la grande unite
eIF3 qui s’attache a la petite unite
eIF sont les facteurs d’initiation
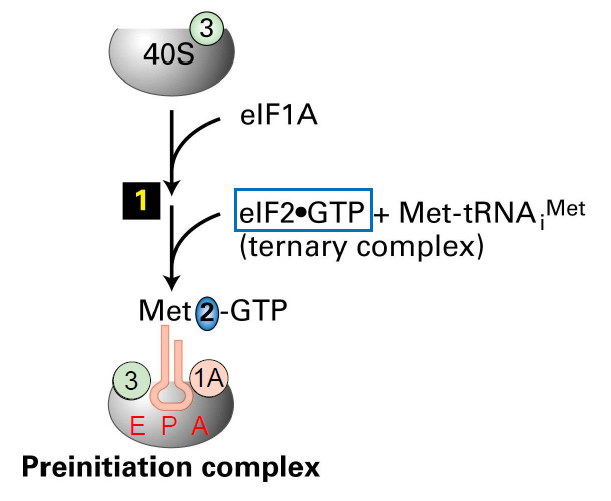
Comment se forme le complexe de pre initiation ?
d’abord le facteur eIF1A se fixe a la sous unit 40S du ribosome au niveau du site A
ensuite le facteur eIF2 lié a un GTP ainsi que le Met-ARNt (code pour codon START) se lient au 40S au niveau du site P
le facteur eIF3 deja lie a 40 S se situe au niveau du site E

Comment se fixe le complexe de pre-initiation a l’ARNm ?
Une fois les facteurs eIF4 (4A, 4B, 4E, 4G) se lient sur le complexte, le complexe peut se lier a la coiffe de l’ARNm
Un des facteurs doit faire quelque chose juste apres l’association du complexe a l’ARNm, lequel c’est et que doit il faire ?
L’helicase (eIF4A) doit defaire les structures secondaires de l’ARNm → pseudo-noeud, tige-boucle, helix, qui sont des repliements
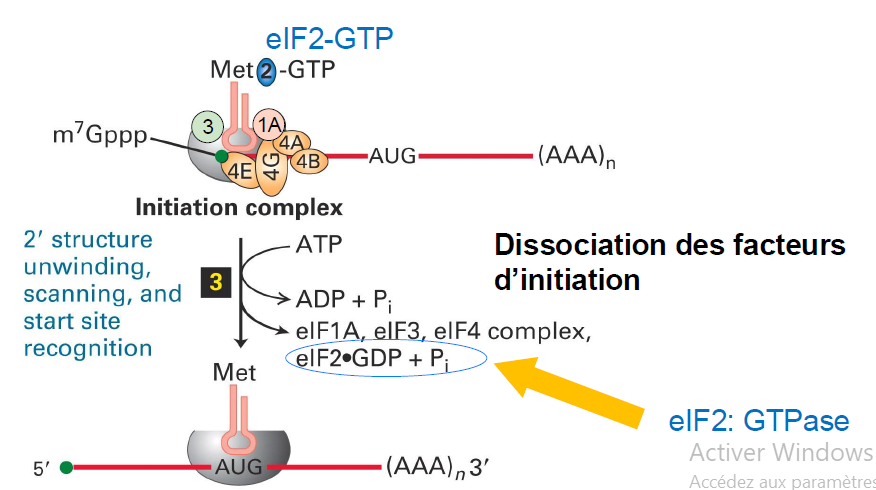
Une fois que le complexe s’est associe a l’ARNm, que fait il ?
Il scanne la partie 5’ UTR de l’ARNm pour identifier le codon start
Comment sait il quand c’est la sequence de debut ?
Il repere la sequence de Kozak, 5’-ACCAUGG-3’
A quoi servent les nucleotides 5’-A et G-3’ ?
Ce sont des nucleotides determinants pour l’efficacite de l’intiation
Que se passe t il une fois que le codon START est repere ?
Le complexe arrete le scan et le facteur GTP associe a eIF2 est hydrolyse en GDP.
Tous les facteurs d initiation se detachent.

Qui hydrolyse les GTP ?
Les GTPase
Qui possede cette activite GTPase ?
les facteurs comme initiation eIF
La GTPase peut etre sous deux formes, lesquels ?
Sous forme active ou inactive, comme un switch (active = hydrolyse GTP)
De quelle famille fait partie les GTPase ?
De la famille des proteines G
Par quoi se complete le complexe d’initiation ?
L’association de la sous unite 60S. Cela se fait grace a :
l’hydrolse du GTP associe a eIF5 (etait lie a 60S)
tous les facteurs lies a 60 S (eIF5 et 6 ainsi que GDP) se separent
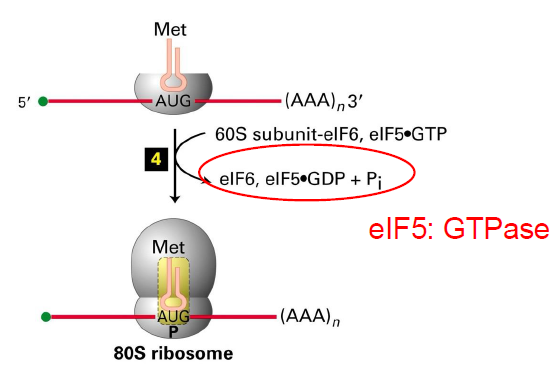

Sur quel site est situe Met-ARNt apres la phase d initiation ?
site P
Quelles sont les etapes du processus d’elongation ?
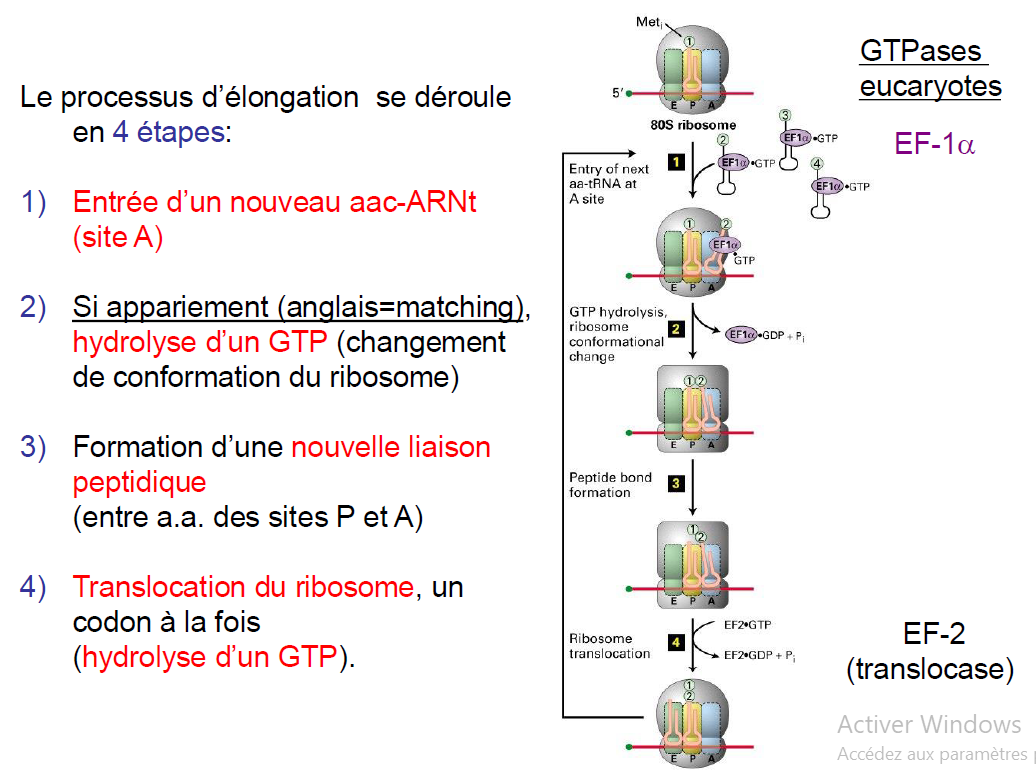

A quoi sert le facteur EF-1alpha ?
Facteur d’elongation qui permet de :
proteger la liaison ester entre l’ARNt et l’acide amine d’une hydrolase
faciliter l’interaction entre l’aminoacyl-ARNt et le ribosome
A quoi sert le facteur EF-2 ?
Permet la translocation du ribosome (deplacement dans le sens 5’→3’
Que se passe t il quand le facteur EF-1alpha hydrolyse GTP ?
Cela change la conformation du ribosome ainsi que celle de EF-1alpha qui lui permet de se liberer
C’est quoi aac-ARNt ?
aminoacyl-ARNt la nouvelle liaison ARNt et aa
Que se passe t il si l anticodon de l’ARNt n’est pas complementaire du codon de l’ARNm ?
L’hydrolase du GTP associe a EF-1alpha ne se fait pas, et donc le ribosome ne change pas sa conformation
C’est quoi l’etape de lecture-correction ?
Le fait que le ribosome verifie que l’aac-ARNt apporte est correct
Quels sont les sites du ribosome ?
site E = exit
site P = peptidyl
site A = aminoacyl / entree
Quels sont les etapes de la translocation vis a vis des ARNt ?
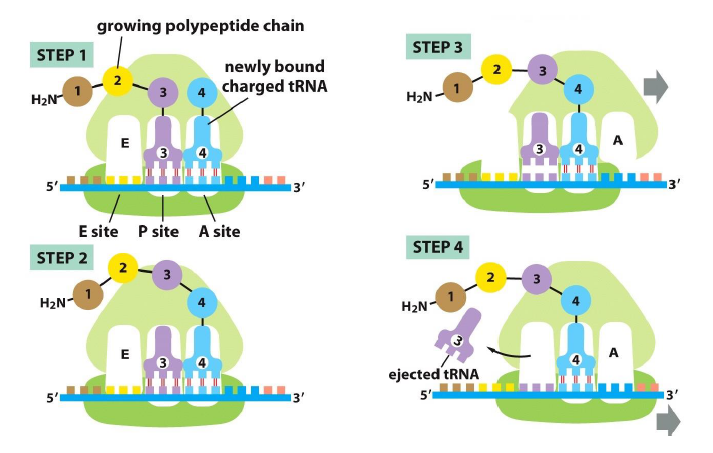

dans quel sens du polypeptide se fait l addition des aa ?
Se fait un par un a l extremite C-terminal
D’ou vient l’energie permettant l’elongation ?
1 ATP par aac-ARNt genere
2 GTP par aa ajoute
De qui est dependante la terminaison ?
De facteurs specifiques de liberation eRFs : eRF1, eRF2 et eRF3
A quoi servent les differents facteurs specfiques de liberation ?
eRF1 et 2 servent a reconnaitres les codons stop sur l’ARNm
eRF3 est associee a eRF1 et au GTP. Elle permet l’hydrolyse de la GTP pour l’energie
Comment est permis le clivage du dernier aa du polypeptide ?
Grace a une molecule d’eau attache a eRF1 qui l’amene au centre catalytique du ribosome
Que permet l hydrolyse du GTP associe au eRF3 ?
Clivage de la chaine polypeptidique et dissociation des facteurs de liberation 1,2 et 3
Y a t il des autres facteurs qui interviennent dans la terminaison et si oui a quoi servent ils ?
Oui des facteurs supplementaires qui :
favorisent la dissociation du ribosome en ses sous unites
empechent la reassociation des sous unites en l absence d un complexe d initiation
Que peut nous permettre de synthetiser beaucoup de la meme proteine assez vite ?
Des polyribosomes, groupement de multiples ribosomes, traduisent une seule molecule d’ARNm
Le recyclage rapide des sous unites du ribosome apre avoir termine la traduction d une proteine.
Quelle forme adopte les polyribosomes et pour quelle raison ?
Forme cyclique qui permet de lire l’ARNm plus efficacement
Quelle est la rapidite de traduction des procaryotes vs eucaryotes ?
procaryotes → 20 aa par seconde
eucaryotes → 4 aa par seconde
Qu est ce que mTOR ?
C’est une kinase qui permet de former un complexe avec Raptor et GbetaL.
Ce complexe peut mesurer la disponibilite des nutritives et d energie dans la cellules pour decider si elle fait differentes choses, comme la synthese de proteines. Elle decide de la Cell growth
Qui peut inhiber mTOR ?
Un agent immuno-suppresseur appele rapamycine qui est produit par des bacteries
Comment est active mTOR ?
Active par des facteurs de croissance et des hormones comme l insuline
Que font les antibiotiques au niveau de la bacterie ?
Inhibent la synthese de leur proteines
Chez les ribosomes procaryotiques, quels antibiotiques peuvent les inhiber et quelle sous unite du ribisome ils inhibent ?
Chloramphenicol et erythromycin inhibent 50S
Tetracycline inhibe 30 S
Chez les ribosomes eucaryotiques, quels antibiotiques peuvent les inhiber et quelle sous unite du ribisome ils inhibent (quelle partie de ce sous unite) ?
Cycloheximide se lie a 60S et inhibe eEF2
Quel anitbiotique qui inhibe les ribosomes procaryotiques peut etre mauvais pour les eucaryotes et que fait il ?
Le chloramphenicol qui bloque les ribosomes des mitochondriess eucaryotiques
Quelle autre antibiotique peut inhiber la synthese proteique et comment le fait elle ?
La puromycine qui a une structure similaire a l extremite 3’ de aac-ARNt, va pouvoir se fixer au site A du ribosome et inhiber l entree d’un aac-ARNt. Elle va se lier au polypeptide et faire une terminaison prematuree de la proteine naissante
Qu est ce que la configuration native de la proteine ?
Son repliement
Quand commence le repliement de la proteine ?
Peut commencer pendant la synthese
Qui a les informations qui specifient la structure native de la proteine ?
La structure primaire de la proteine (sequence aa)
Quelle autre molecule permet de replier les proteines et pourquoi on a besoin d elle ?
Les chaperones moleculaires car le processus spontane de repliement est lent et incomplet
Quelles sont les deux familles de chaperones moleculaires ?
La Hsp 70 et GroEL/ES (procaryotes) ou Hsp 60/Hsp10 (eucaryotes)
Comment fonctionne Hsp 70 ?
Permet de lier la chaine polypeptidique naissante entre elle.
Quand elle se lie a l’ATP, Hsp 70 est sous forme ouverte exposant une poche hydrophobe qui peut lier les regions hydrophobes de la chaine polypeptidique. L’ATP est hydrolyser en ADP pour permettre le fonctionnement.
Une proteine denature (mal repliee) peut etre naturee par Hsp 70
Quelle est la structure de GroEL/Hsp 60 ?
Forme de baril constituee de 2 chambres contenant chacun 7 GroEL.


Comment fonctionne GroEL/ES et Hsp60/10 ?
Le nouveau polypeptide entre dans la chambre de la chaperonne et GroES/Hsp10 vont former un couvercle ce qui va permettre a la proteine de se replier dans un endroit protege.
Quelle est l energie necessaire au fonctionnement de GroEL/ES et Hsp60/10 ?
1 ATP pour fixer le chapeau et 1 ATP pour enveler le chapeau pour que polypetide sorte
Quelle est la difference entre Hsp60/10 et Hsp70
Hsp 70 peut commencer pendant que le polypeptide est entrain d etre synthetiser
Par quoi est defini le niveau des proteines ?
Par leur taux de synthese et degradation. (Bcp synthese peu degrade → haut niveau)
Quelles types de proteines sont instables ?
Les proteines avec des fonctions regulatrices
Quelles sont les proteines stables ?
Avec une fonction constitutive ou structurelle (hemoglobine ou crystalline)
Duree de vie stable vs instable
Stable peut durer jusqua la fin de vie de la cellule
Instable peut durer une dizaine de minutes
Quelles sont les deux voies utilisees pour la degradation cellulaire ?
voie ubiquitine-proteasome
lysosomes
Quels types de proteines degrade la voie ubitiquine-proteasome ?
Les proteins cytosoliques (dans le cytosol)
Les proteines qui sont mal repliees
De quoi est forme un lysosome ?
Organite rempli d’enzymes hydrolitiques (proteases)
Quels types de proteines sont degrades par les lysosomes ?
Les proteines transmembranaires (dans la membrane), extracellulaires et des proteines d’organites agees ou defectueuses
Quel est le fonctionnement de l’ubitiquine ?
L’ubitiquine se lie a la proteine cible
Comment se lie l ubitiquine a la proteine cible ? (etapes)
Elle se lie de maniere covalente avec sa proteine cible (modification post-traductionnelle). C’est le groupe carboxylate C-terminal de l’ubitiquine qui se lie a la lysine de la proteine cible. Voici les etapes :
ubitiquine activation : formation liaison thioester entre E1 et ubiquitine grace a l’ATP qui permet d’activer l’Ub
ubtiquine conjugation : E1 remplace par E2 avec denouveau une liaison thioester. E2 sert de transporteur de Ub
ubitiquine ligase : E3 choisit le substrat et permet de catalyser la liaison entre ubitiquine et subsrat
E1,2,3 sont des enzymes
Quelle autres modifications post traductionnelles sont possibles au niveau de l’ubitiquine ?
L’ubitiquine peut etre soit unique → monoubiquination, soit une chaine polyubiquitine apres la liaison a la proteine cible.
Ou peuvent se lier les autres ubitiquine a la principale ?
L’ubitiquine a 7 residus de lysine et un N-terminus qui peuvent etre des points d ubiquitination.
A quoi peuvent ressembler les chaines d’ubitiquine ?
Lineaires ou branchees
Quelles sont les 2 chaines auxquelles nous nous interessons ?
K48 et K11
A quoi ressemble la chaine K48 ?
Plusieurs ubitquines lineaires liees par la lysine 48