Videregående biokemi og genetik - modul 4.2
1/192
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No analytics yet
Send a link to your students to track their progress
193 Terms
Nukleus
Den er omgivet af en dobbelt lipidmembran kaldet nukleær envelope, som har porer til transport af RNA og proteiner. Den indeholder kromatin, som består af DNA og associerede proteiner som kaldes for histoner.
Funktioner:
Opbevaring af genetisk information (DNA)
Styrer genekspression via regulering af transskription af mRNA
Organiserer celledeling ved at kopiere og fordele DNA til datterceller
Nukleolus
Er en tæt, sfærisk struktur inde i nukleus, som ikke har en membran.
Funktion:
Danner rRNA
Samler ribosmale underenheder
Produktion af protein som er involveret i celledeling
Ribosomer
Består af en lille og en stor subunit, som er sammensat af rRNA og proteiner. Den kan enten være frit flydende i cytoplasma eller bundet til rER.
Funktion:
- Oversætter mRNA til proteiner via proteinsyntese
- Frie flydende producerer proteiner til brug i cellen, mens bundet producerer proteiner til sekretion eller membraner
Ru endoplasmatisk reticulum
Er et netværk af flade membraner, som er forbundet med nukleus ydre membran. Den er dækket af ribosomer som giver et ru udseende.
Funktion
Dannelse af proteiner, især dem der skal eksporteres fra cellen eller indgå i cellemembranen
Foldning af proteiner
Modtagelse af post-translationelle modifikationer
Transport til Golgi-apparatet
Glat endoplasmatisk reticulum
Ligner rER, men mangler ribosomerne, hvilket giver den glatte udseende.
Funktion:
Lipidsyntese
Afgiftning
Calciumlagring
Golgi-apparatet
Er en stak af flade membransække kaldet cisternae. Den har en cis-side som modtager proteiner fra rER, og en trans-side som sender modificerede proteiner til deres endelige destination.
Funktion:
Modtager, modificerer og sorterer proteiner og lipider
Tilføjer kulhydratgrupper (glukosylering)
Tilføjer fosfatgrupper (fosforylering)
Pakker proteiner i vesikler til sekretion, lysosomer eller cellemembranen
Sekretorisk granula
Små, membranbundne vesikler fyldt med proteiner, hormoner eller enzymer. De findes især i endokrine celler og nerveceller
Funktion:
Transporterer og frigiver stoffer via eksocytose
Lysosomer
Er membranbundne vesikler fyldt med hydrolytiske enzymer såsom proteaser, lipaser og nukleaser.
Funktion:
Nedbrydning og genbrugning af skadede organeller, proteiner og indtagne partikler.
Opretholdelse af lav pH (5) ved hjælp af protonpumoper
Deltager i autofagi, nedbrydning af cellens egne strukturer, og fagocytose
Peroxisomer
Er membranbundne organeller, som indeholder oxidative enzymer.
Funktion:
Nedbryder fedtsyrer gennem beta-oxidation og neutraliserer giftige stoffer ved at omdanne brintoverilte til vand vha. katalase
Centrioler
Er cylindriske strukturer som er dannet af mikrotubuli, som er arrangeret i en 9+3 struktur.
Funktion:
Organiserer mitosetenen under celledeling og danner basallegemer til cilier og flagceller
Proteasomer
Er store protein-komplekser, som indeholder en central kanal til proteinnedbrydning.
Funktion:
Nedbryder beskadigede eller unødvendige proteiner mærket med ubiquitin.
Regulerer proteinniveau og fjerner fejlbehæftede proteiner
Mikrofilamenter
Intracellulære trådformede proteiner
Plasmamembran, Plasmalemma
Hele cellen omgives yderst af plasmalemma
Funktion: afgrænser cellen, regulerer transport af stoffer ind og ud af cellen
Opbygget som et dobbeltlag af fosfolipider, hvori der indbygget/indlejret proteiner med specifikke funktioner
Kernemembran
Adskillelse af nucleoplasma fra cytoplasma opretholdes af denne membran
Aktinfilamenters funktion
Mikrofilamenter opbygget af G-aktin. Deres funktion er at bevare form, sikre bevægelse, og så fungerer de som et anker til ECM
Cytoplasma
Samlebetegnelse for det cellemateriale der ligger uden for cellekernen og inden for cellemembranen (plasmamembranen). Indeholder cellens organeller
Det centrale dogme
Replikation (DNA), transkription (RNA) og translation (protein)
DNA
deoxyribonukleinsyre, dobbeltstrenget
RNA
ribonukleinsyre, enkeltstrenget
RNA sekundær strukturer
Hairpin
Bulge
Loop
Pseudoknot
RNA er i modsætning til DNA altid enkeltstrenget, men segmenter af RNA-molekylet er ofte komplementære og kan baseparre til intramolekylære dobbeltstrengede strukturer, i særdeleshed hårnålestrukturer. RNA-molekylerne kan således folde til mange forskellige strukturer alt efter nukleotidsekvensen og er et langt mere fleksibelt molekyle end DNA.
Hvorfor er RNA så ustabil?
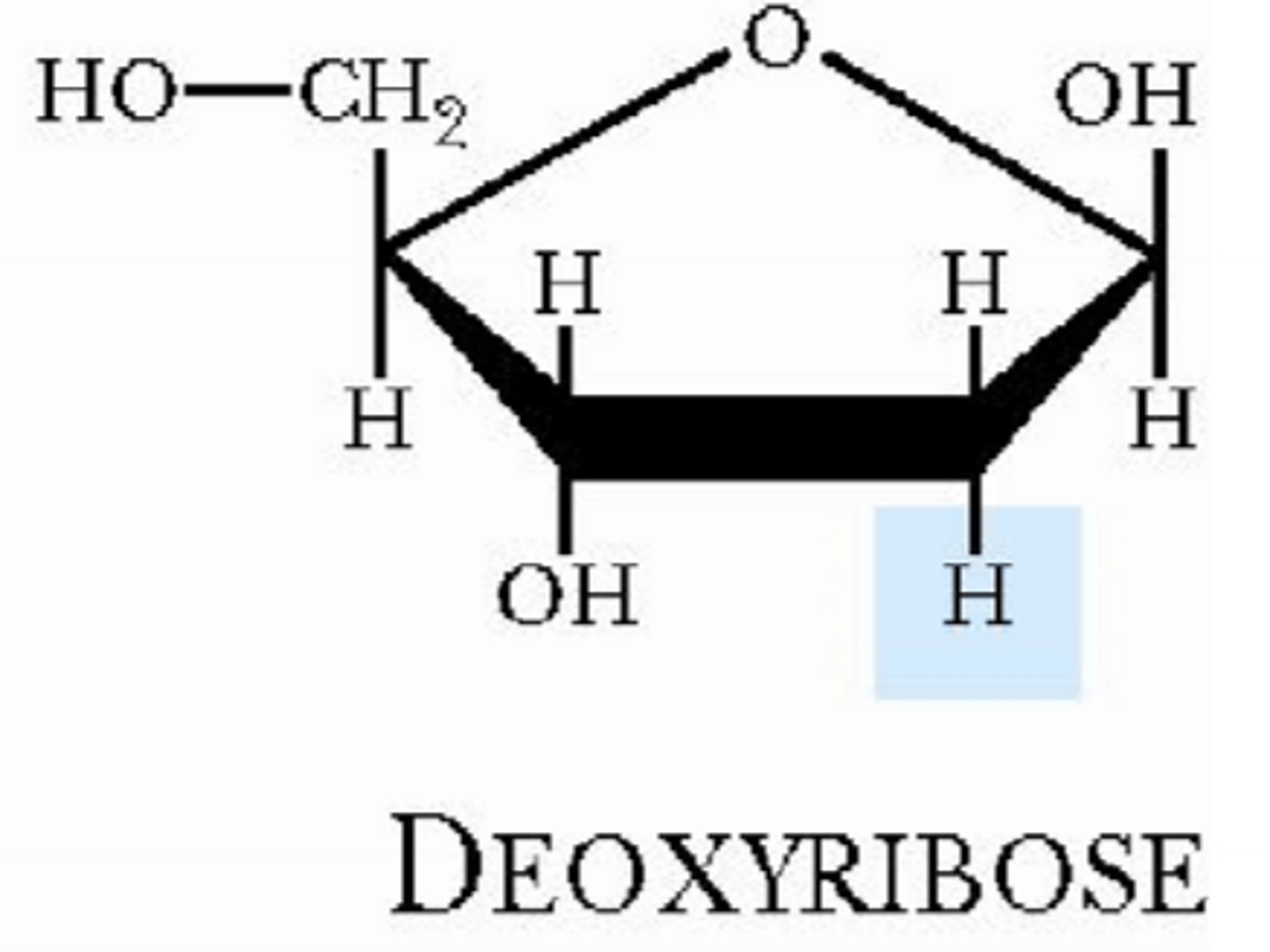
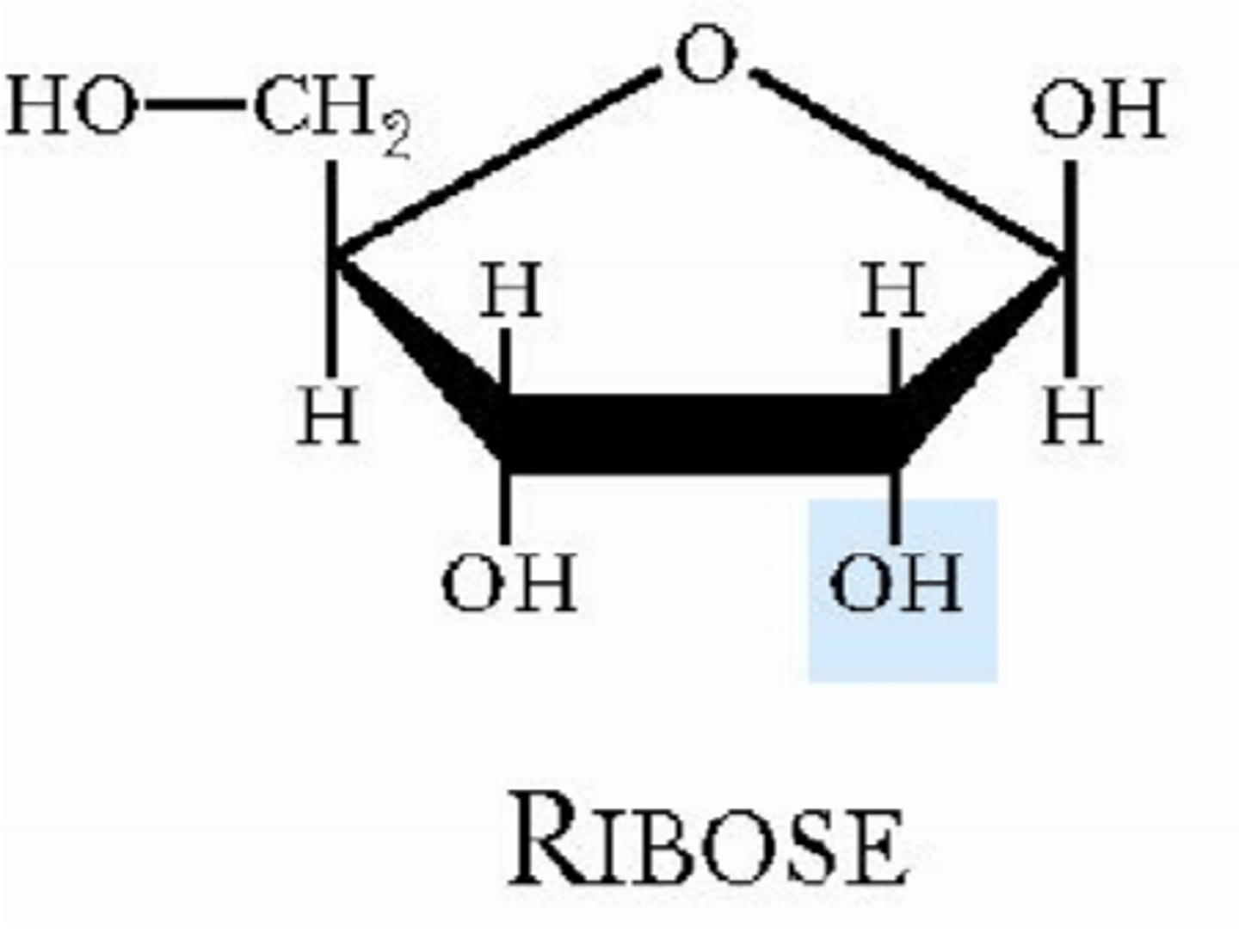
Kulhydratmolekylet i RNA er ribose, der indeholder en ekstra hydroxygruppe (-OH) i forhold til DNA-molekylernes deoxyribose. Den ekstra hydroxygruppe betyder, at RNA-molekyler er mere ustabile end DNA, idet 2'-OH-gruppen kan angribe 3'-5'-fosfodiesterbindingen i nukleotidstrengen og føre til brud på RNA-strengen
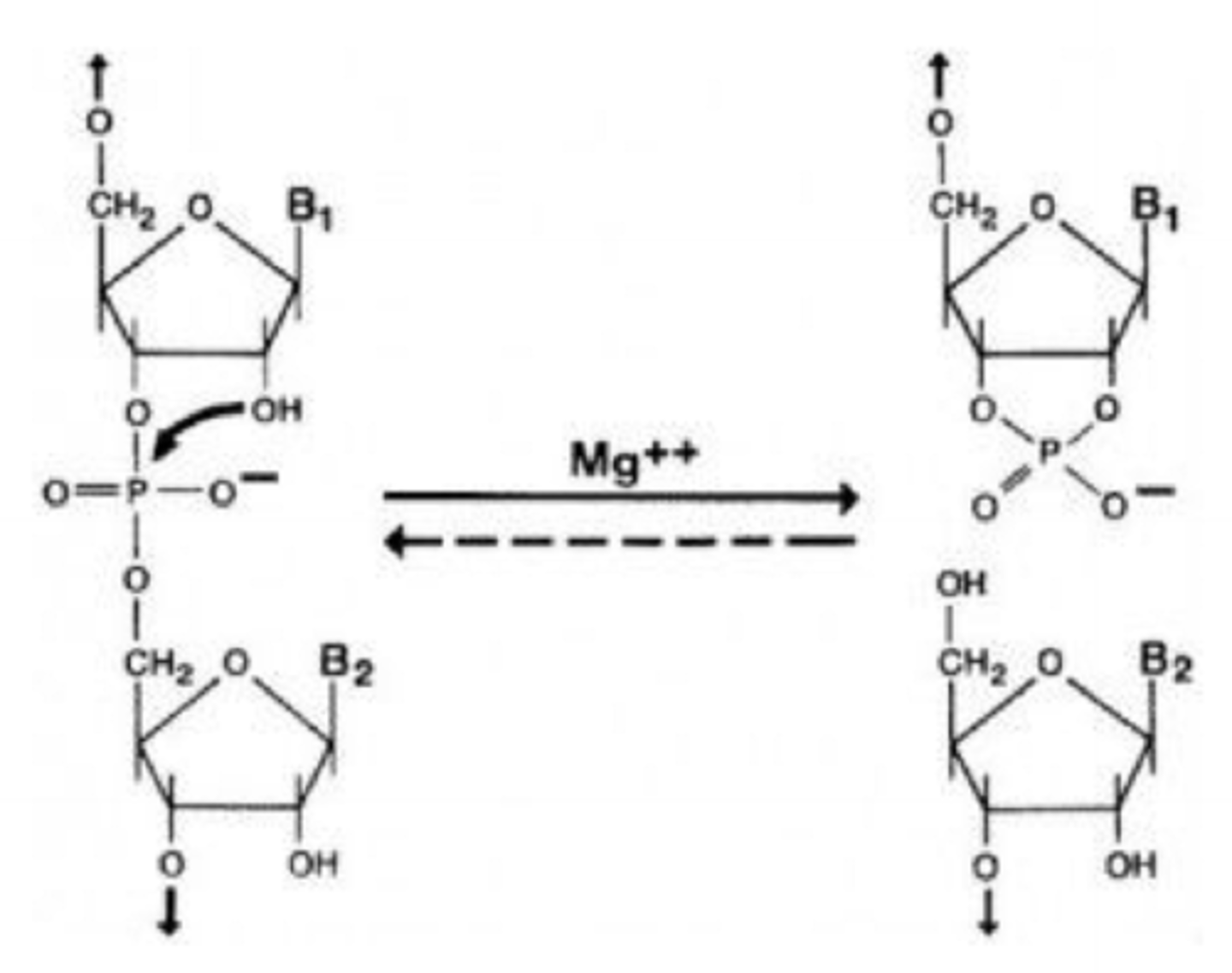
RNase
an enzyme that cuts the sugar-phosphate backbone in RNA
Hvorfor ikke uracil i DNA?
RNA indeholder uracil, hvor DNA indeholder thymin. Thymin er en methyleret udgave af uracil. Tilstedeværelsen af uracil i DNA vil medføre en øget mutationsrate, fordi uracil kan forveksles med en deamineret cytosin. I RNA tolereres brugen af det mindre bekostelige uracil sandsynligvis, fordi RNA i modsætning til DNA ikke er unikt arvemateriale, men altid kan dannes ved transkription af DNA
Deaminering, fjernelse af aminogrupper (-NH2) fra molekyler i organismen. Vigtigst er deaminering af aminosyrer
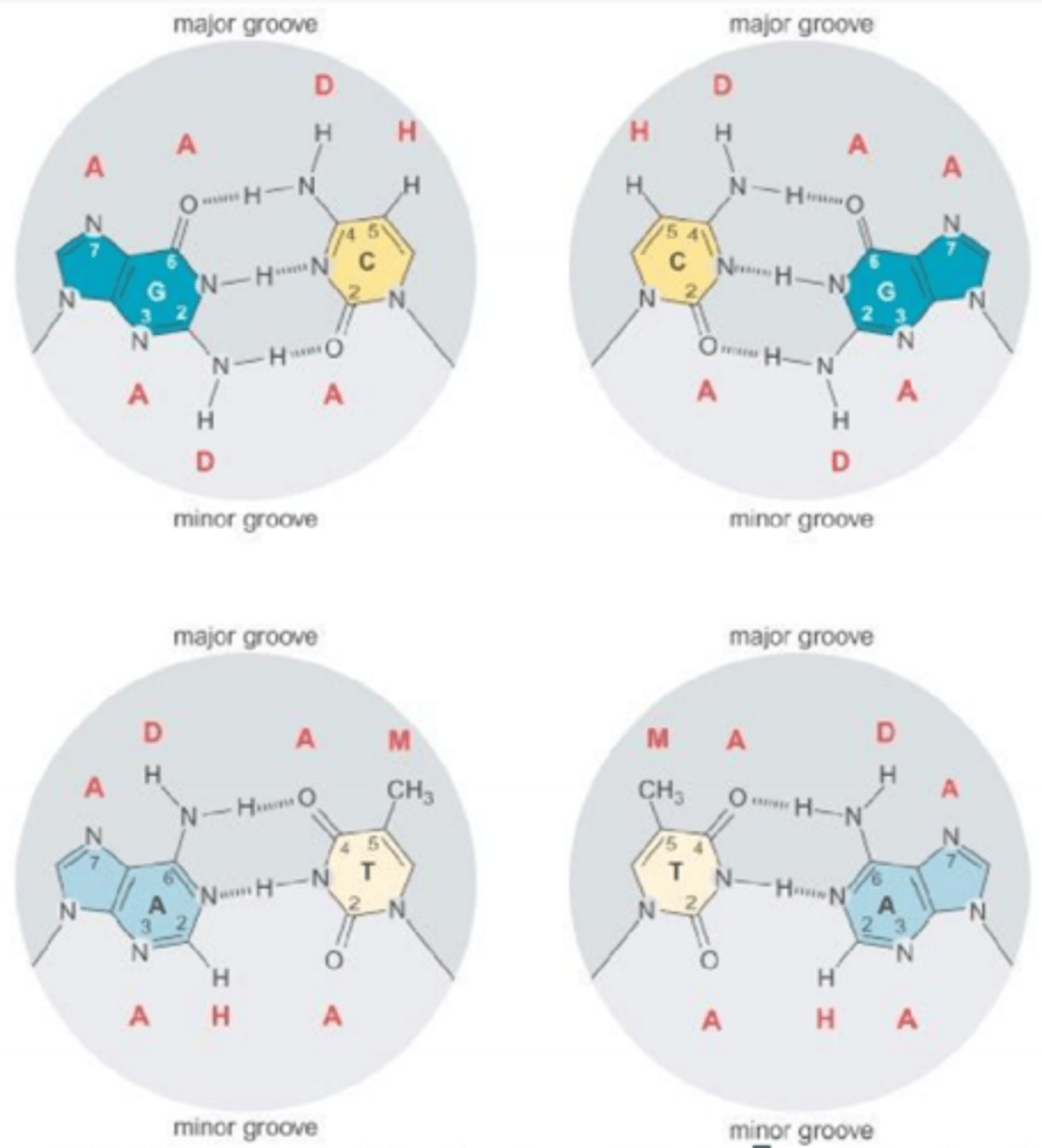
Major groove of DNA
is the main site of protein binding
Major groove kemisk kode:
ADAM- AT par
AADH- GC par
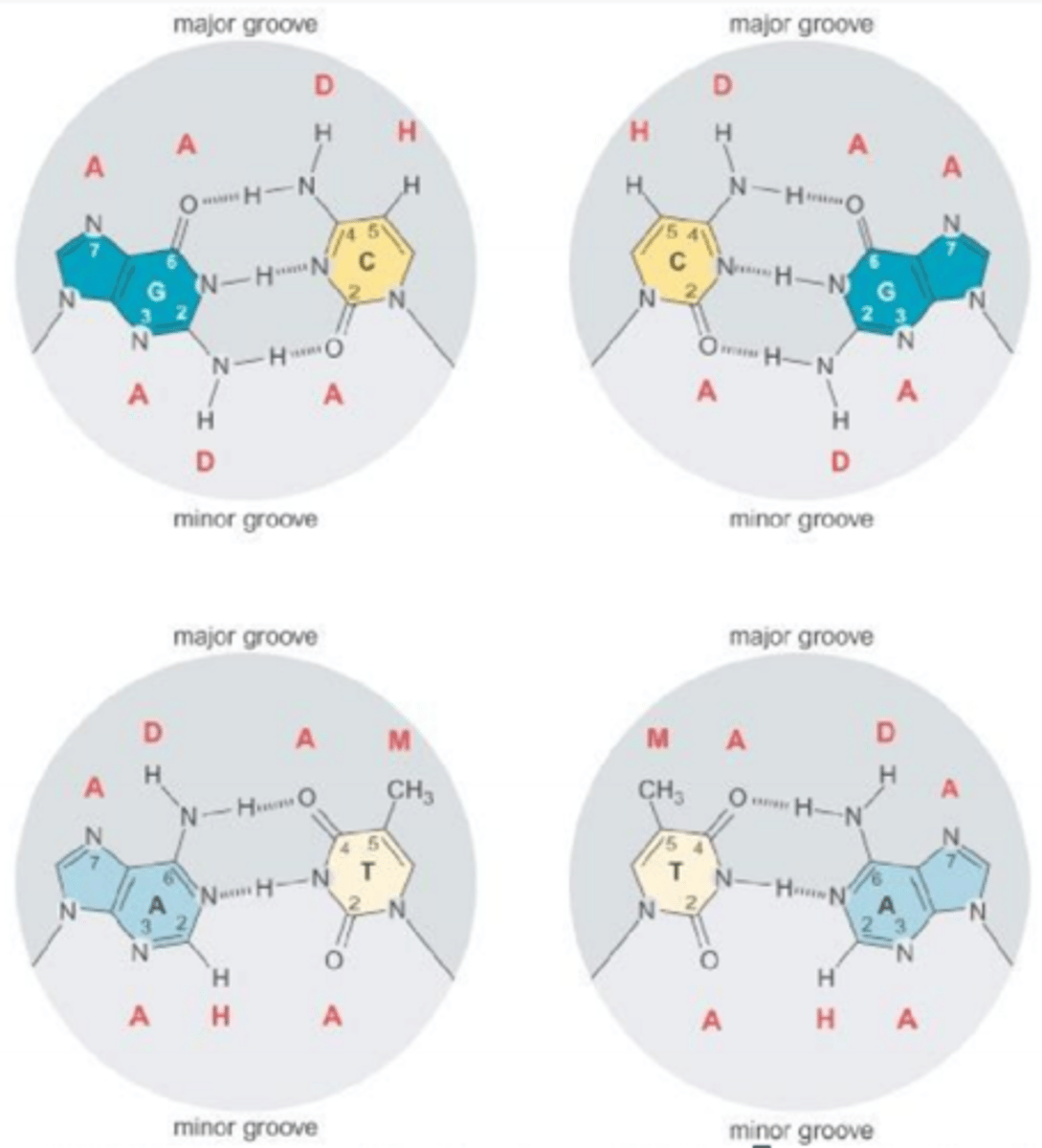
Minor groove of DNA
Area where bases are closer together and not easily accessed
Minor groove kode er ikke præcis nok (kan ikke skelne mellem den kodende og komplementære streng)
B-DNA
Den mest almindelige form
Right-handed
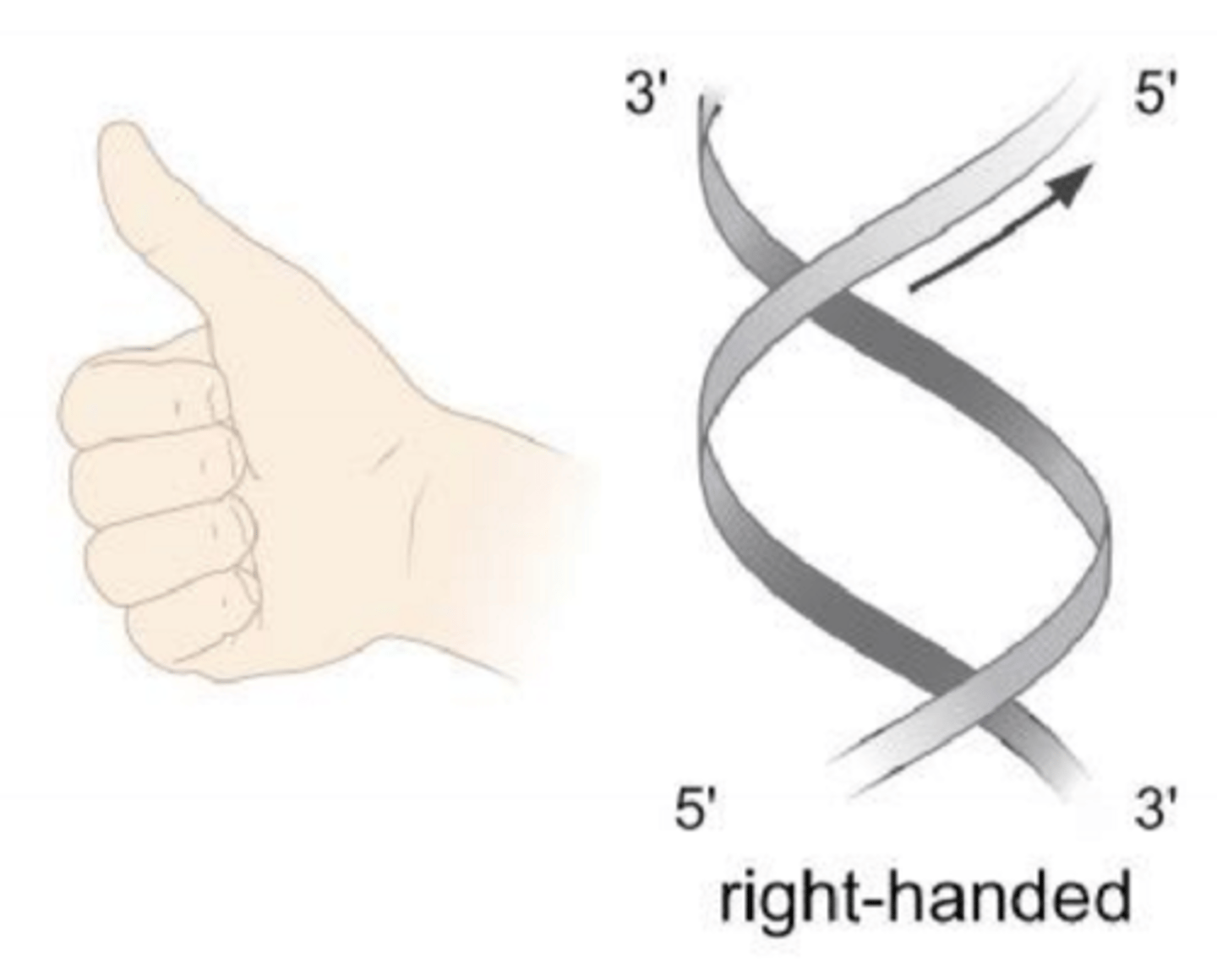
A-DNA
i protein-DNA komplekser; dobbeltstrenget RNA
Right-handed
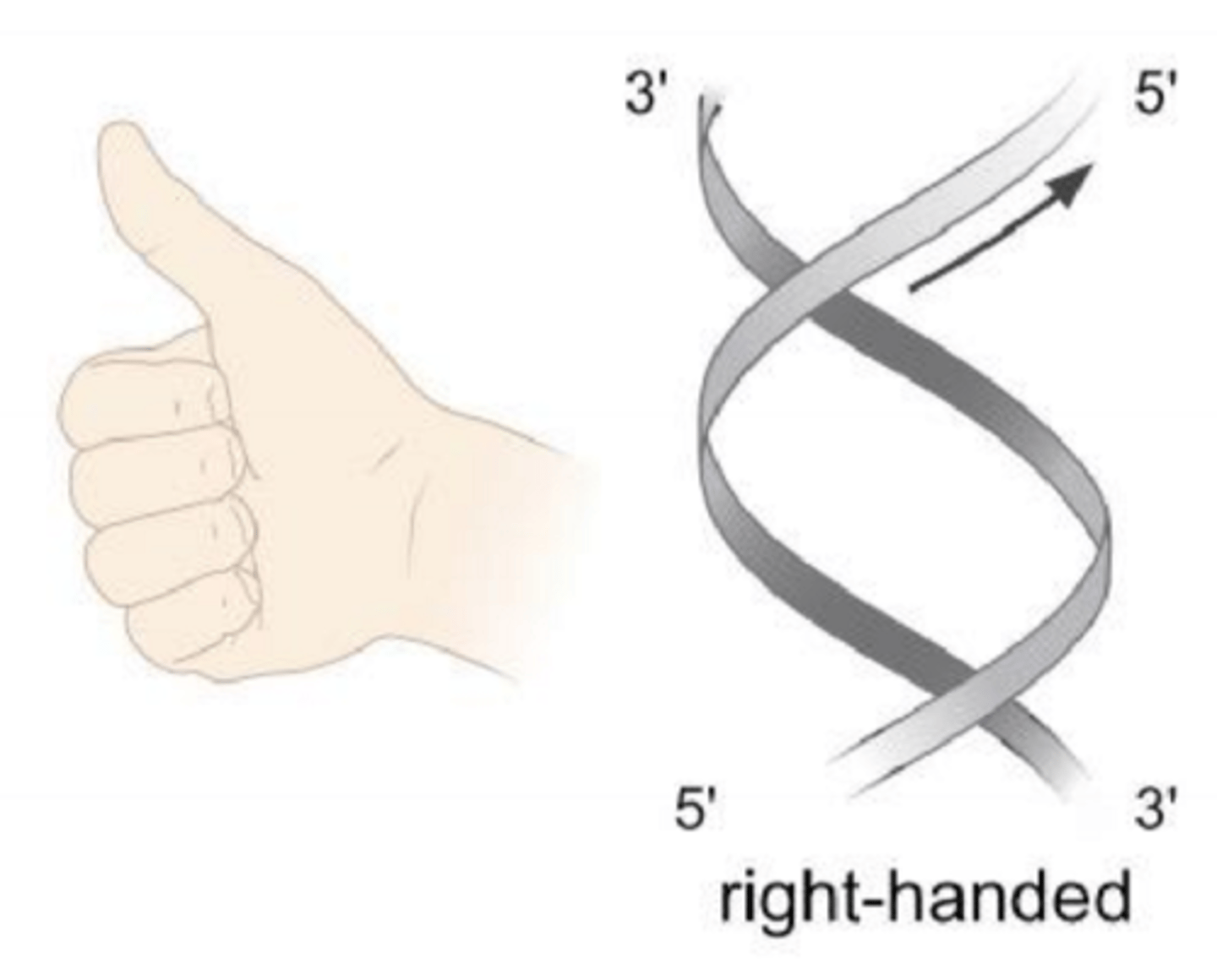
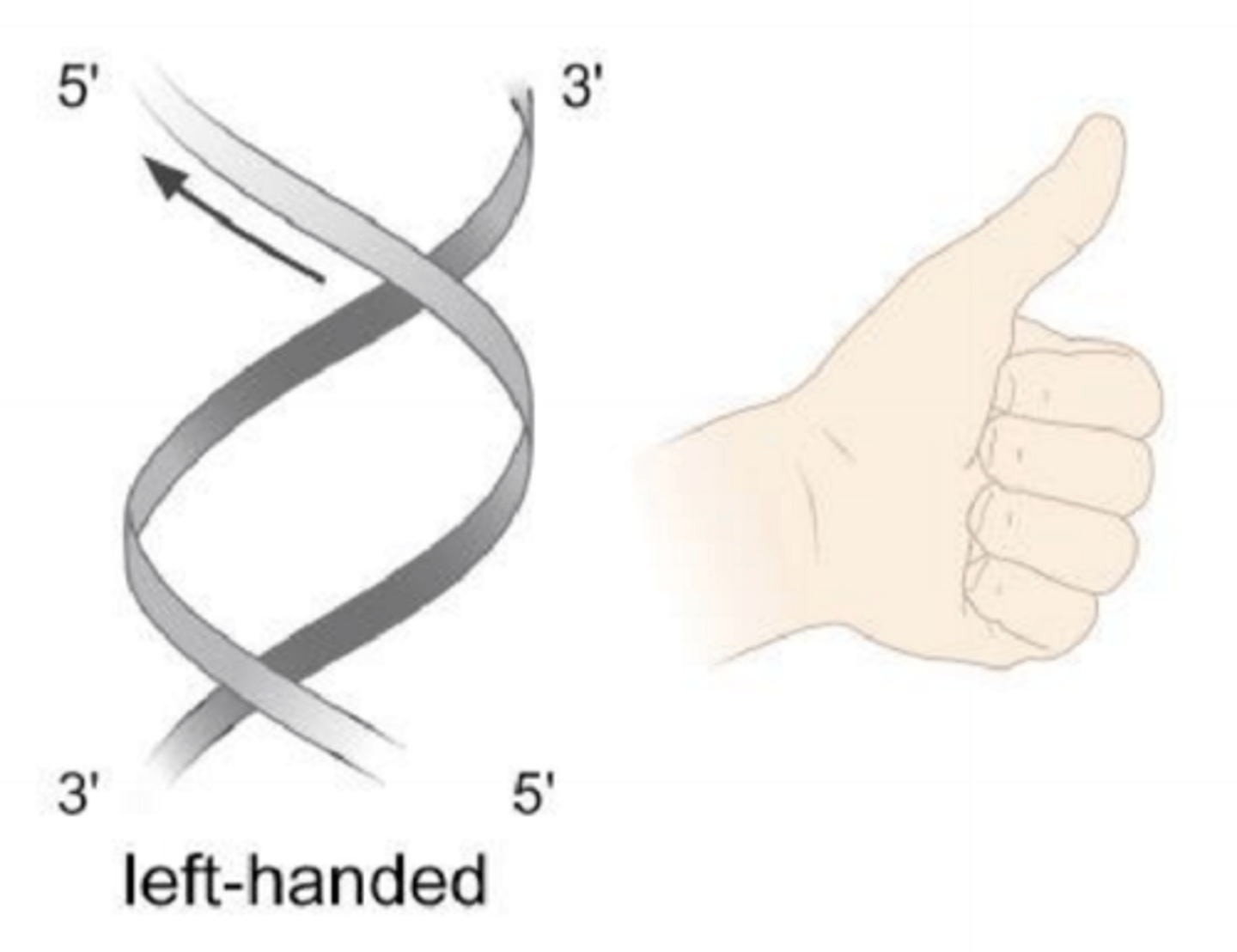
Z-DNA
Zigzag DNA, left-handed; når der er høj koncentration af positive ioner (fx Na+)
Left-handed
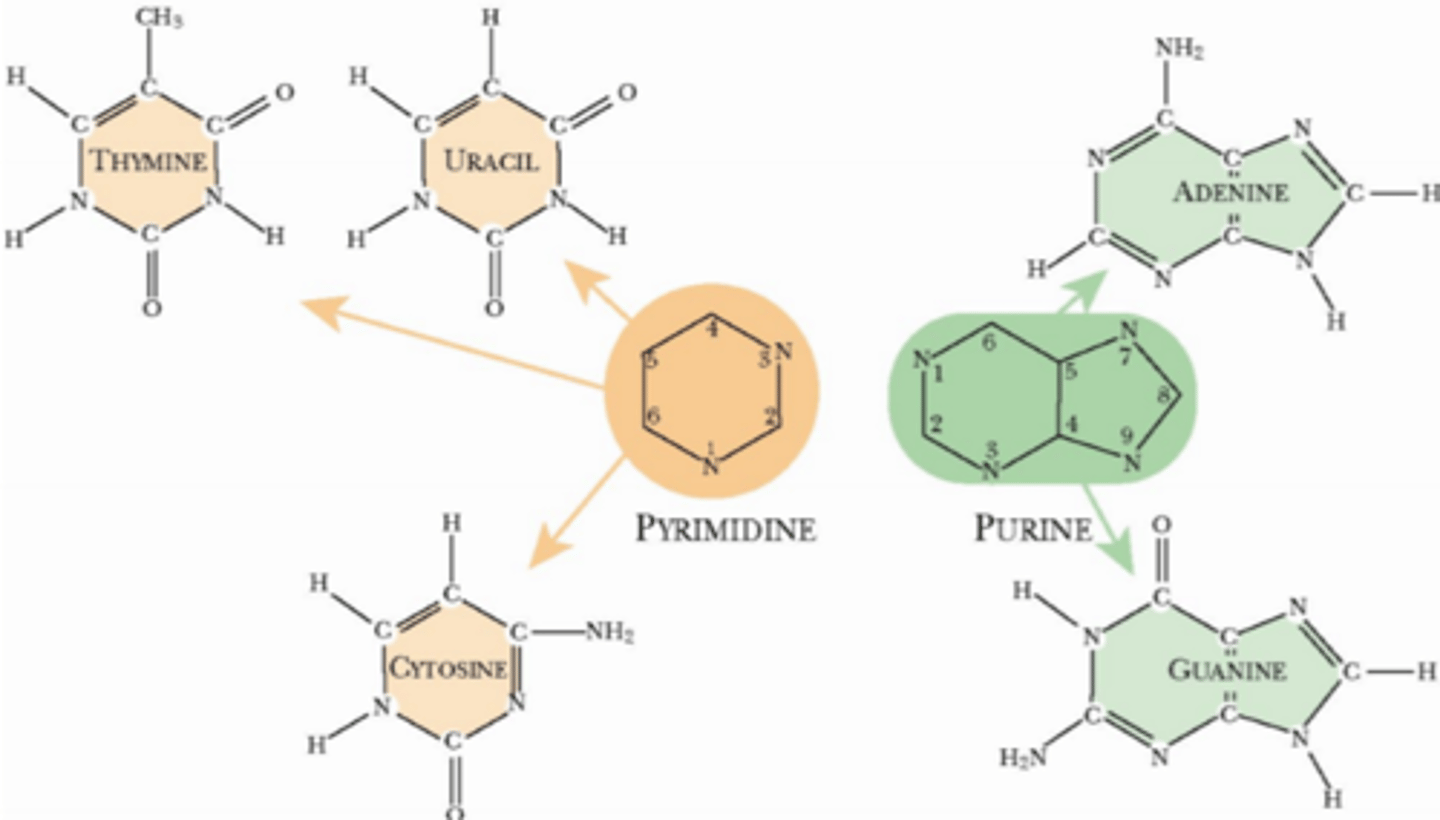
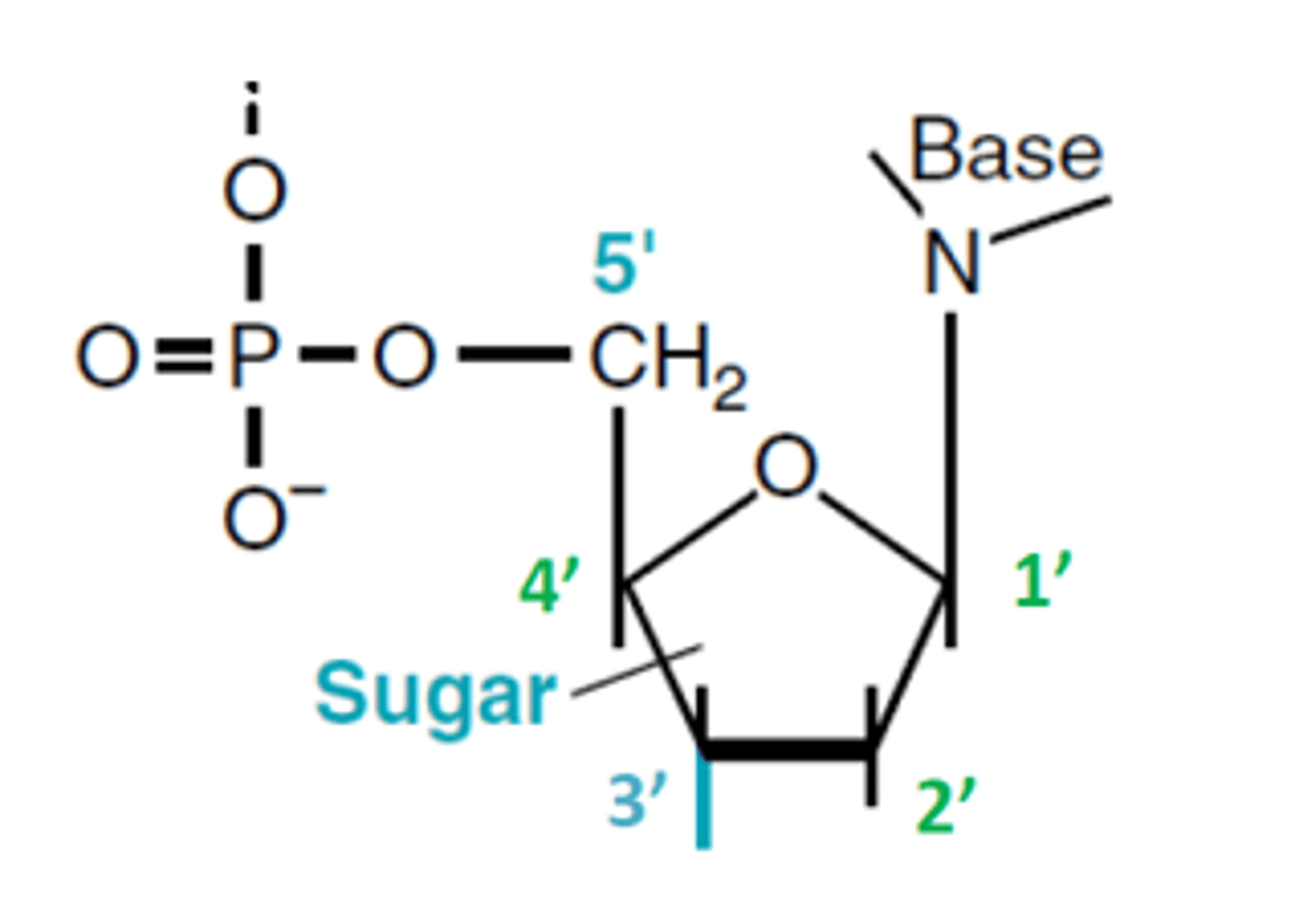
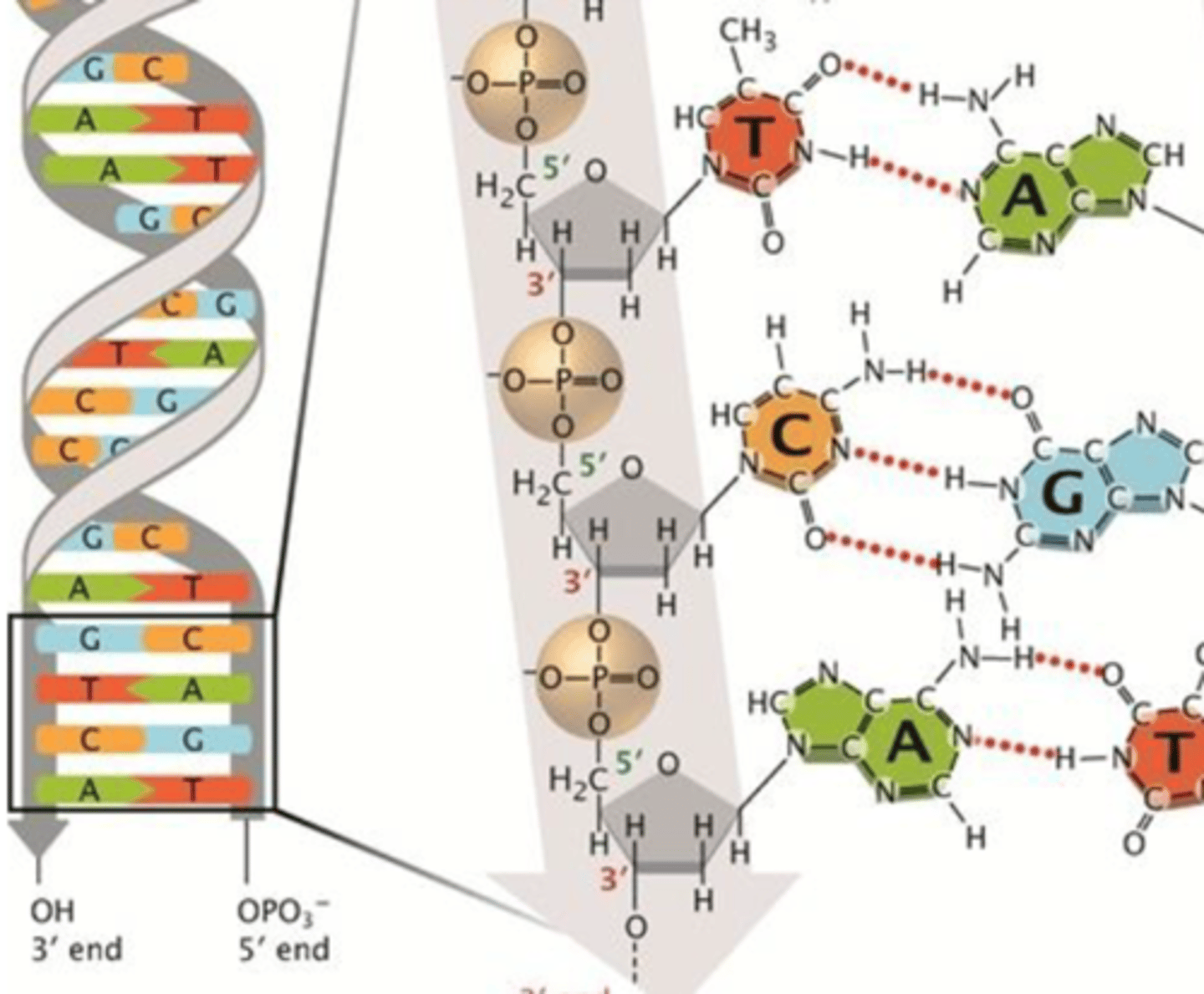
Nukleotid
Et nukleotid består af en fosfatgruppe, en sukkergruppe (deoxyribose: Ringformet pentose) og en base
Fire forskellige baser: Adenin, cytosin, guanin, thymin
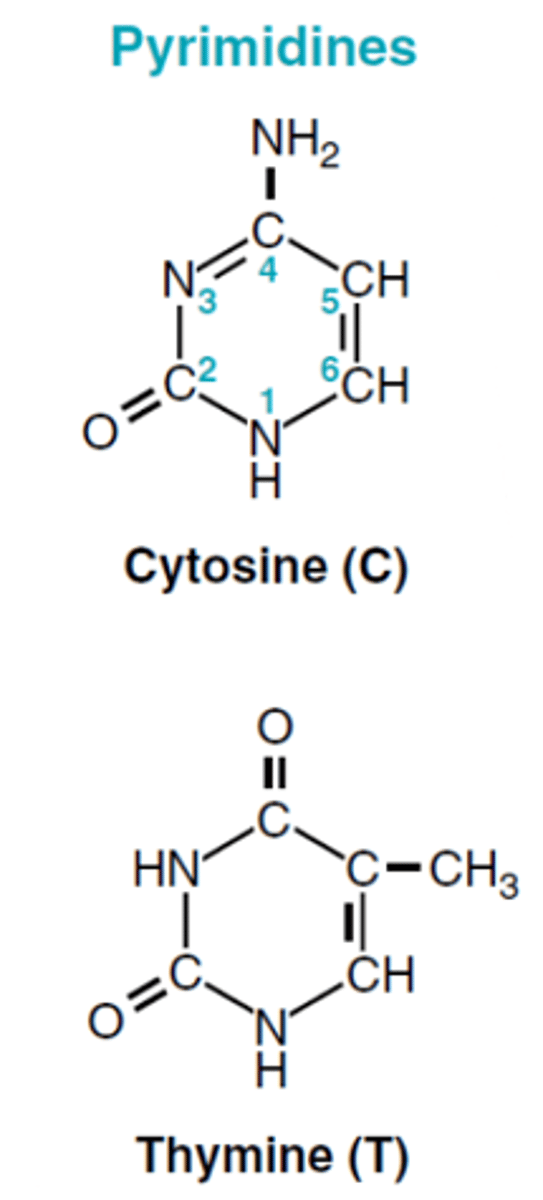
Cytosin
single carbon-nitrogen ringe; pyrimidines
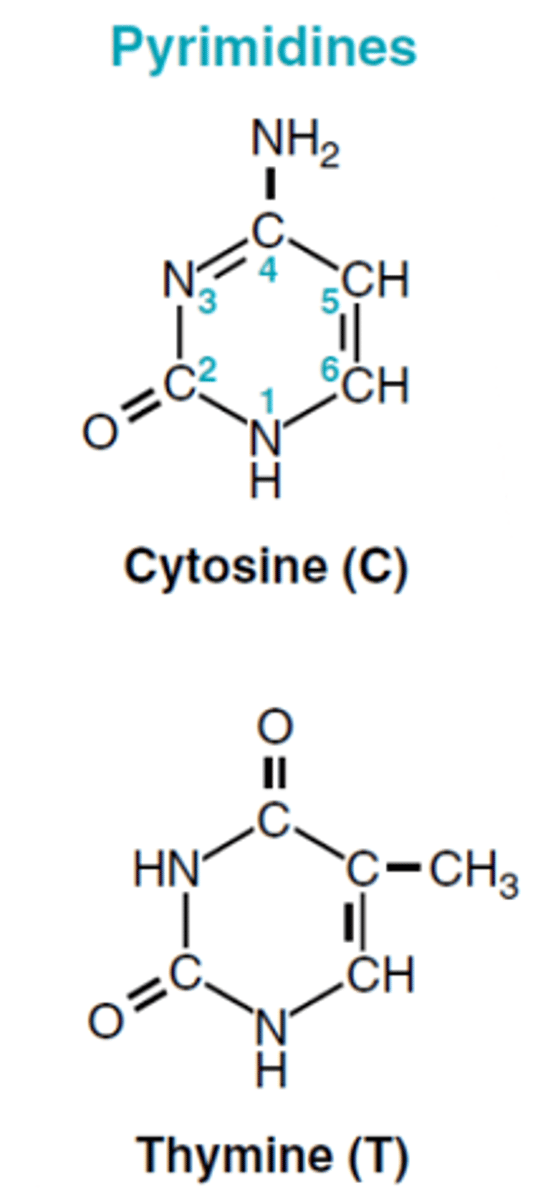
Thymin
single carbon-nitrogen ringe; pyrimidines
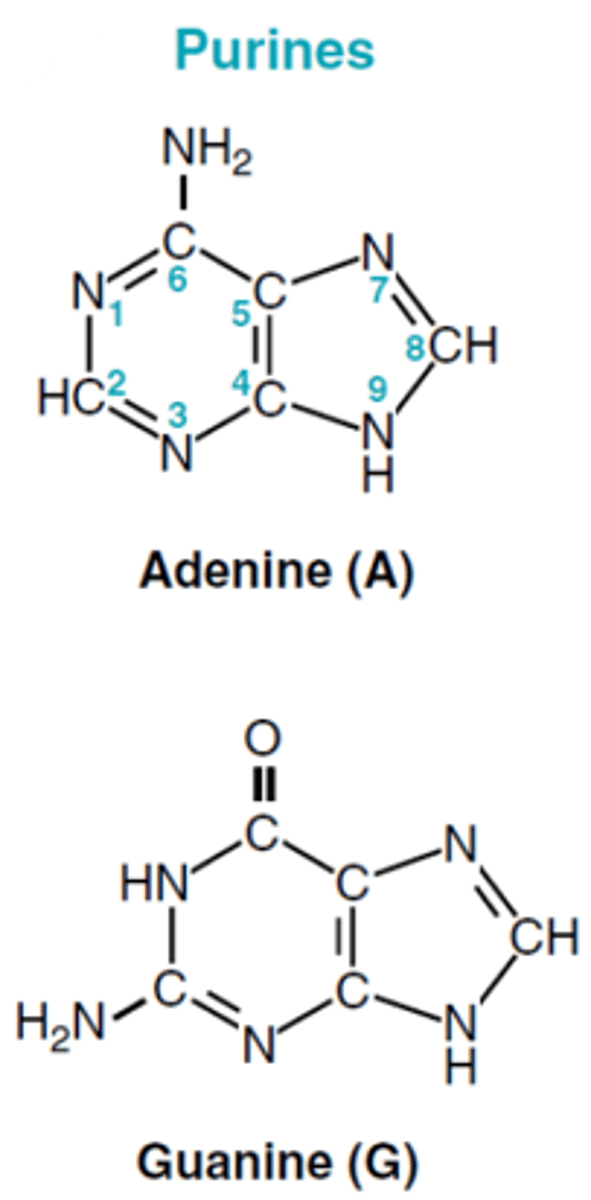
Adenin
double carbon-nitrogen ringe; purines
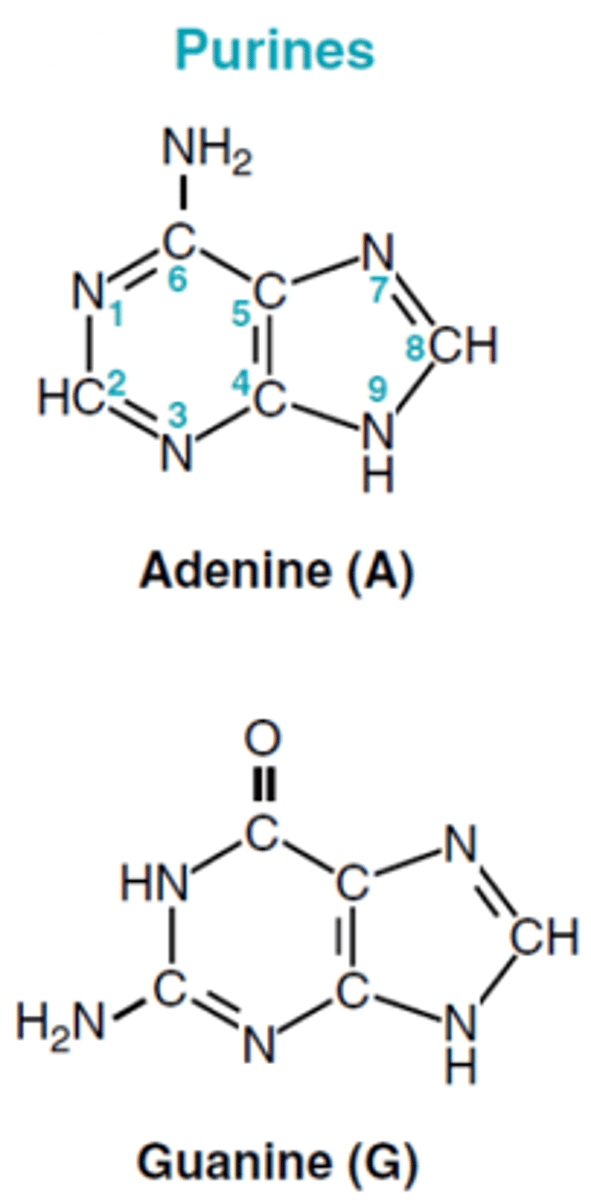
Guanin
double carbon-nitrogen ringe; purines
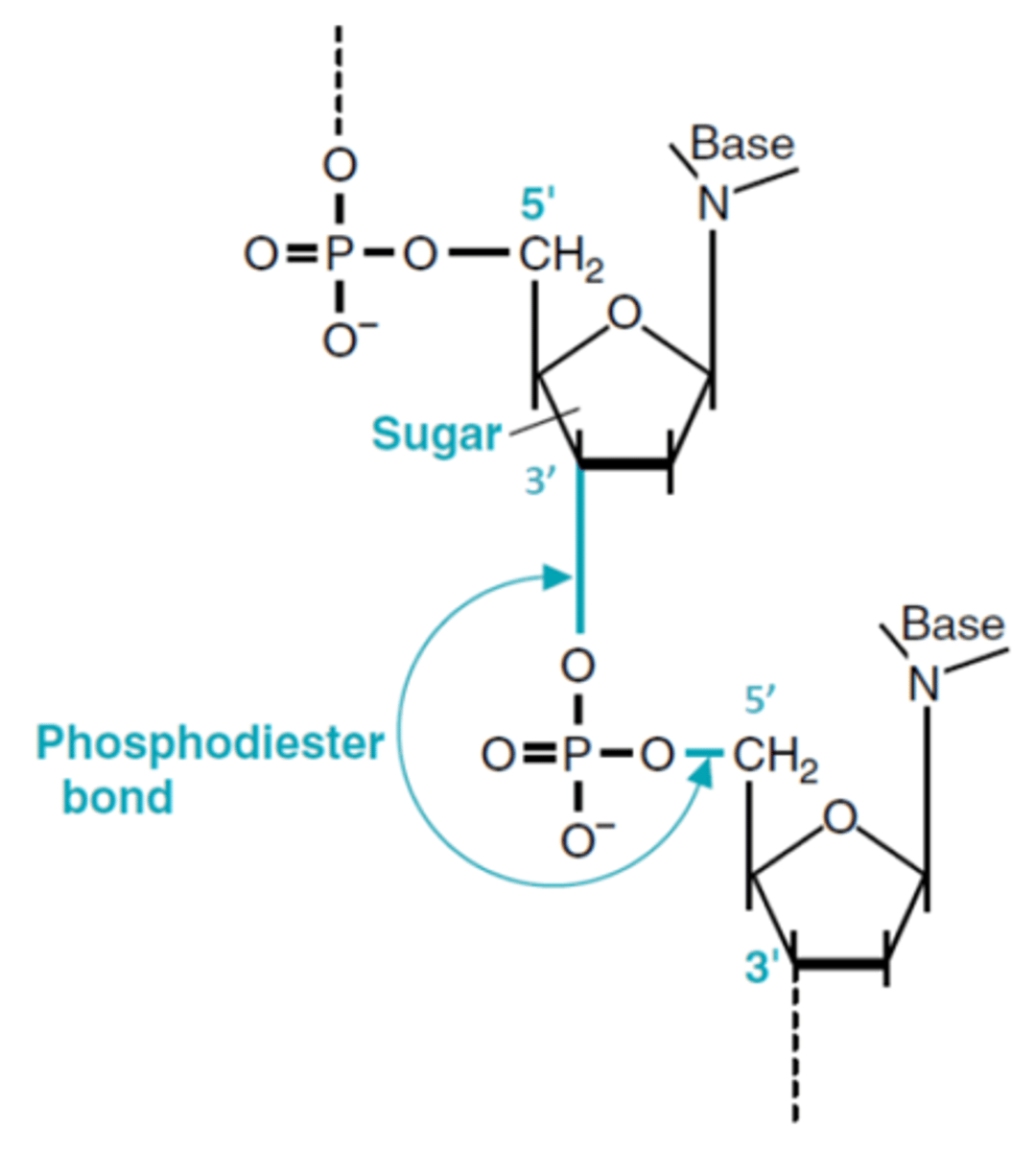
Fosfatgruppen
fungerer som ”linker” og gør DNA-molekylet negativt ladet
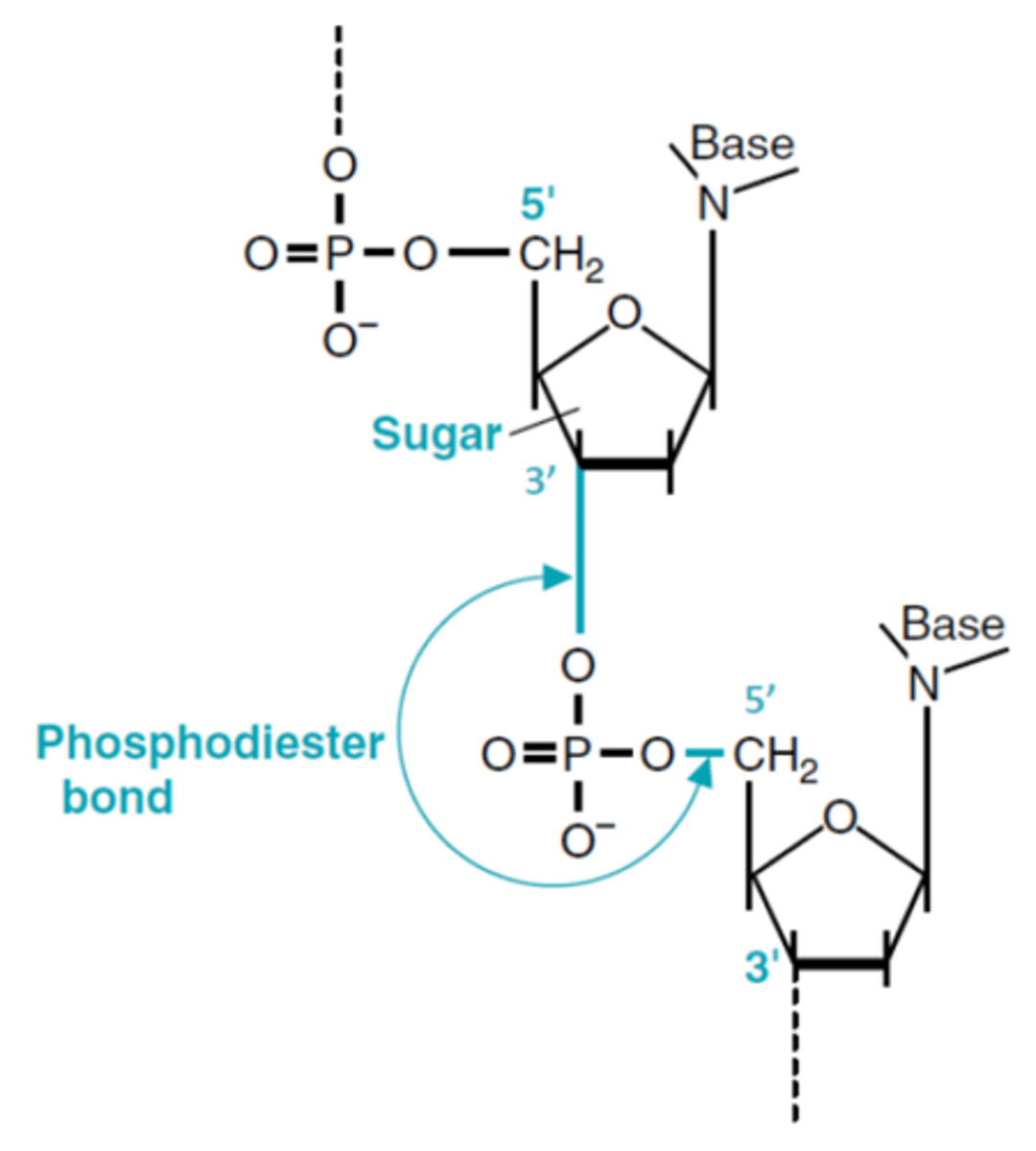
Sukkergruppen
carbon 5 binder den overliggende nukleotids fosfatgruppe og carbon 3 binder til den underliggende fosfatgruppe. 3’,5’-fosfodiesterbindinger binder nukleotider i samme DNA-streng. DNA læses fra 5’ mod 3’!
Hydrogenbindinger
(H-O/N) forbinder baser i modsatte streng!
Adenin og thymin har 2 hydrogenbindinger
Cytosin og guanin har 3 hydrogenbindinger
Resulterer i lidt forskellige strengafstande
Histonoktamer
To af hver af histon H2A, H2B, H3 og H4 er bundet sammen i noget der hedder en histonoktamer
Nukleosomkerne
147 basepar af DNA-molekylet vikles næsten to gange omkring en histonoktamer. De otte histoner og 147 basepar DNA danner en enhed, der kaldes en nukleosomkerne
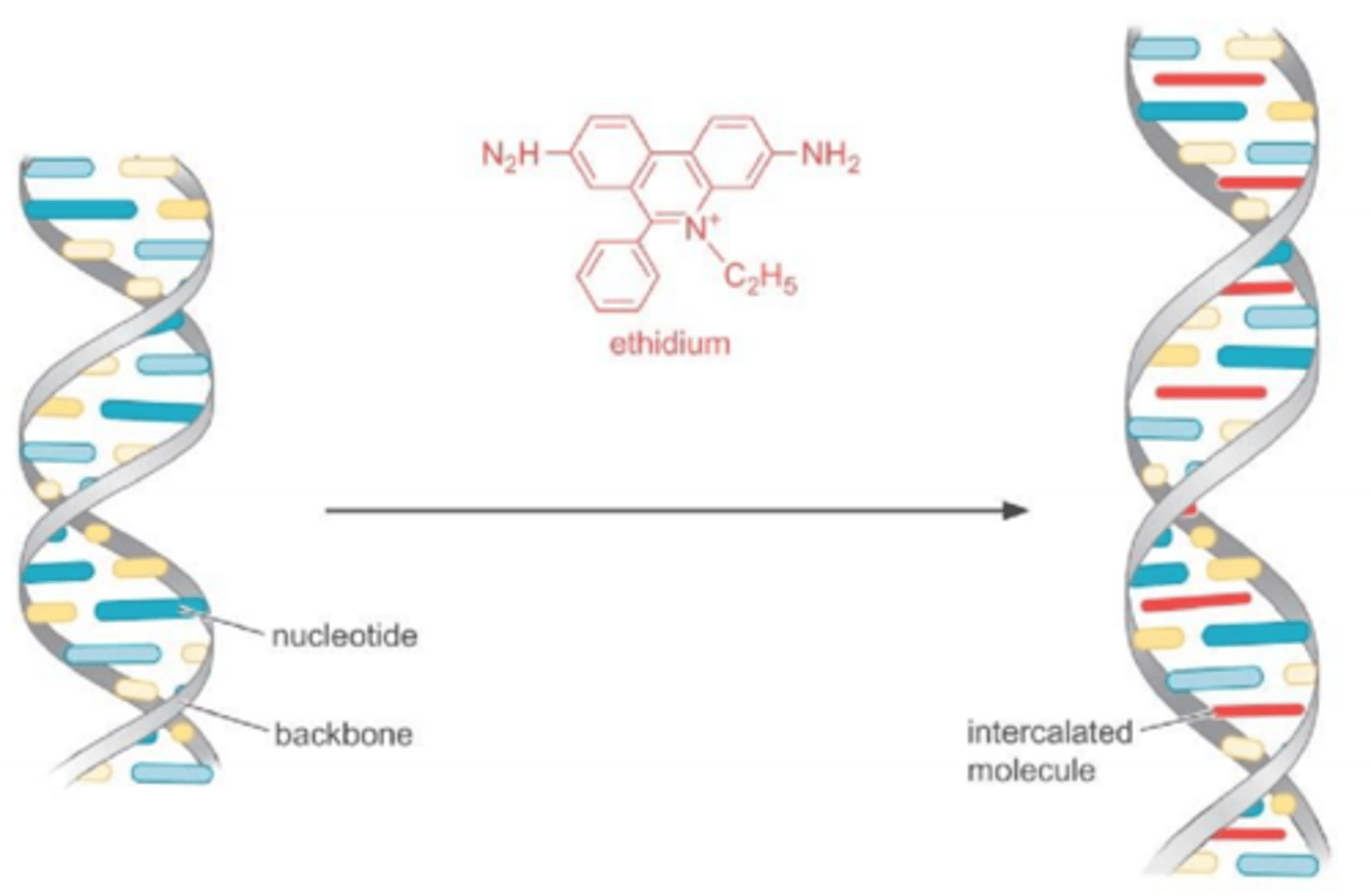
Ethidium bromide intercalation
1) Øger basepar afstand
2) Introducerer positive supercoils
3) Forstyrrer sugar-phosphate backbone
En DNA supercoil er en slags tredimensionel struktur som dannes automatisk når en DNA-streng bliver snoet.
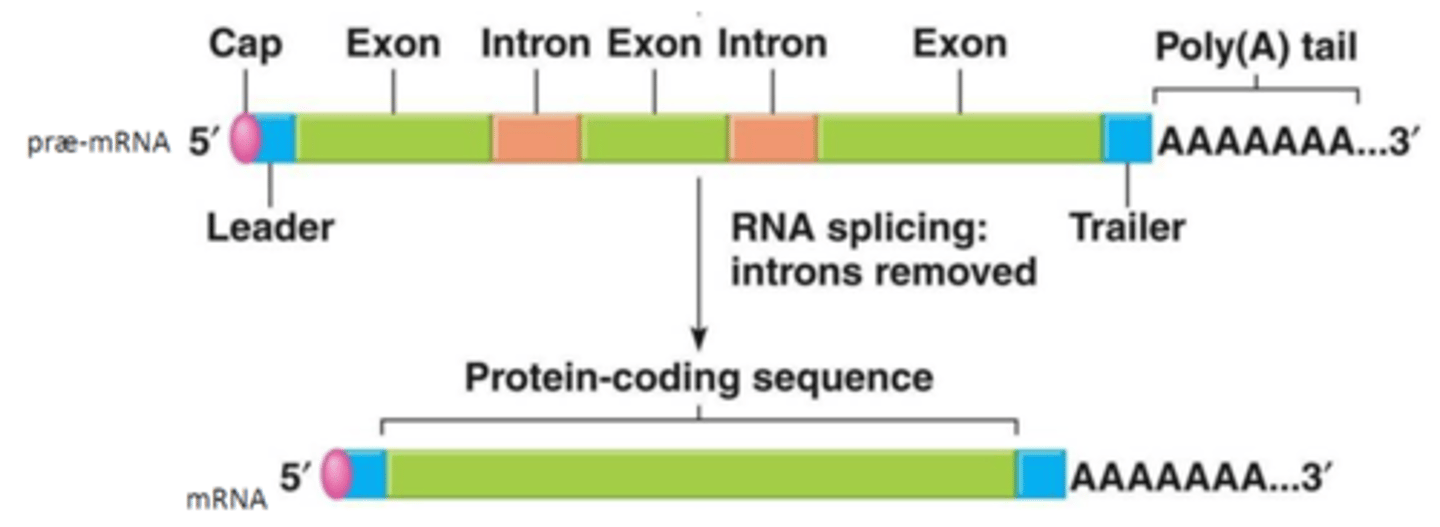
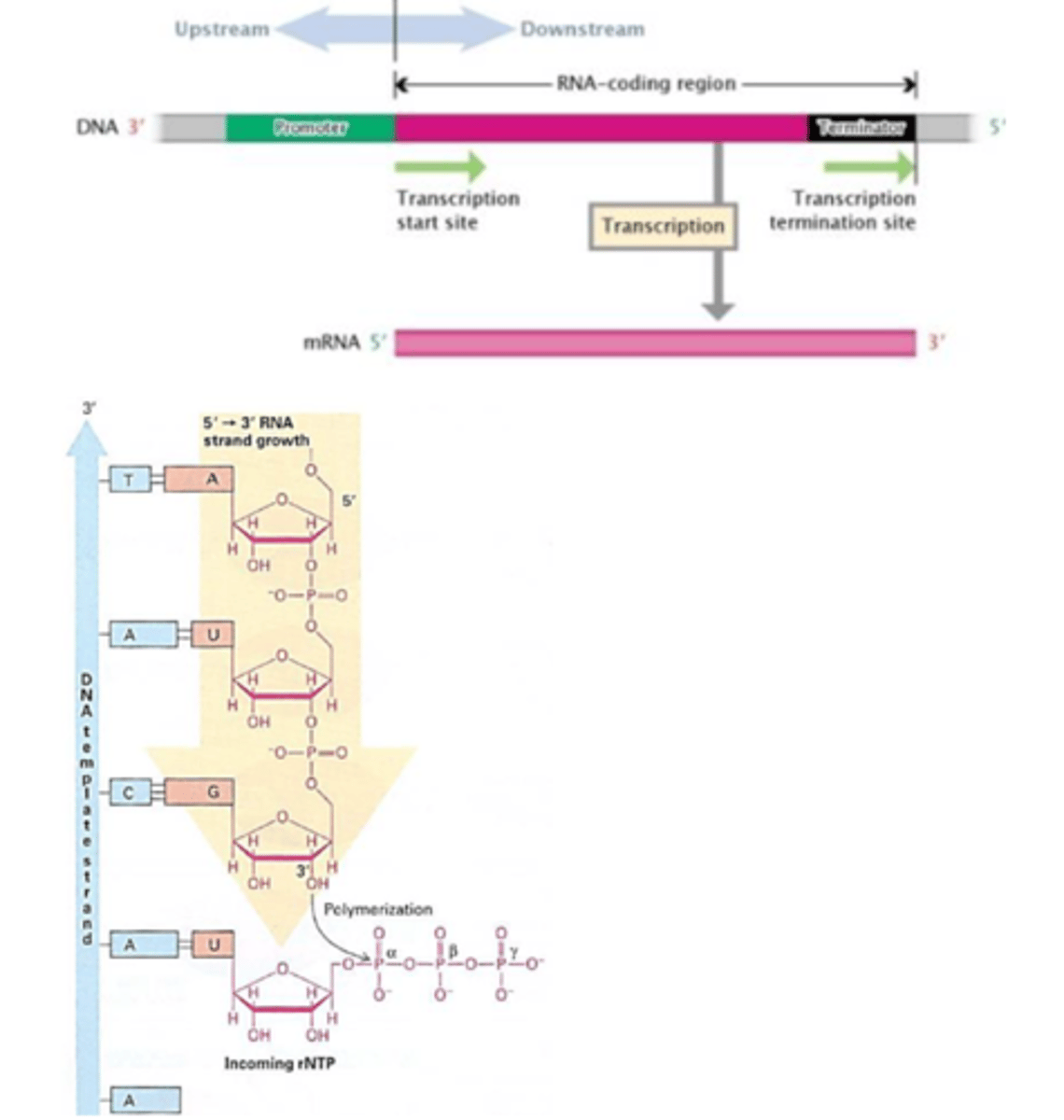
Transkriptionsprocessen
foregår ved "omskrivning" af en DNA-sekvens (et gen) til en RNA-sekvens ved komplementær baseparring
er den proces, hvori der udvælges og overføres information fra DNA-molekylet til et RNA-molekyle.
intron
den ikke proteinkodende del
exon
den proteinkodende del
Transkriptionsprocessens 3 trin
1) Initiering
RNA-polymerasekomplekset binder til promoteren, og DNA-helixen åbnes
Transkriptionsprocessens 3 trin
2) Elongering
syntese af en RNA-streng ved komplementær baseparring
Transkriptionsprocessens 3 trin
3) Terminering
RNA polymerase II og den nysyntetiserede mRNA dissocierer fra DNA templaten
DNA template (anti-sense)
er den DNA-streng, som er komplementær til mRNA strengen
DNA non-template (sense)
er den DNA-streng, som har samme sekvens, som den endelige mRNA-streng
5’capping
Processen foregår co-transkriptionelt (dvs. mens RNA syntetiseres) og dannes når RNA-strengen er 20-40 nukleotider lang.
Der påsættes et methyleret guaninnukleotid på det 5’-terminale nukleotid af mRNA-strengen.
Yder beskyttelse mod RNA-nedbrydende enzymer (5’exonukleaser) og er vigtig for transport ud af nucleoplasmaet
Polyadenylering
Påsætning af kæde af 100-250 adenin-nukleotider (poly(A)-hale) i 3'-enden af mRNA. Udføres af en polyadenyleringspolymerase og foregår posttranskriptionelt
Vigtig for transport fra nucleus til cytoplasma, beskytter mod nedbrydning af deltager i translation
RNA-splejsning
Fjernelse af ikke-kodende introns + ligering af exons
Processen katalyseres af et enzym kaldet spliceosome
Translation
oversættelse af en mRNA-sekvens til en aminosyresekvens
Den genetiske kode
består af triplets, det vil sige sekvenser på tre nukleotider i mRNA-strengen. En sådan triplet kaldes en codon, og hver codon specificerer én bestemt aminosyre eller angiver translationsstop, idet tre codons er stop-codons, der ikke koder for aminosyrer, men i stedet signalerer termineringen (afslutningen) af translationen. AUG-startcodon definerer den korrekte læseramme for det ønskede protein. Sekvensen mellem et startcodon og et stopcodon kaldes en åben læseramme, da denne sekvens er ”åben” for translation.
Telomerer
I enderne af kromosomerne optræder en særlig struktur kaldet telomeren (græsk: telos = ende). Telomererne består af 500-5.000 gentagelser af DNA-sekvensen 5’TTAGGG3’
Telomerkomplekset beskytter kromosomenden mod nedbrydning af nukleaser. Nukleaser er en gruppe af enzymer, som katalyserer nedbrydningen af nukleinsyrer (DNA og RNA)
For hver replikation af kromosomet sker der en gradvis forkortelse af kromosomenderne. Da de mange repeterede basesekvenser i telomererne ikke indeholder gener, fungerer de telomeriske DNA-sekvenser som en buffer mod tab af vigtig kodende sekvenser. Længden af telomeren har derfor betydning for det antal af replikationer og derved det antal af delinger, en celle kan gennemløbe
Replikationsstartsteder
er specielle DNA-sekvenser, hvor DNA-replikation initieres. Eukaryote kromosomer indeholder flere hundrede replikationsstartsteder, der ligger jævnt fordelt langs kromosomet. Hvert replikationsstartsted består af en sekvens, der er rig på A-T-basepar, og som genkendes og bindes af replikationskomplekset, der er ansvarlig for initiering af replikationsprocessen. A-T-basepar har kun 2 hydrogenbindinger og er derfor lettere at splitte.
Kinetokoret
Efter replikation af DNA-molekylerne i S-fasen af cellecyklus holdes de replikerede kromosomer sammen i centromer-regionen, og til regionen bindes en række proteiner, der tilsammen danner kinetokoret. Under celledeling binder tentrådene til kinetokoret, og centromeret er derfor nødvendigt for separation af replikerede kromosomer i henholdsvis mitose og meiose
Intergenisk DNA
Størstedelen af det humane genom består af intergenisk DNA. Dette DNA blev tidligere kaldt ”junk DNA” eller ”nonsens-DNA”, da det synes overflødigt for organismens livsfunktioner og overlevelse
Fossile rester, som i evolutionens løb er blevet efterladt i genomet og ikke bortselekteret, fungerer som mutationsbuffer, hvor mutationer eller overkrydsningsfejl i meiosen kan ramme og ophobes uden at have betydning for organismens overlevelse.
En stor del af det intergeniske DNA bliver dog transkriberet til RNA (og derfor indeholder gener). Disse RNA-molekyler oversættes ikke til protein, men spiller en rolle for regulering af genekspression.
Andet af det intergeniske DNA spiller strukturelle roller, eksempelvis opbygning af centromerer og telomeriske regioner i kromosomerne.
Tandem-repeats (satellit DNA)
simple repeterede sekvenser.
Det samlede område af repeteret DNA kaldes satellit-DNA. Fragmenter af repeteret DNA har en anderledes densitet end andet fragmenteret genomisk DNA og vil bevæge sig i særlige bånd, som satellitter, ved densitetscentrifugering.
Satellit-DNA findes i udbredt grad i telomer- og centromerregionerne. De mange tandem-repeats opstår som følge af fejl i DNA-replikationsprocessen eller ved skæv rekombination under meiose og mitose.
Ribosomalt RNA (rRNA)
Ribosomalt RNA indgår som strukturelle og katalytiske komponenter i ribosomerne. 2/3 af ribosomets masse består af RNA, og da en typisk eukaryot celle indeholder flere millioner ribosomer, består omkring 80 % (83 %) af cellens totale mængde RNA af rRNA-molekyler.
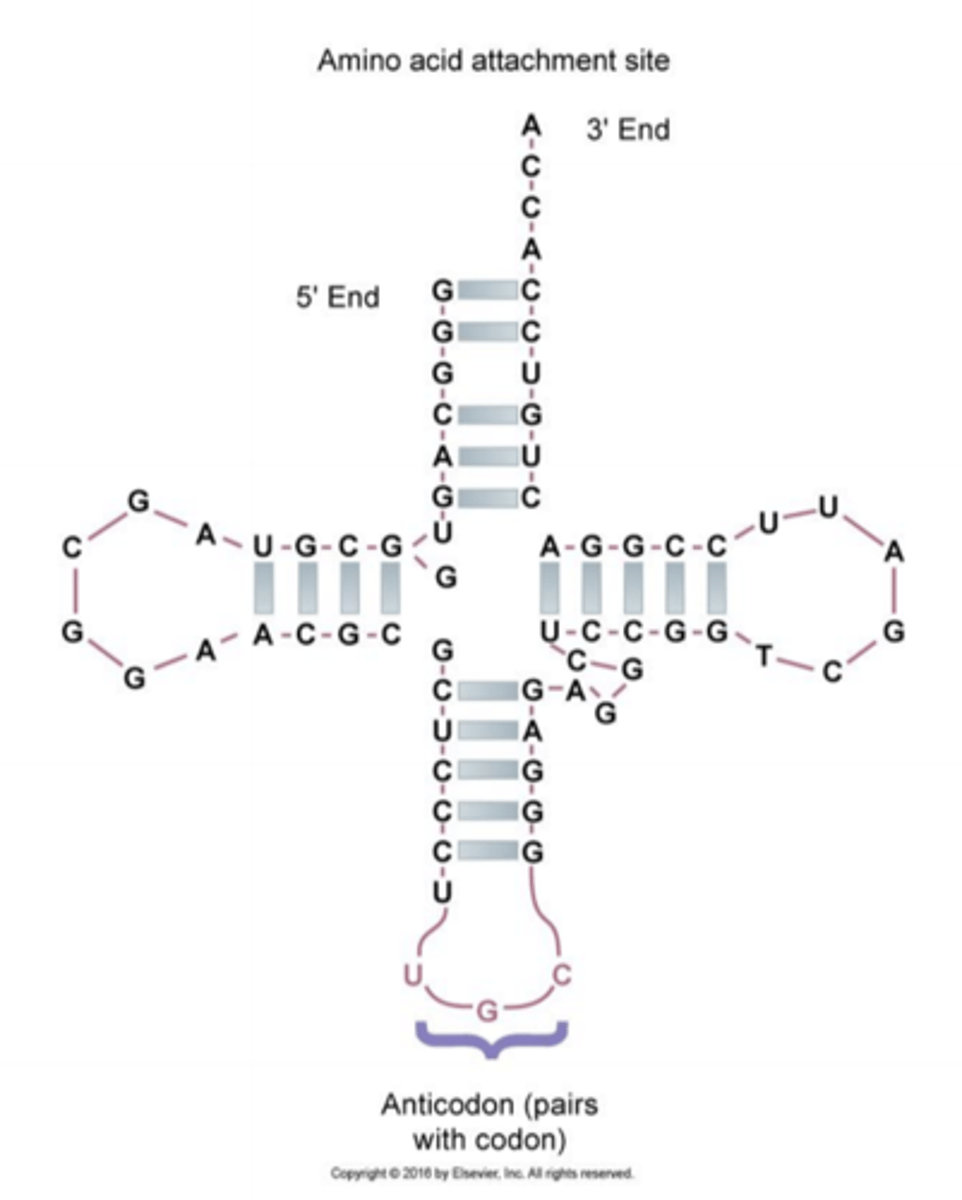
Transfer RNA (tRNA)
Transfer-RNA binder og bærer aminosyrerne til ribosomerne under translationsprocessen (15 %)
Et tRNA-molekyle indeholder to bindingssteder: ét for en bestemt sekvens (den genetiske kode) i mRNA-strengen og ét for den bestemte aminosyre, der skal indsættes i polypeptidkæden ifølge den genetiske sekvens. tRNA-molekylerne kan således læse koden i mRNA og bringe aminosyren til den rette plads i peptidsekvensen
tRNA-molekylerne bærer aminosyrerne til ribosomet, hvor også mRNA-strengen er bundet. tRNA binder sekvensspecifikt til mRNA-strengen og indsætter aminosyrerne, som det dikteres af mRNA-strengen. Ribosomet katalyserer altså sammenkoblingen af aminosyrerne til en polypeptidkæde(aminosyrer som er kovalent sammenbundet via peptidbindinger - danner en kæde)
Messenger RNA (mRNA)
Messenger-RNA overfører information fra protein-kodende gener i DNA-molekylet til ribosomerne (2%)
Replikation
DNA kopiering
Replikationsprocessen består af tre hovedtrin: Initiering, hvor DNA-helixen åbnes, så baserne eksponeres til baseparring med nye nukleotider. Dernæst sker elongering, hvor DNA-polymeraser katalyserer syntese af en ny DNA-streng. Replikation terminerer, når to replikationsgafler mødes, eller enden af kromosomet nås
Enzymer med helicase-aktivitet
katalyserer brydning af hydrogenbindinger mellem baseparrene, så DNA-helix-strukturen åbnes. Herved dannes et replikations-"øje"
Primer
DNA-polymeraser kan ikke påbegynde dannelse af en nukleotid-streng fra bunden. De skal have en fri 3’-hydroxygruppe at forlænge ud fra. I modsætning hertil kan RNA-polymeraser starte de novo. Derfor initieres DNA-syntese med dannelsen af en RNA-sekvens, en såkaldt primer, på omkring 10 ribonukleotider. Efter syntese af den initiale primer forlænges nukleotidstrengen med deoxyribonukleotider af DNA-polyemrasen
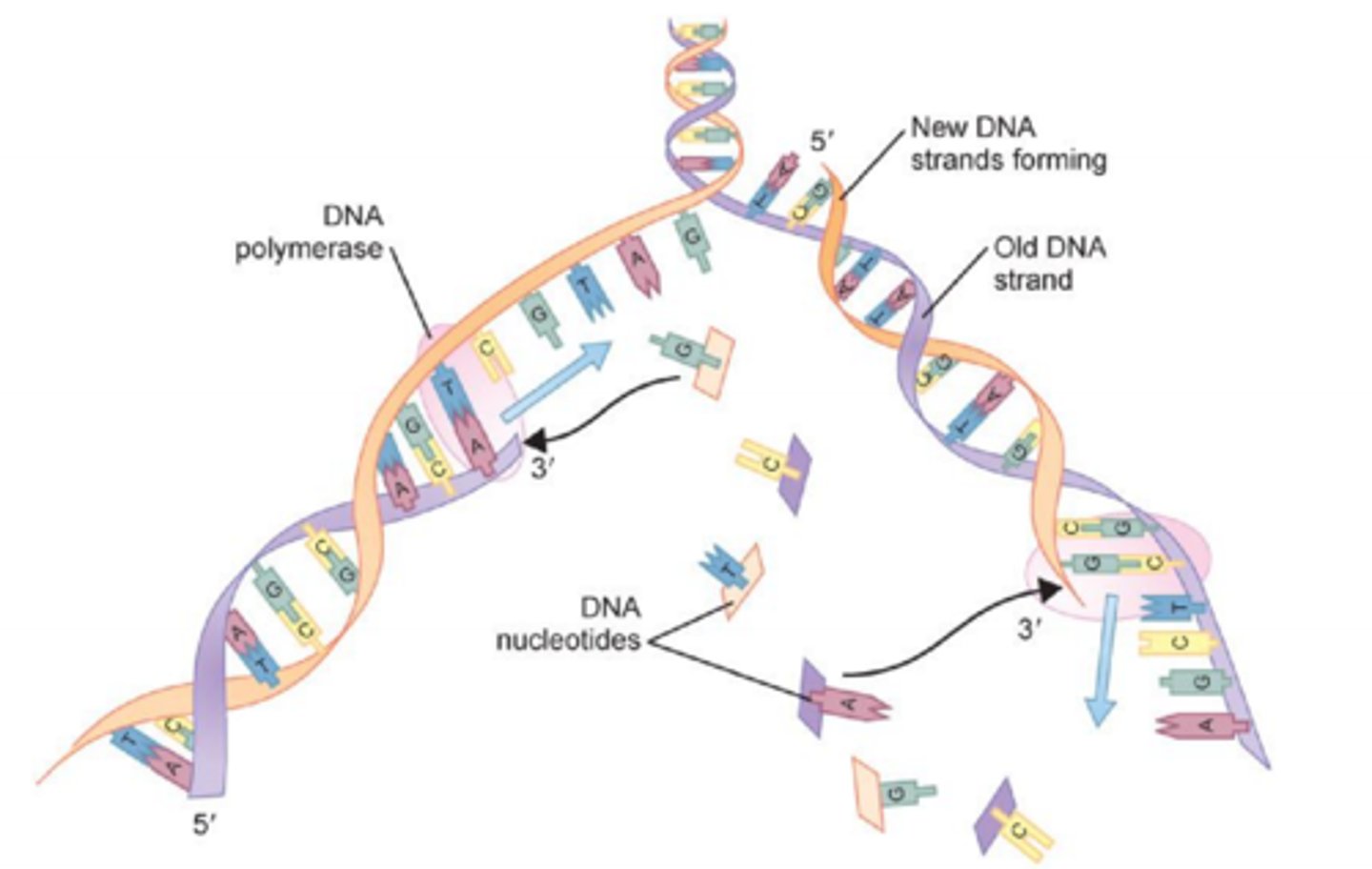
leading-strand-syntese
DNA-polymeraser kan kun aflæse 3’ mod 5’, mens de syntetiserer den nye DNA-streng 5’ mod 3’ ("DNA polymerase moves along the old strand in the 3'–5' direction, creating a new strand having a 5'–3' direction")
Replikation af skabelonstrengen, der forløber 3’ mod 5’ væk fra replikationsstartstedet er derfor ikke noget problem. Efter anlæggelse af en initial primer kan DNA-polymerasen syntetisere en ny streng ud fra primeren, i takt med at replikationsgaflen åbner sig. Syntese af denne streng kaldes leading-strand-syntese
lagging-strand-syntese
Replikation af den anden skabelonstreng, den der forløber 5’ mod 3’ væk fra replikationsstartstedet, er problematisk, da DNA-polymeraser jo kun kan aflæse 3’ mod 5’
Den nye streng syntetiseres derfor diskontinuerligt i små isolerede fragmenter, der bagefter ligeres sammen. Syntese af denne streng kaldes lagging-strand-syntese, fordi færdiggørelse af denne streng foregår med lidt forsinkelse i forhold til leading-strand
Replikation af telomererne (terminering)
I 5'-enderne af hver nysyntetiserede streng befinder sig en RNA-primer.
Denne primer vil typisk blive fjernet efter endt replikation, og når den fjernes, vil sekvensen ikke kunne erstattes med DNA, idet DNA-polymerasen ikke har nogen nukleotidsekvens at forlænge ud fra.
Herved forkortes kromosomerne gradvist for hver replikation, det vil sige for hver cellegeneration.
Det er sjældent, at primerne anlægges lige akkurat i enden af kromosomet, og for hver celledeling mistes typisk omkring 50-100 nukleotider, alt efter hvor langt fra enden af kromosomet primeren blev anlagt.
Opretholdelse af en telomerlængde på en vis størrelse er essentiel for kromosomets stabilitet.
Når telomersekvensen bliver for kort, vil cellen gå ind i en aldringsfase, hvor den stopper med at dele sig.
Genomets organisering
Prokaryoter
forholdsvis simpel struktur, generne ligger i en række, ofte i operoner, meget lidt eller intet ikke-kodende DNA, altid mellem generne
Genomets organisering
Eukaryoter
komplicerede arrangementer, gener reguleret hver for sig (ingen operoner), rigtig meget ikke-kodende DNA, som ligger både inde i generne og udenfor kromosomerne
Single-copy DNA
Are seen only once (or possibly a few times) in the genome
Single copy DNA accounts for about half of the genome (45 %) and includes the protein-coding genes
Den proteinkodende del (exons) udgør kun 2 %, resten er introns
Dispersed repetitive DNA
Half of the genome consists of repetitive DNA, sequences that are repeated over and over again in the genome, often thousands of times
There are two major classes of repetitive DNA: Dispersed repetitive DNA and satellite DNA
Dispersed repeats, as the name implies, tend to be scattered singly throughout the genome; they do not occur in tandem
Dispersed repetitive DNA makes up about 45 % of the genome, and these repeats fall into several major categories: The two most common categories are short interspersed elements (SINEs) and long interspersed elements (LINEs).
SINEs (short interspersed nuclear elements)
repetitive DNA sequences, approximately 500 bp in length on average, interspersed throughout the genome
One of the most important types of SINEs in humans is the 300-bp Alu element, so named because each repeat contains a DNA sequence that can be cut by the Alu restriction enzyme. About 1 million Alu repeats are scattered throughout the genome; they thus constitute approximately 10 % of all human DNA
LINEs (long interspersed nuclear elements)
repetitive DNA sequences, approximately 6500 bp in length on average, interspersed throughout the genome
Sattelit DNA
Satellite repeats are clustered together in certain chromosome locations, where they occur as tandem repeats (i.e., the beginning of one repeat occurs immediately adjacent to the end of another)
Satellite DNA accounts for approximately 8 % to 10 % of the genome
Alpha-satellite DNA
occurs as tandem repeats of a 171-bp sequence that extend to several million base pairs or longer. This type of satellite DNA is found near the centromeres of chromosomes
a 171-bp sequense is repeated near the centromere of chromosomes
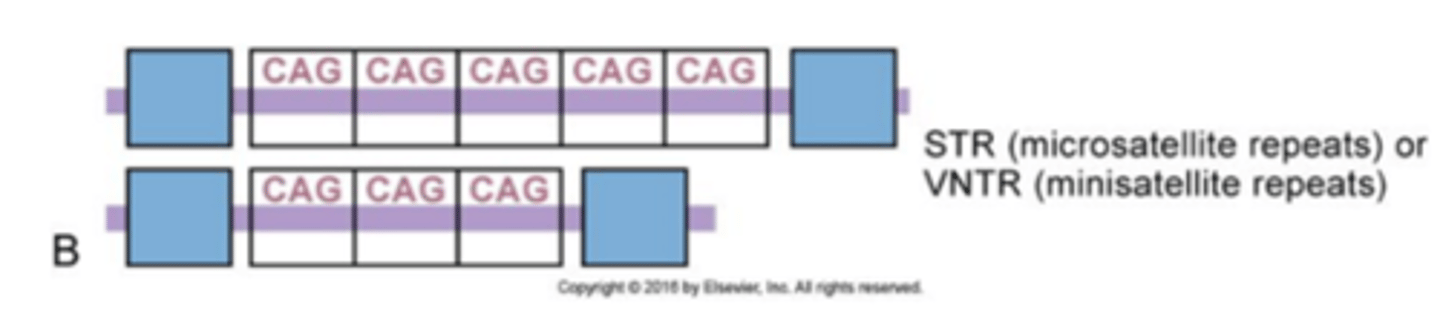
Minisatellites
are blocks of tandem repeats (each 11 bp long or greater) whose total length is much smaller, usually a few thousand base pairs
a 10-60 bp long nucleotid sequence is repeated
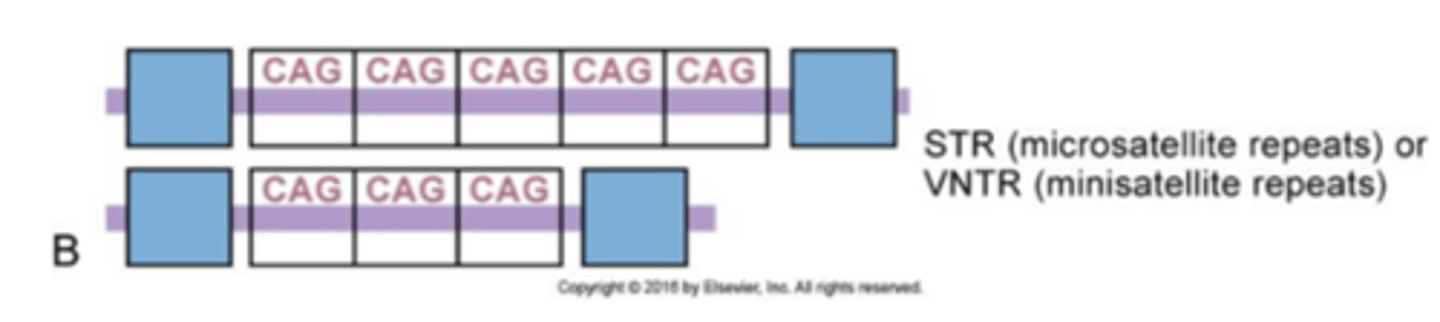
Microsatellites
are smaller still; the repeat units are 1 to 10 bp long, and the total length of the array is usually less than a few hundred base pairs
Pseudogener
DNA-sekvenser med gen-lignende sekvens, men ikke funktionelle. Indeholder defekter/mangler, som gør, at protein ikke kan produceres: Mangler et start-codon, mangler eller har for mange stop-codons eller ændringer i de regulatoriske elementer
Mutationer i denne sekvens har ikke nogen betydning (ingen konsekvenser, fordi genet ikke er funktionelt!). Mutationer i pseudogener er neutrale og derfor fri for den evolutionære selektion. Pseudogener har derfor en evolutionær betydning, som prækursor af nye gener (kan give ophav til nye gener)
VNTR (variable number of tandem repeats)
tandem (next to each other) repeats of a short DNA sequence (9 to 80 base pairs) with varying numbers of repeats among individuals
Mendels 1. og 2. lov
1. De to alleler segregerer uafhængigt og ligeligt ved gametdannelsen (Mendels 1. lov), dvs., at ved gametdannelsen udspalter heterozygoten ”Aa” gameter med allelerne A og a i forholdet 1:1
2. Alleler i forskellige loci segregerer uafhængigt af hinanden ved gametdannelsen (Mendels 2. lov)
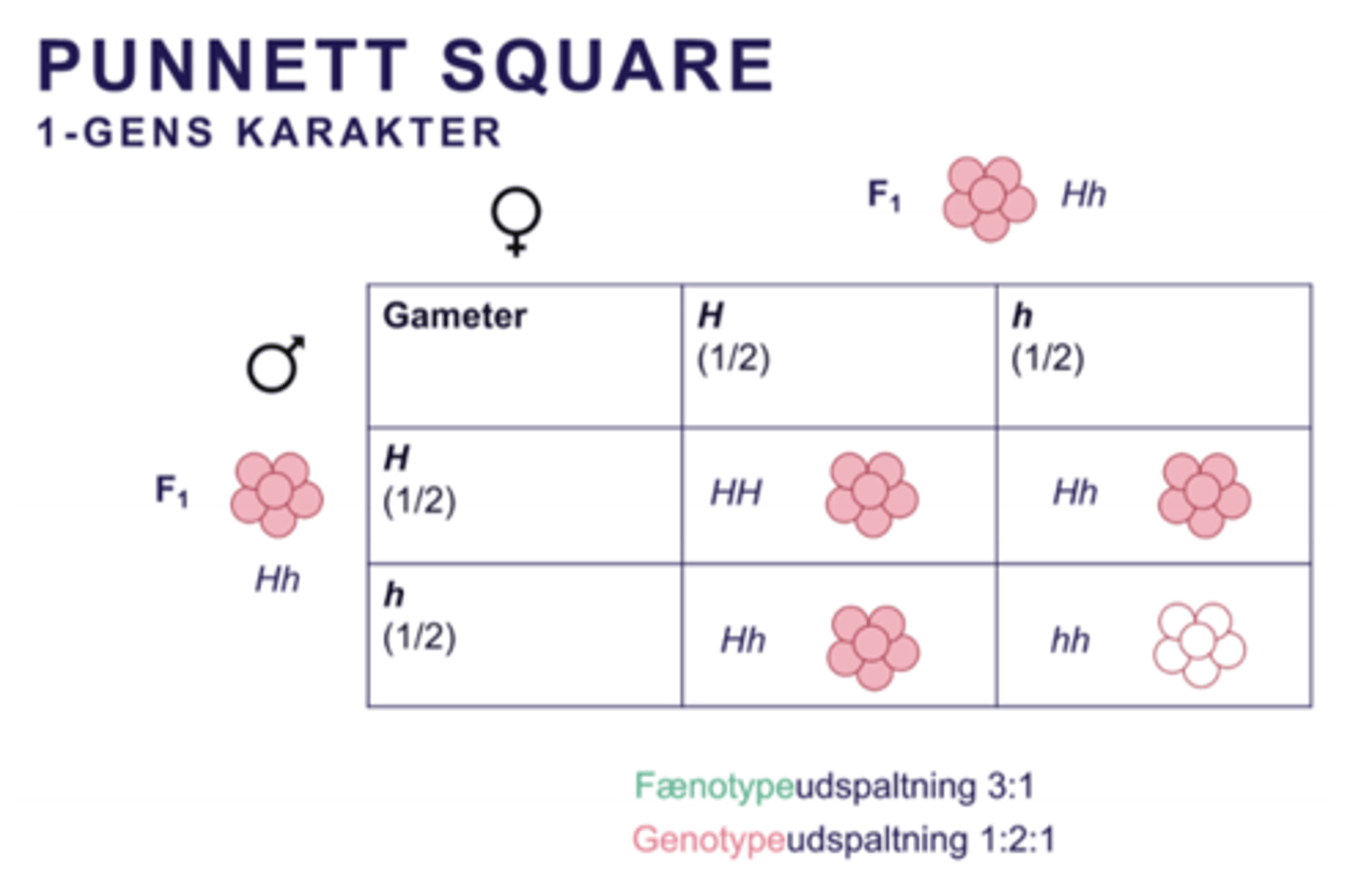
Monohybrid krydsning
1-gens kryds, nedarvning af 1-gens karakter
Forventede fænotypeudfald 3:1
Forventede genotypeudfald 1:2:1
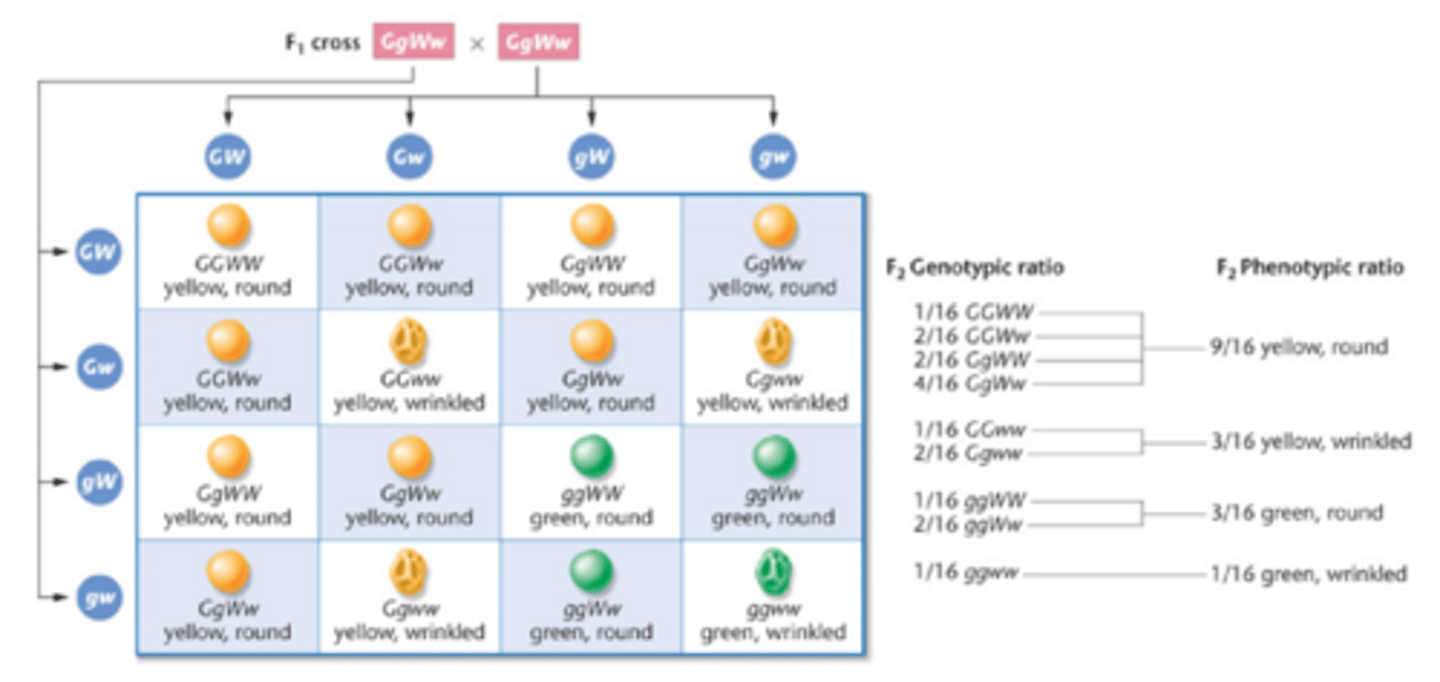
Dihybrid krydsning
2-gens kryds, samtidig nedarvning af 2 karakterer
2 gener, hver med 2 alleler og 2 fænotyper
Forventede fænotypeudfald 9:3:3:1
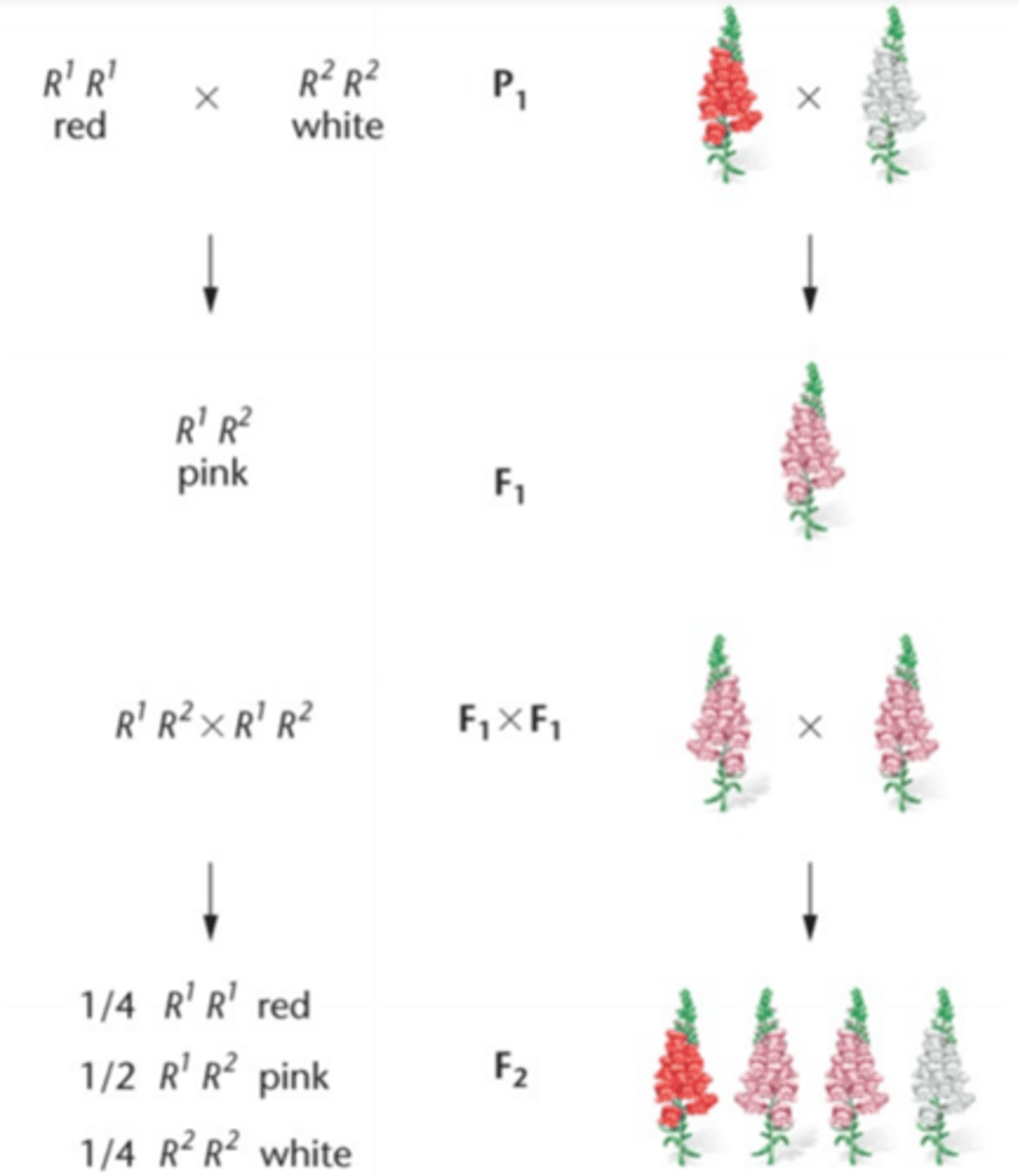
Monohybrid krydsning
Partiel dominans
interaktion mellem alleler (indenfor det samme locus)
Heterozygoter har intermediær fænotype, fx krydser man en rød blomst med en hvid blomst får man en tredje fænotype = lyserød; den intermediære fænotype
Monohybrid krydsning
Co-dominans
interaktion mellem alleler
Heterozygoter udviser begge fænotyper (indenfor det samme locus)
Dette kender vi fra vores blodgruppe-system MN (to alleler; M og N)
Eksempel: har man to forældre, som begge er heterozygote for MN, så er det forventede udspaltningsforhold (fænotypeudfald) IKKE 3:1, som man normalt vil forvente ved 1-gens udspaltning, men det er i stedet forholdet 1:2:1

Monohybrid krydsning
Multiple alleler
Et gen, mange alleler
Ofte er der ikke kun 2 alleler, der er mange fx øjenfarve hos bananfluen
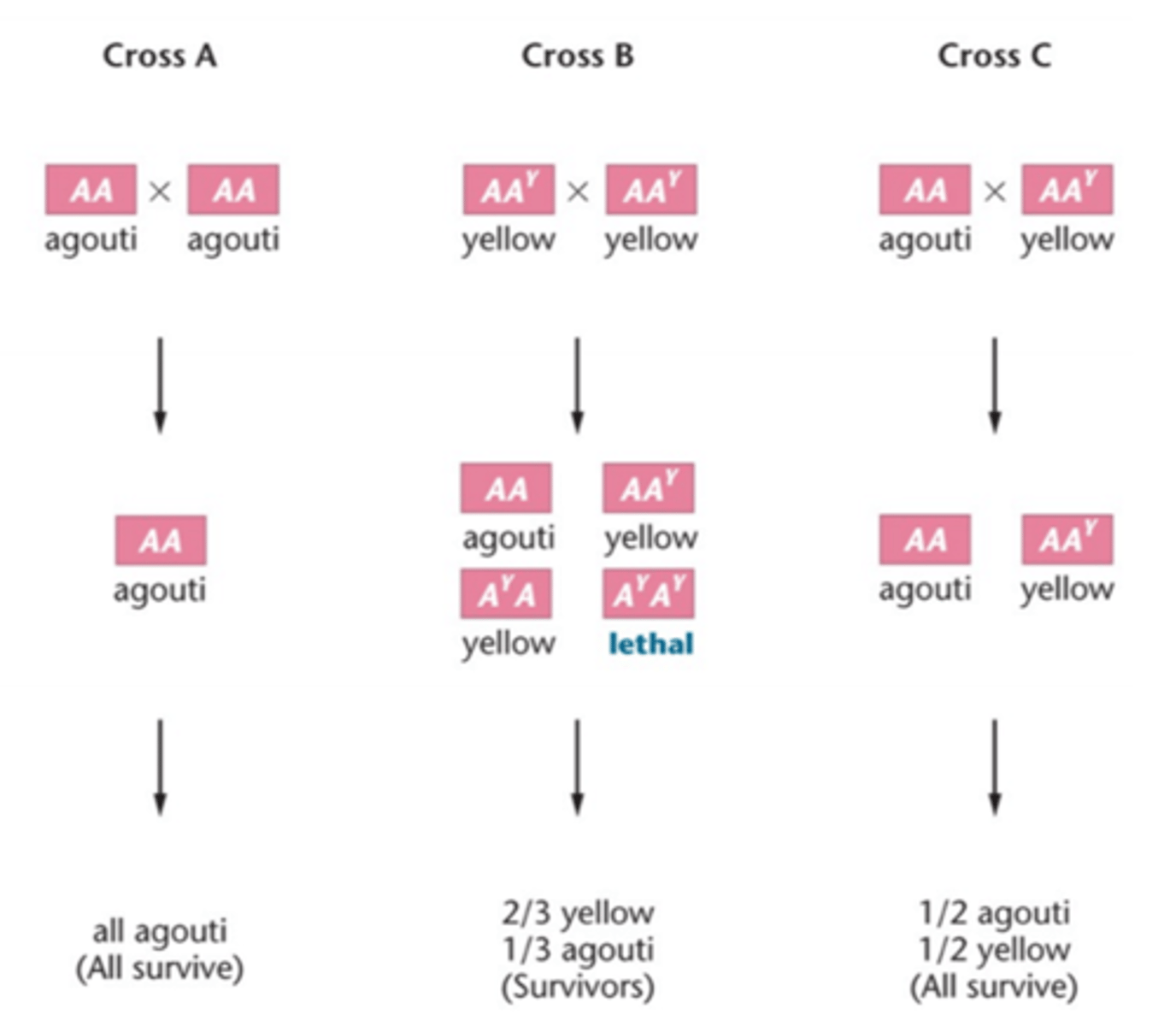
Lethale alleler
Dødelige
Findes både dominante og recessive
Epistasi
Gen-interaktioner
Epistasi er når effekten af et gen afhænger af tilstedeværelsen af ét eller flere ”modifier genes”
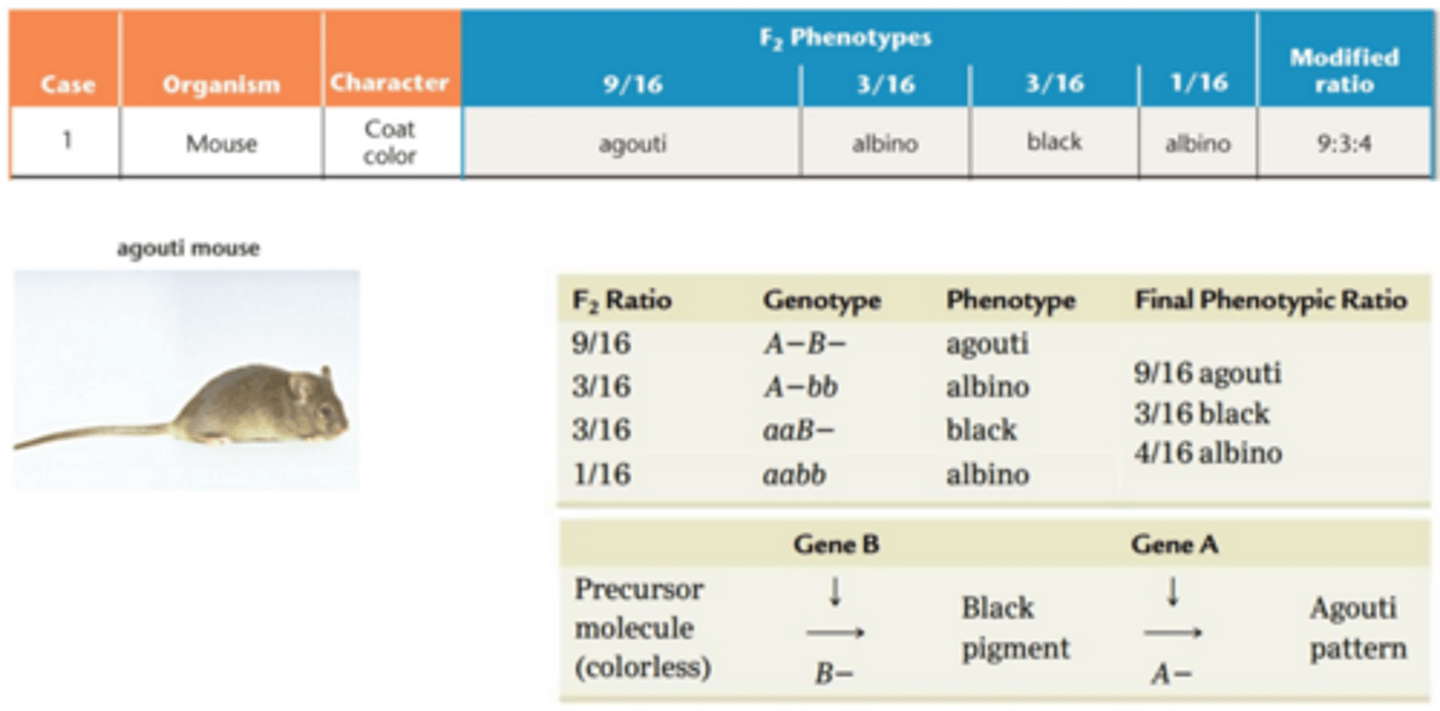
Eks: modificeret dihybrid udspaltning, pelsfarven på mus er en kombination af fænotypeklasser
Fænotypeudfald 9:3:4 afviger fra det forventede udfald 9:3:3:1 ved dihybrid/to-gen krydsning
Den neutrale population
1) Tilfældig selektion
2) Ingen selektion
3) Ingen drift (uendelig stor populationsstørrelse)
4) Ingen migration
5) Ingen mutation
Mutation
fejl under DNA-replikation og transkription, der introducerer nye alleler i populationen
Naturlig selektion
"survival of the fittest" hvilket vil sige, at genotypehyppighederne ikke er konstante, men vil ændre sig over tid
Eks: Mennesket i Norden har udviklet den lysere hudfarve, da det ifølge teorierne har haft den fordel, at mere UV-stråling kunne slippe ind til huden – og dermed sørge for mere produktion af D-vitamin
Genetisk drift
Differential transmission af alleler til næste generation som konsekvens af tilfældigheder (random sampling), hvilket har størst betydning i små populationer
1. Tilfældig udvælgelse af alleler i en population
2. En evolutionær kraft som er stærk i små populationer
Gen-flow
Spredning af alleler fra en population til en anden via migration, hvilket gør dem mere genetisk ens
Eksempel på Gen-flow - Seglcelleanæmi: En ændring i genotypen medfører en ændring i fænotypen. Allelfrekvensen er meget hyppig i centralafrika i forhold til i resten af verden, fordi man er beskyttet mod malaria, hvis man har seglcelleanæmi. Hvis cellerne har segl-form, kan de ikke inficeres med malaria-parasitten
Basepar substitutioner
et nukleotid ændres til/udskiftes med et andet nukleotid
et basepar erstattes af et andet, hvilket kan resultere i ændring af aminosyresekvensen
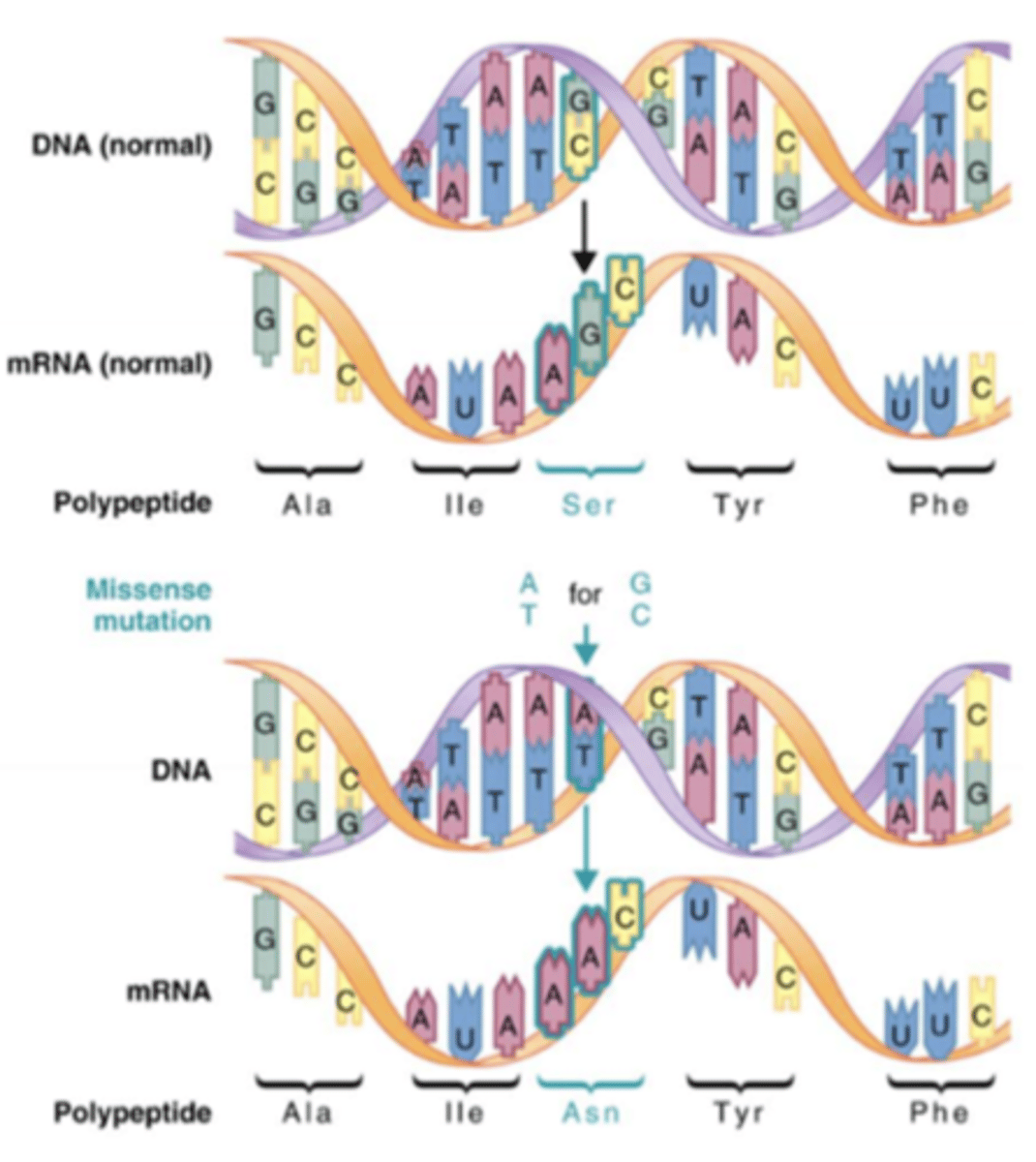
Missense mutation
Basepar-substitution,
der kodes for en anden/ny aminosyre
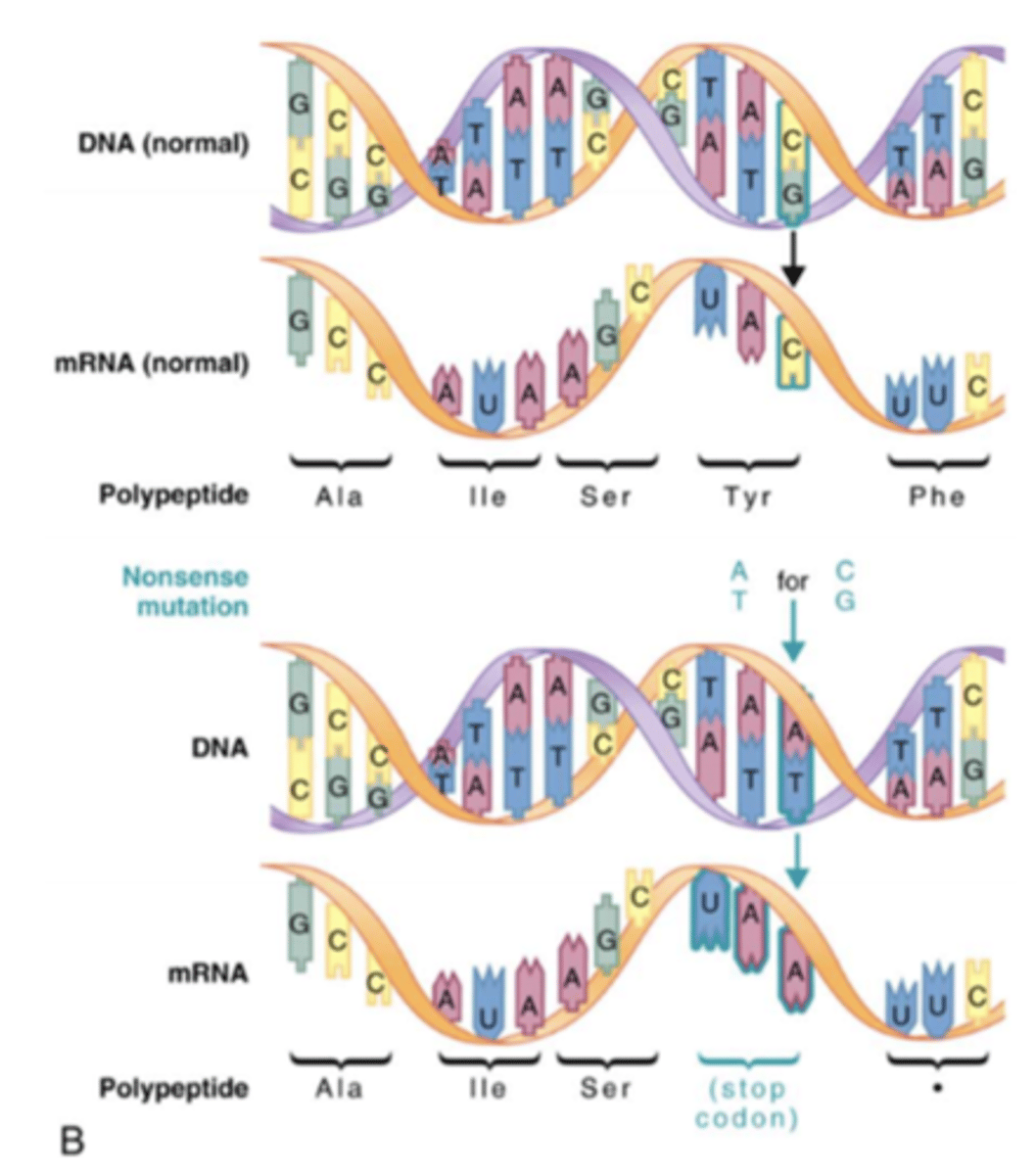
Nonsense mutation
der introduceres et af de tre stop-codons (UAA, UAG eller UGA)
På billedet: CG erstattes af AT og den nye triplet i DNA-strengen koder for et af de tre stop codons (UAA, UAG eller UGA) i mRNA-strengen. Stop codons afslutter translationen (dvs. oversættelsen) af mRNA til aminosyrer, og resulterer derfor i tidlig terminering (afslutning) af polypeptidkæden eller i ødelæggelse af selve transkriptet (mRNA-strengen) gennem en proces kendt som ”nonsense-mediated mRNA decay”.
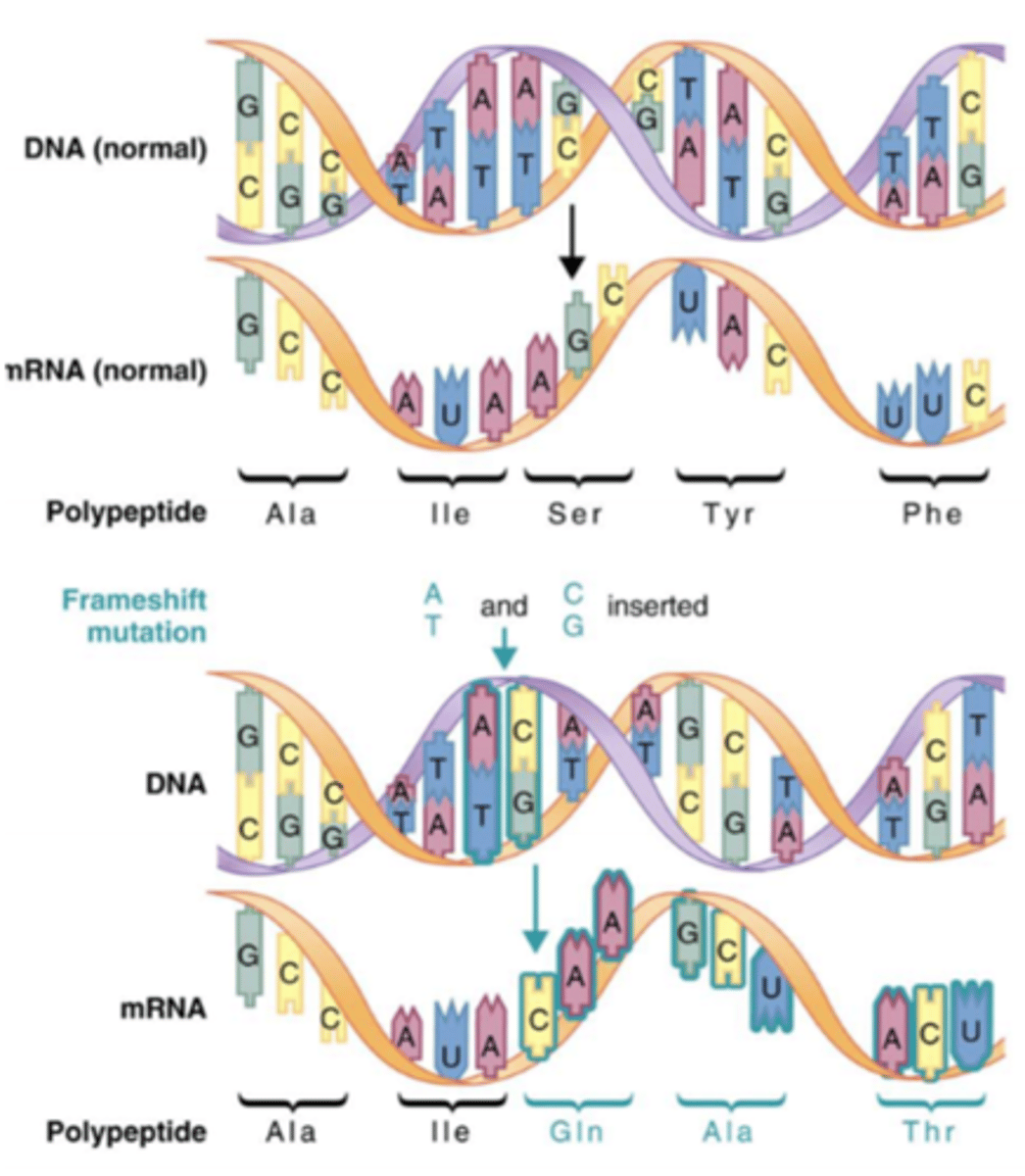
Deletioner
der fjernes baser
Insertioner
der indsættes baser
frame shift mutation (deletioner eller insertioner)
I) Frameshift mutationer opstår når indel (insertioner eller deletioner) øger eller reducerer antallet af baser med et tal, der ikke er et multiplum af 3, dvs. fjernes eller indsættes tre baser (en triplet) vil læserammen ikke blive forskudt, men en aminosyre vil enten ”forsvinde” eller tilføjes
II) På figuren nedenfor ses en insertion. Der er tilføjet to basepar (AT og CG) mellem baseparrene TA og AT i den oprindelige kodende DNA-streng. Dette medfører, at læserammen ”forskydes”, og der kodes derfor for andre/nye aminosyre, så proteinets aminosyresekvens ændres (se figuren nedenfor). Dette ødelægger proteinets funktion, fordi alt efter insertionen er ”ulæseligt”
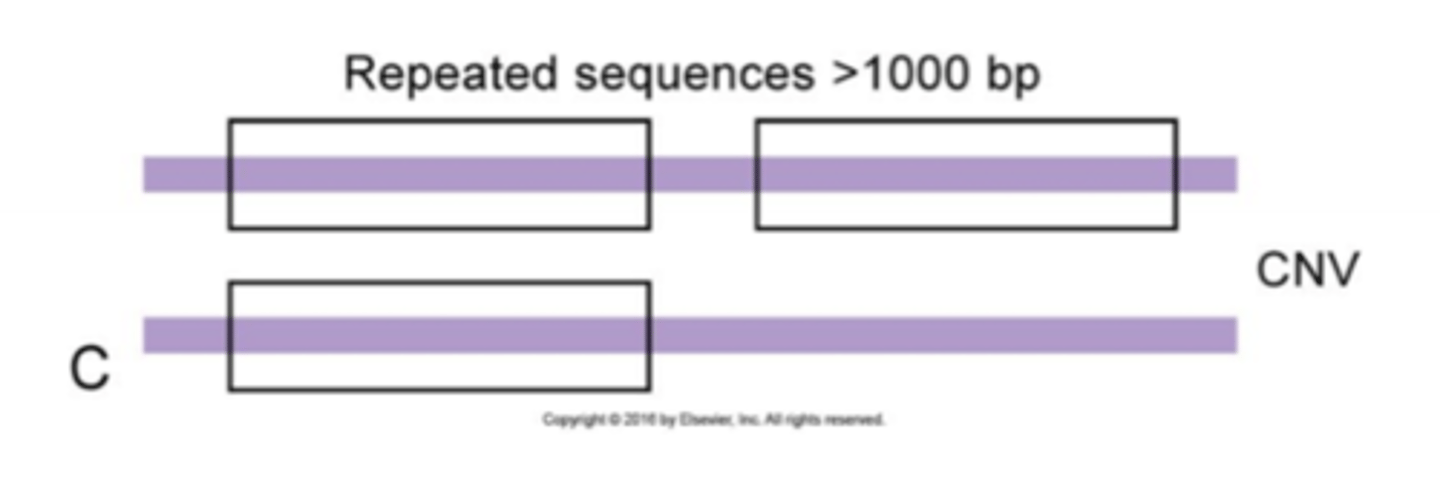
Copy Number Variation (CNV)
Difference among individual organisms in the number of copies of any large DNA sequence (larger than 1000 bp).
1. Større områder af vores arvemasse (> 1000 bp), som vi kan have flere eller færre af (forskelligt blandt individer)
2. Områderne kan være fra 50 basepar til kromosomstørrelseslængder
Single Nucleotide Polymorphisms (SNPs)
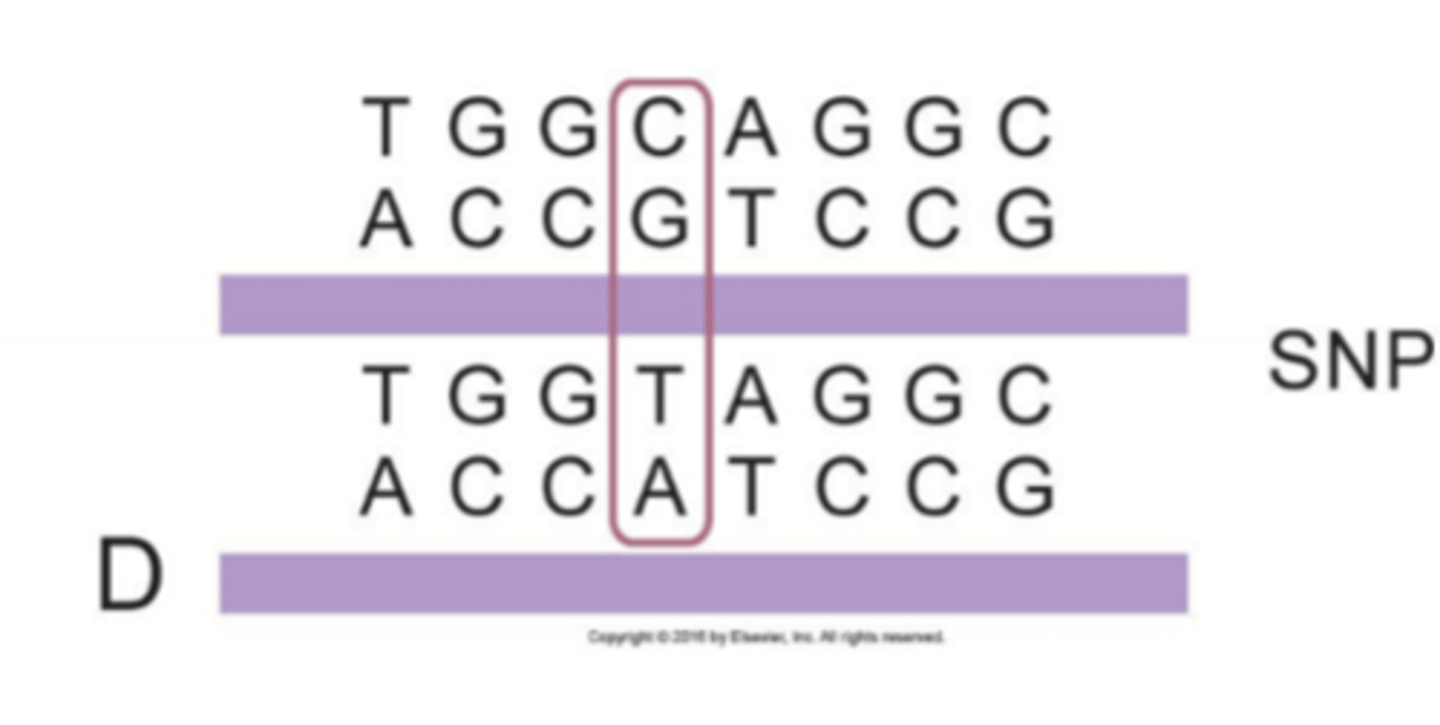
Inherited variations in a single nucleotide base in DNA that differ between individuals.
1. Når vi sammenligner et enkelt sted i vores arvemasse på tværs af individer, så er der polymorfi, hvilket vil sige, at der er forskellige nukleotider her på dette sted
2. Vi har ca. 90 millioner SNPs i vores arvemasse blandt de 3 milliarder basepar vores genom er
3. Denne type varianter finder vi typisk ved SNP array, Whole Exome Sequencing (WES) eller hel genom sekventering (WGS)
Synonyme mutationer
giver/medfører IKKE ændringer i aminosyresekvensen. Den "nye" triplet med et udskiftet nukleotid koder for den samme aminosyre, som den "gamle" triplet, og derfor vil det ikke give ændringer i proteinet
