CH2 Acides nucléiques et flux de l’information génétique
1/22
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No analytics yet
Send a link to your students to track their progress
23 Terms
Ou se trouve l’information génétique dans la cellule?
Stocké dans l’ADN, principalement dans le noyau
une petite quantité se trouve aussi dans les mitochondries.
Quel est le flux de l’information génétique (Dogme central)?
ADN to ARN to Protéines
Transcription: ADN to ARN
Traduction: ARN to Protéines
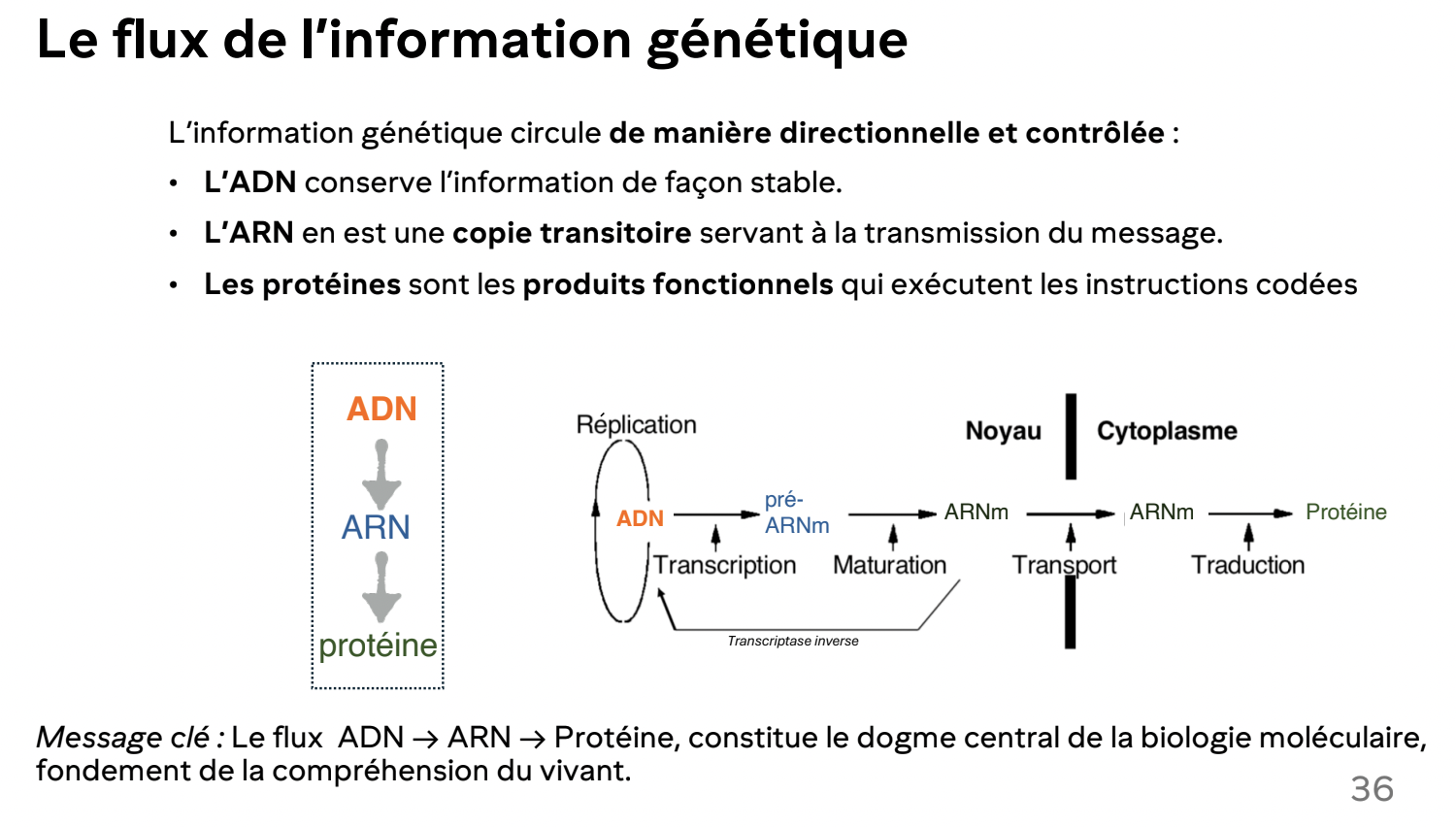
Formes stables vs transitoires de stockage génétique
Stable: ADN, stockage permanent
Transitoire: ARN (m, r, t, etc.), expression et régulation
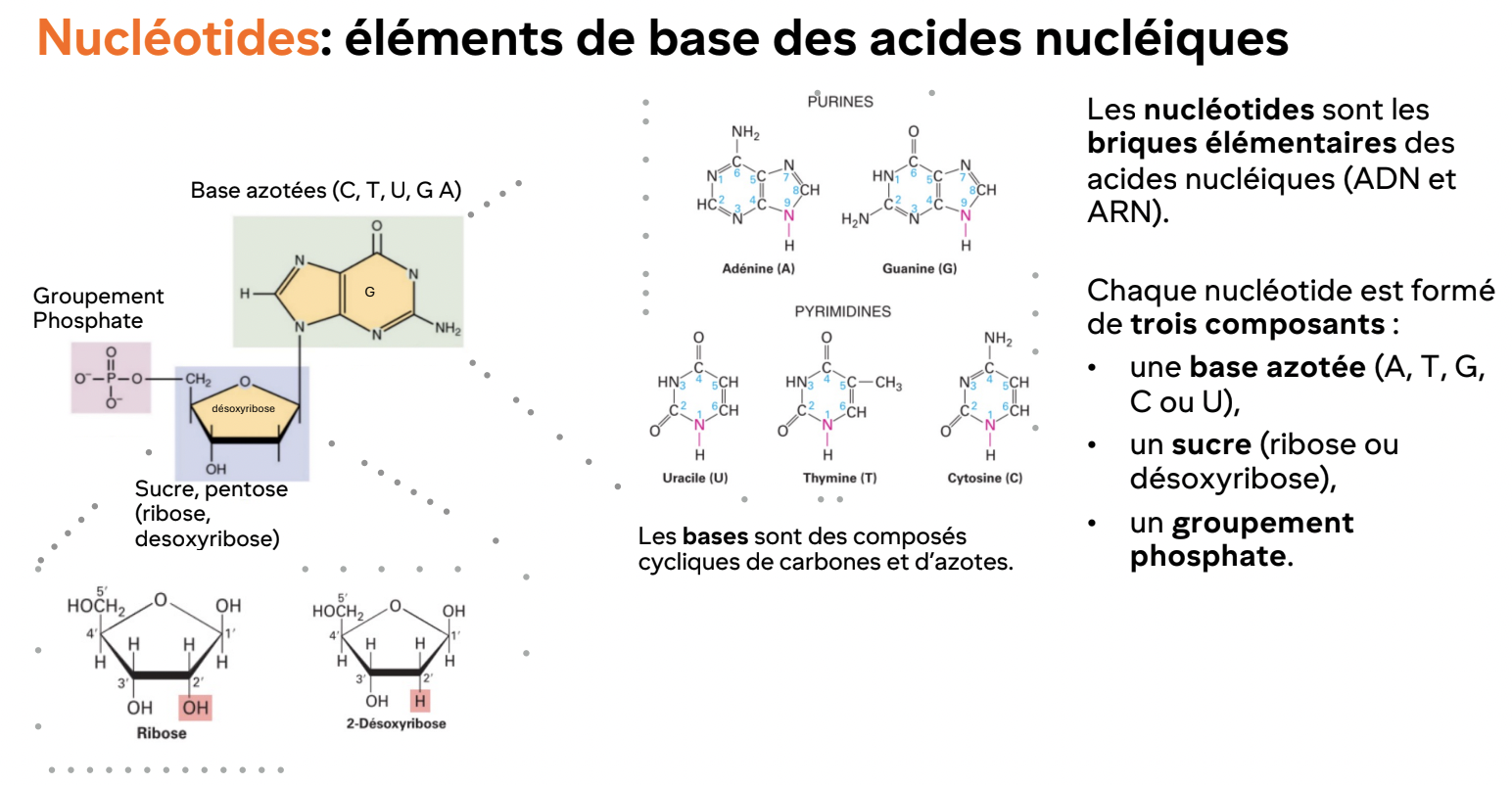
Structure d’un nucléotide
Base azotée (A, T/U, G, C)
Pentose (ribose ou déoxyribose)
Groupement phosphate
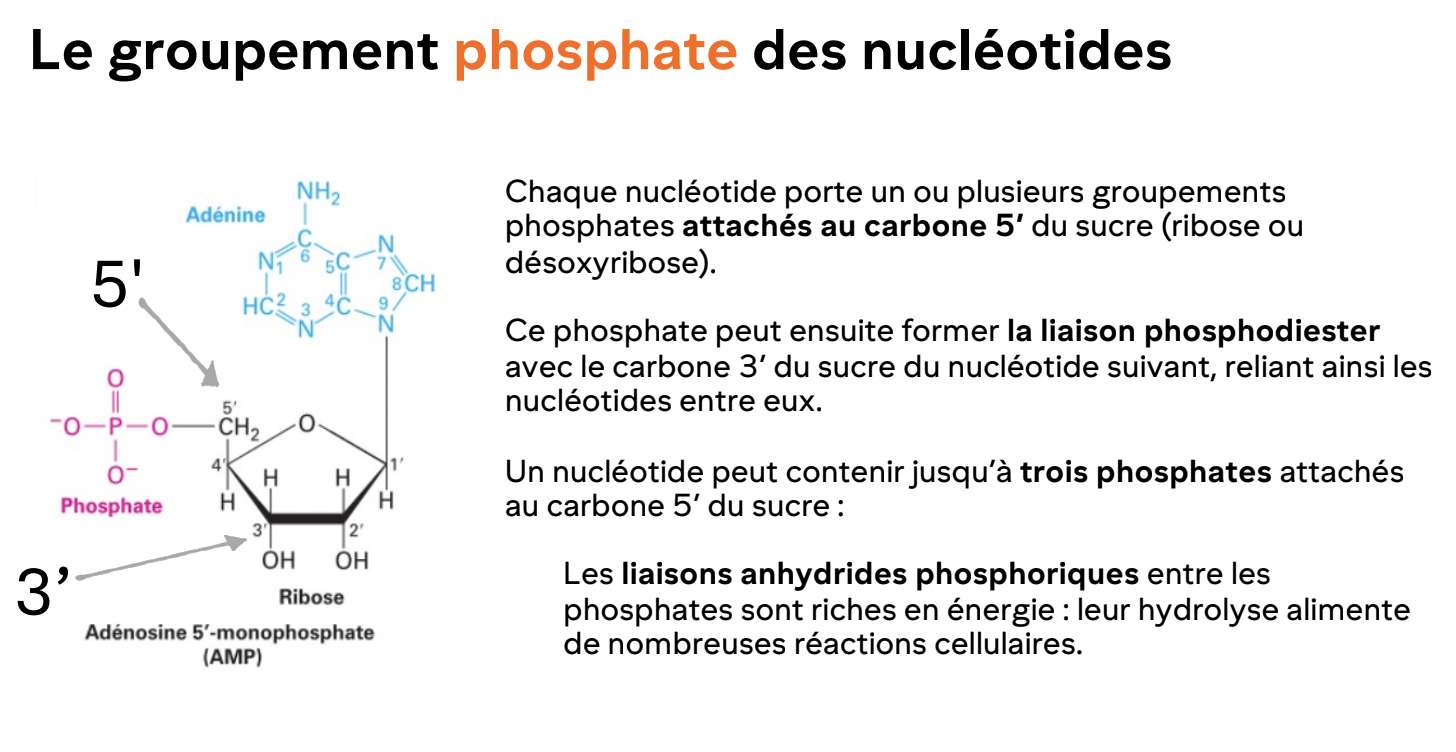
Importance des positions 5’ et 3’ sur le nucléotide
L’extrémité 5’ porte généralement le phosphate
L’extrémité 3’ porte un OH libre, indispensable à L’élongation de l’ADN/ARN
Toute synthèse se fait dans le sense 5’ à 3’
Différence Ribose vs Déoxyribose
Ribose (ARN): OH sur C2’
Déoxyribose (ADN): H sur C2’
Structure et rôle de l’ATP
Nucléotide dérivé de l’adénine
Composé d’un ribose + 3 phosphates liés par des liaisons phosphoanhydrides riches en énergie
Hydrolyse donne ADP ou AMP et libère de l’énergie pour les réactions cellulaires
Autres molécules dérivées de nucléotides et leurs fonctions
NAD+/FAD: Coenzymes d’oxydoréduction
CoA (Coenzyme A): Métabolisme énérgetique des acides gras
cAMP: Signalisation cellulaire (2nd messenger)
NMPc: exemples et fonctions
Les nucléotidescycliques (eg. cAMP, cGMP) sont des messagers intracellulaires
Permettent l’activation de kinases et la transmission hormonale
Qu’est-ce qu’une liaison phosphodiester 3’-5’?
Liaison reliant le phosphate en 5’ d’un nucléotide au OH en 3’ du suivant.
Donne un brin orienté: 5’ à 3’

Purines vs Pyrimidines
Purines: Adenine (A), Guanine (G), plus grandes et structures double ring
Pyrimidines: Cytosine (C), Thymine (T), Uracil (U), plus petites et structure single ring
Différence Thymine (T) vs Uracile (U)
Thymine = Uracil + CH3
Thymine = ADN
Uracile =ARN
Modifications de l’ADN et de l’ARN
ADN: méthylation de la cytosine, donne 5-méthylcytosine qui joue un rôle dans l’épigénétique
ARNt: bases modifiées permettant le repliement et la reconnaissance du codon
Déamination des bases + réparation
La cytosine peut se déaminer en uracile, erreur potentielle
La glycosylase reconnait l’erreur et l’enlève
Puis endonuclease + ADN polymérase + ligase réparent
ADA (déficit en adénosine désaminase)
empêche la dégradation de l’adénosine. Toxicité pour lymphocytes
Provoque immunodéficience sévère (SCID)
Règles d’appariement (Watson-Crick)
A-T, 2 liaisons H
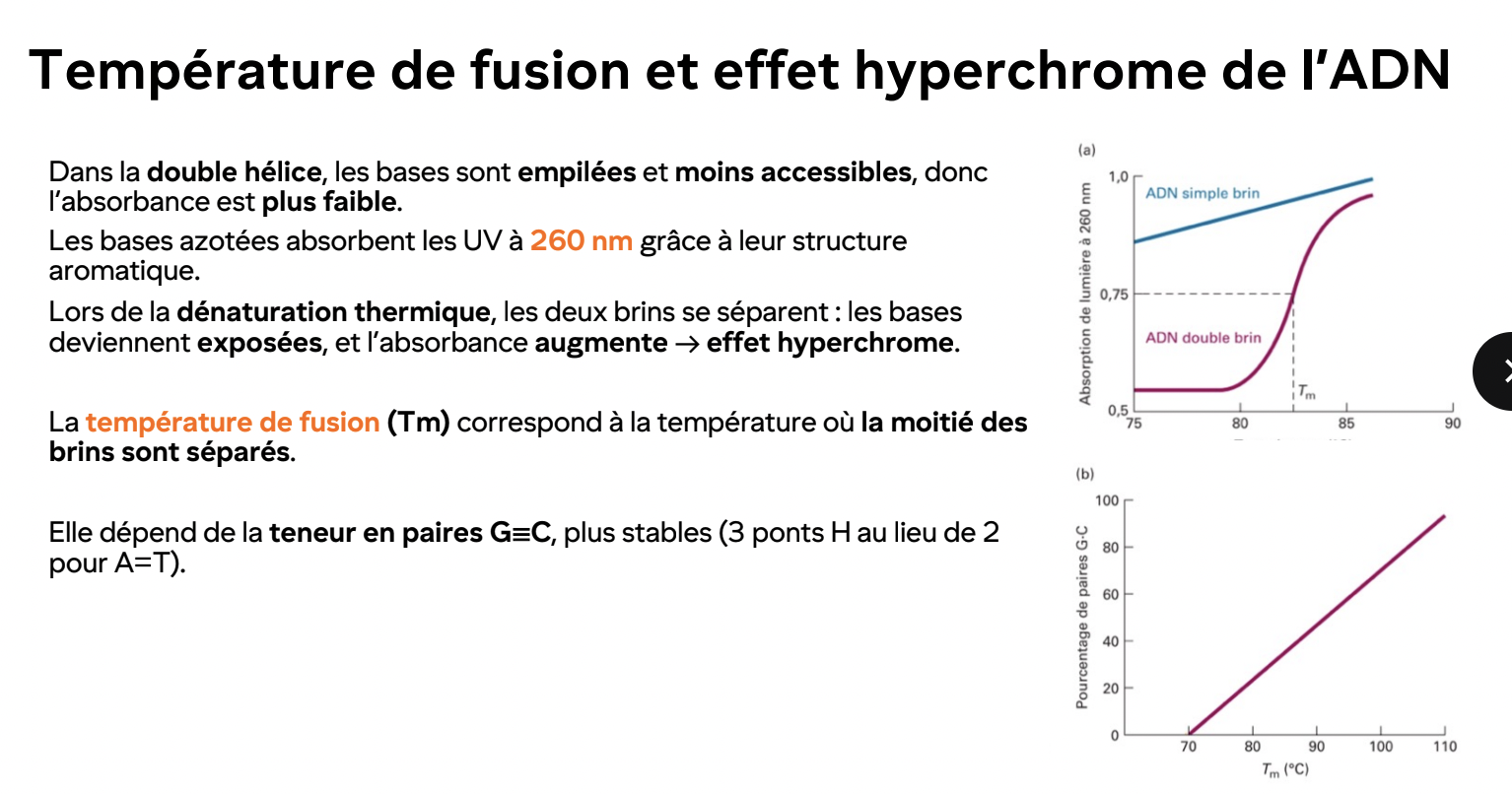
G-C, 3 liaisons H, therefore plus stable
Brins antiparallèles: 5’ à 3’ vs 3’ à 5’
Regions riches en G-C plus stables et plus haute Tm (temp. de fusion)
Effet hyperchrome
Lors de la dénaturation, l’ADN absorbe plus à 260 nm
Utilisé pour mesurer Tm et l’intégrité de l’ADN
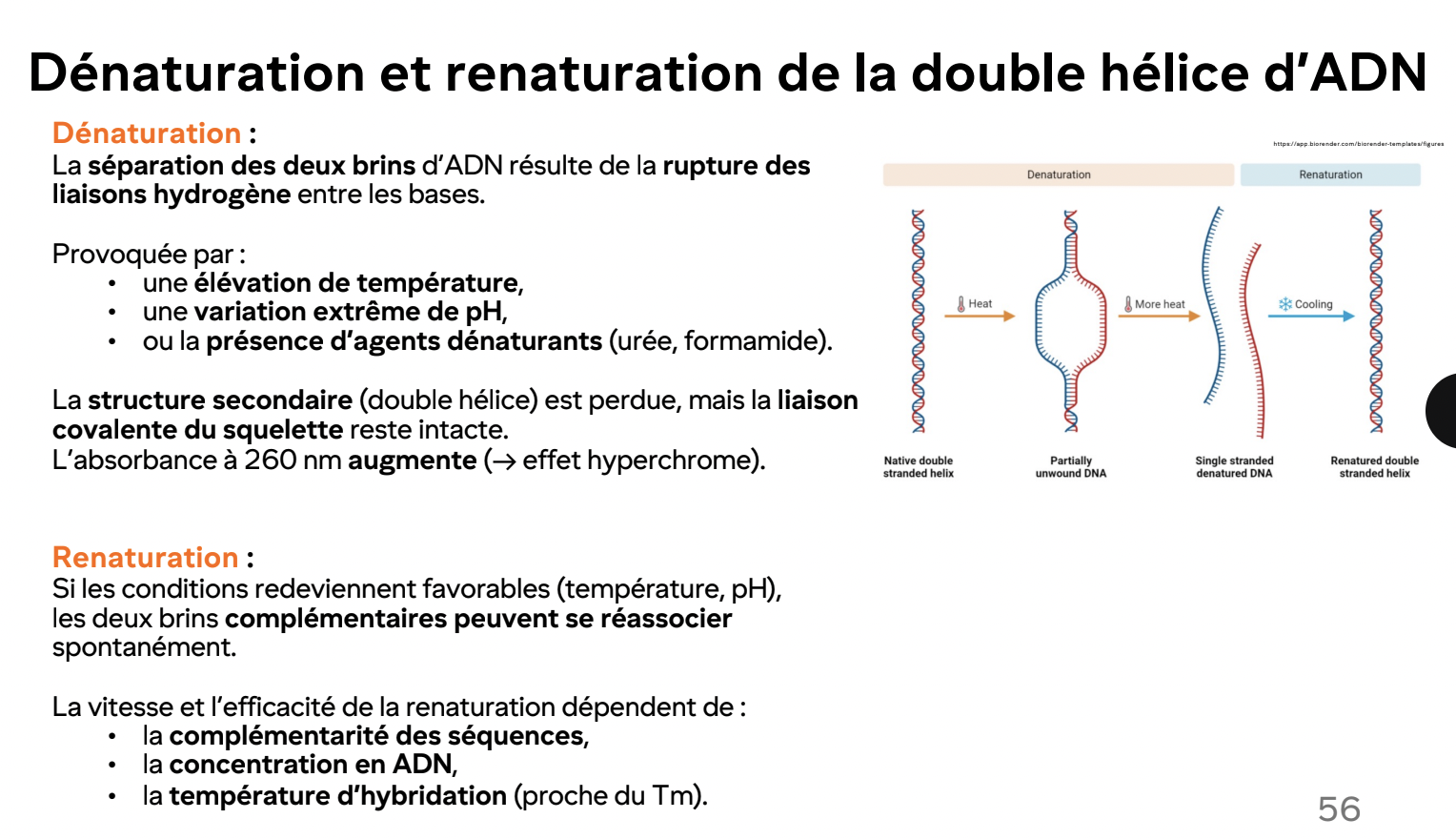
Facteurs incluençant la dénaturation de l’ADN
température haute: séparation des brins
pH extrème: rupture liaisons H
Ions (Na+) stabilisent la double hélice
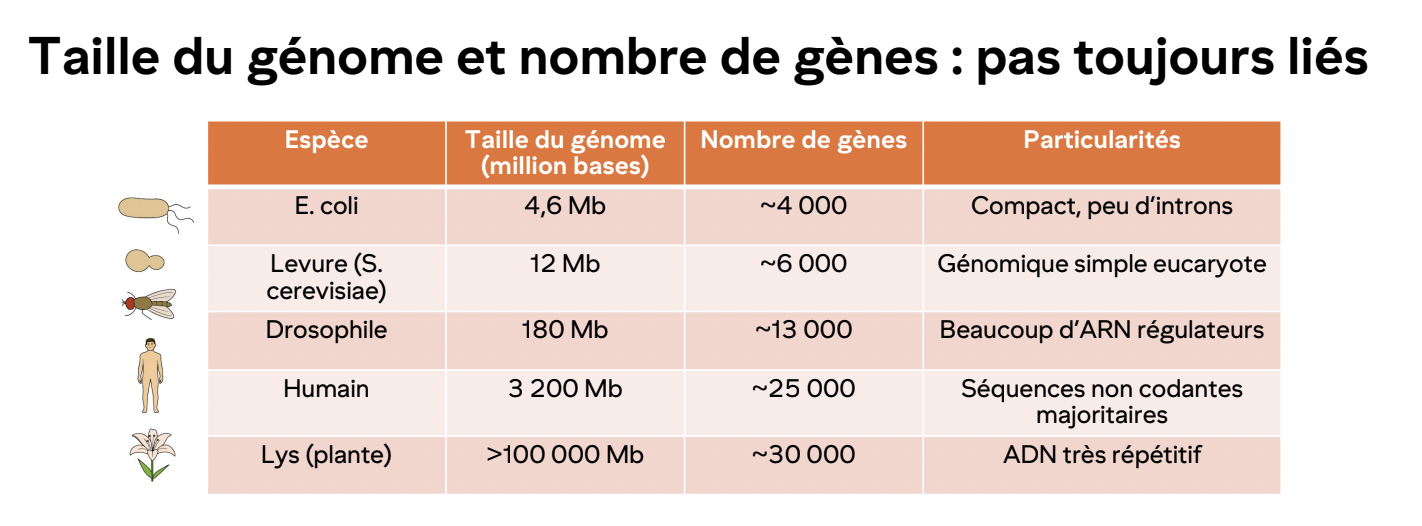
Nombre approximatif de gènes humains vs taille du génome

ADN codant vs. non codant
Codant: 1-2%, code protéines
Non codant: 98-99%, régulation, structure, expression
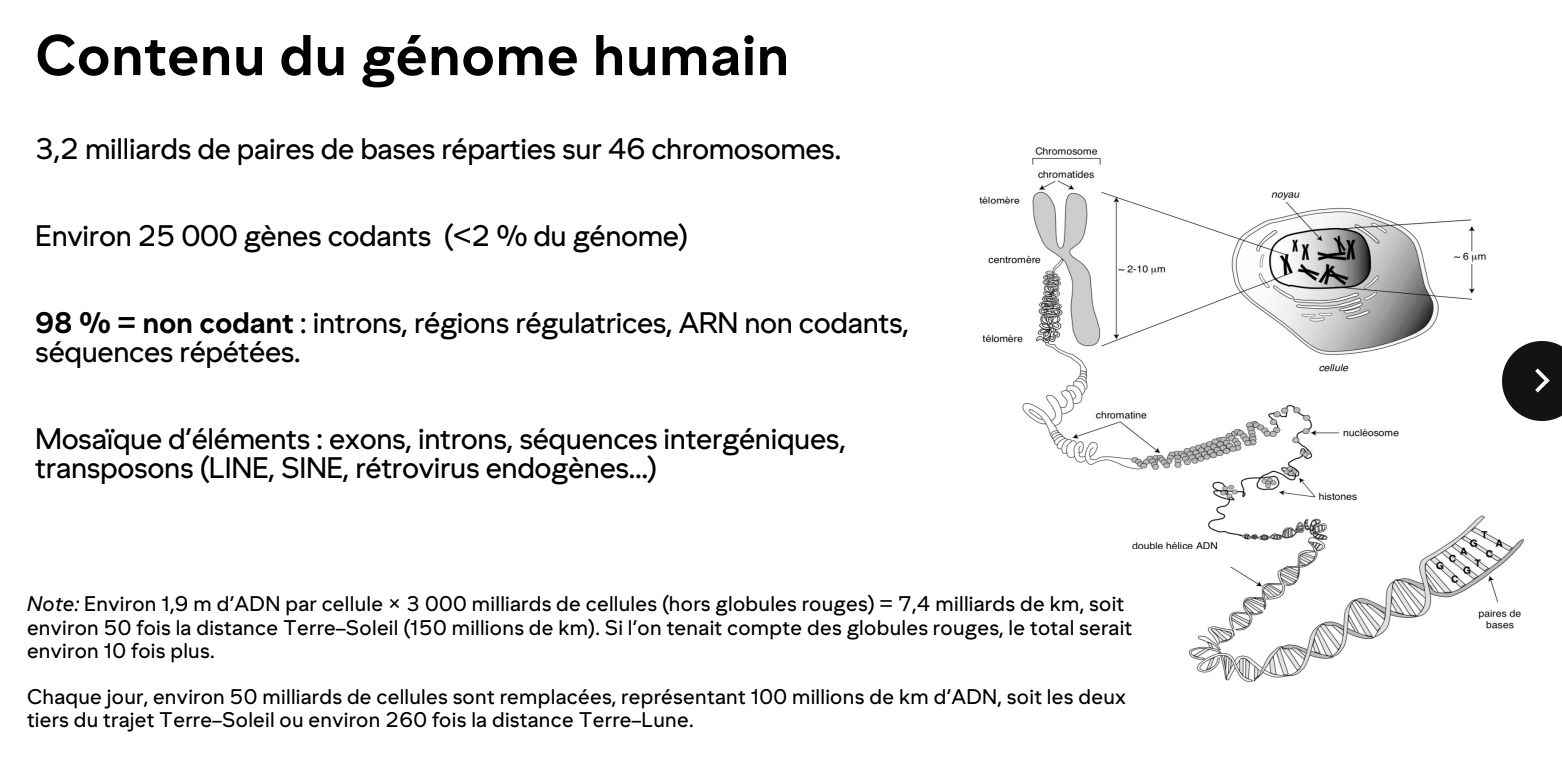
Types d’ADN non codant (ask ab this flashcard)
ADN satellite: régions répétées (centromères)
LINE: rétrotransposons autonomes
SINE: non autonomes (ex. Alu)
Insertions virales anciennes: éléments endogènes hérités
Paradoxe de la valeur C
Taille du génome n’est pas proportionelle à la complexité de l’organisme
Eg. Salamandre possède un génomeplus grand que l’humain
la différence vient du non codant

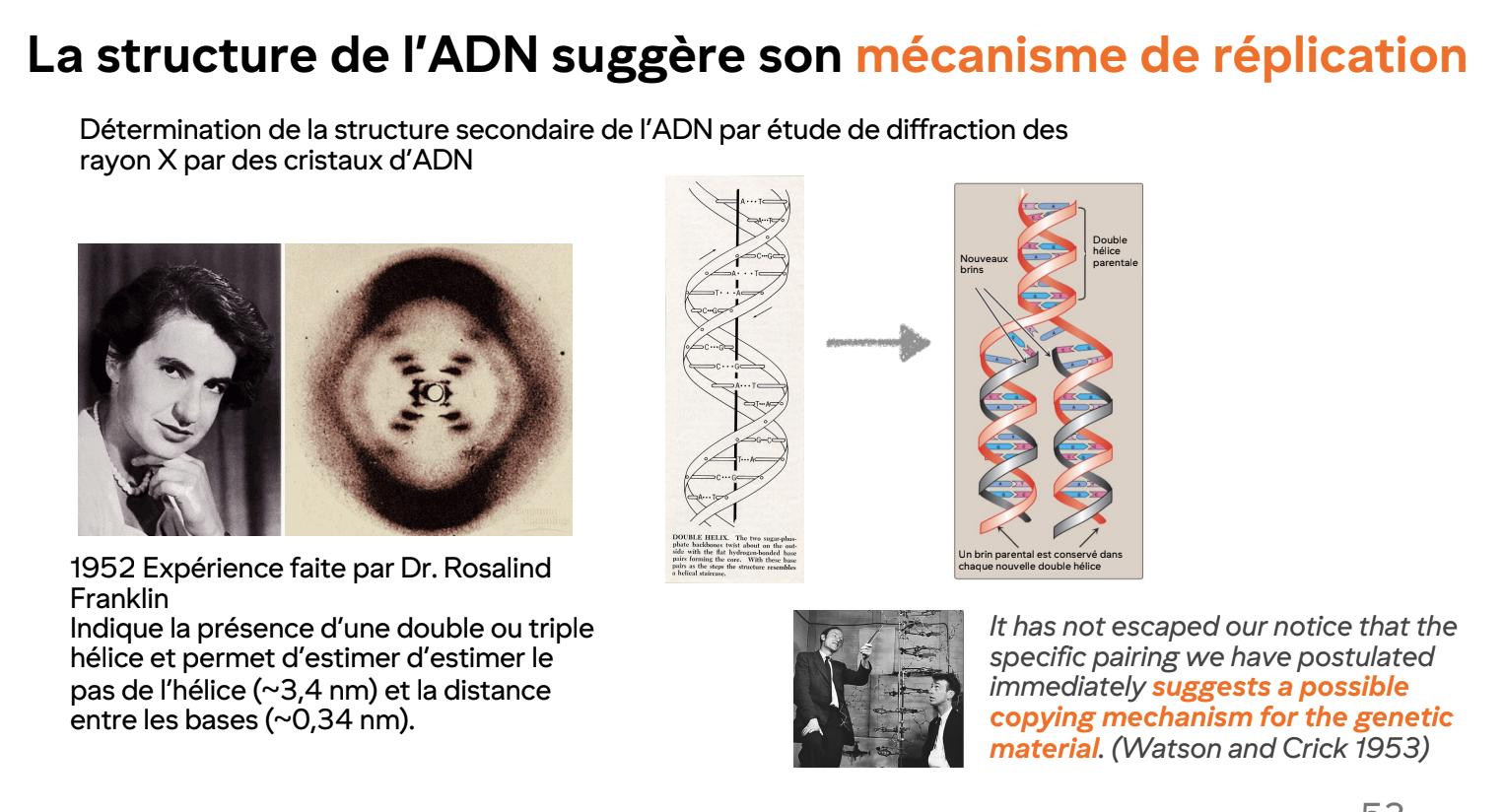
Contribution de Rosalind Franklin in ADN structure discovery
Cristallographie aux rayons X
Montre hélice + distance entre base
Sans elle, Watson et Crick n’auraient pas pu modéliser l’ADN