MB1 Minimalwissen
5.0(1)
Card Sorting
1/425
Earn XP
Description and Tags
HU Berlin Monobachelor Biologie MB1
Last updated 9:37 AM on 2/23/23
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No analytics yet
Send a link to your students to track their progress
426 Terms
1
New cards
Was ist die Aufgabe der Biochemie´?
Die Betrachtung von biologischen Vorgängen aus Sicht der Chemie.
2
New cards
Wie viele Aminosäuren bilden Peptidbindungen aus?
20 Aminosäuren.
3
New cards
Woraus bestehen Nukleinsäuren?
Purin- und Pyrimidinbasen, Zucker und einem Phosphatrest.
4
New cards
Was bildet biologische Moleküle und Makromoleküle aus?
Kovalente und nicht kovalente Bindungen bilden biologische Moleküle und Makromoleküle aus.
5
New cards
Zähle die biologischen Elemente auf.
Kohlenstoff C,
Wasserstoff H,
Sauerstoff O,
Stickstoff N,
Schwefel S,
Phosphor P,
Kalium K,
Calcium Ca,
Eisen Fe,
Magnesium Ma.
Wasserstoff H,
Sauerstoff O,
Stickstoff N,
Schwefel S,
Phosphor P,
Kalium K,
Calcium Ca,
Eisen Fe,
Magnesium Ma.
6
New cards
Nenne die Wechselwirkungen zwischen Biomolekülen.
Dipol-Dipol-Wechselwirkungen (Von-der-Waals-Kräfte),
Kovalente Bindung,
Wasserstoffbrückenbindung,
Elektrostatische Wechselwirkung (Ionische Bindung).
Kovalente Bindung,
Wasserstoffbrückenbindung,
Elektrostatische Wechselwirkung (Ionische Bindung).
7
New cards
Nenne die verschiedenen Stoffklassen.
Aminosäuren,
Kohlenhydrate,
Nucleotide,
Lipide.
Kohlenhydrate,
Nucleotide,
Lipide.
8
New cards
Nenne die Besonderheiten der wässrigen Umgebung für Lebensprozesse.
Wasser wird teilweise sogar als erstes Biomolekül bezeichnet, die menschliche Vorstellung von Leben ist unmittelbar mit Wasser verbunden. Wir können uns kein Leben ohne Wasser vorstellen.
9
New cards
Benenne verschiedene Aminosäure-Klassen.
Aminosäuren mit unpolaren und aromatischen Seitenketten (Glycin, Alanin, Valin),
Aminosäuren mit polaren Seitenketten (Serin, Cystein, Glutamin),
Aminosäuren mit geladenen Seitenketten (Asparaginsäure, Glutaminsäure, Lysin).
Aminosäuren mit polaren Seitenketten (Serin, Cystein, Glutamin),
Aminosäuren mit geladenen Seitenketten (Asparaginsäure, Glutaminsäure, Lysin).
10
New cards
Zeichne den Aufbau einer Aminosäure.
.
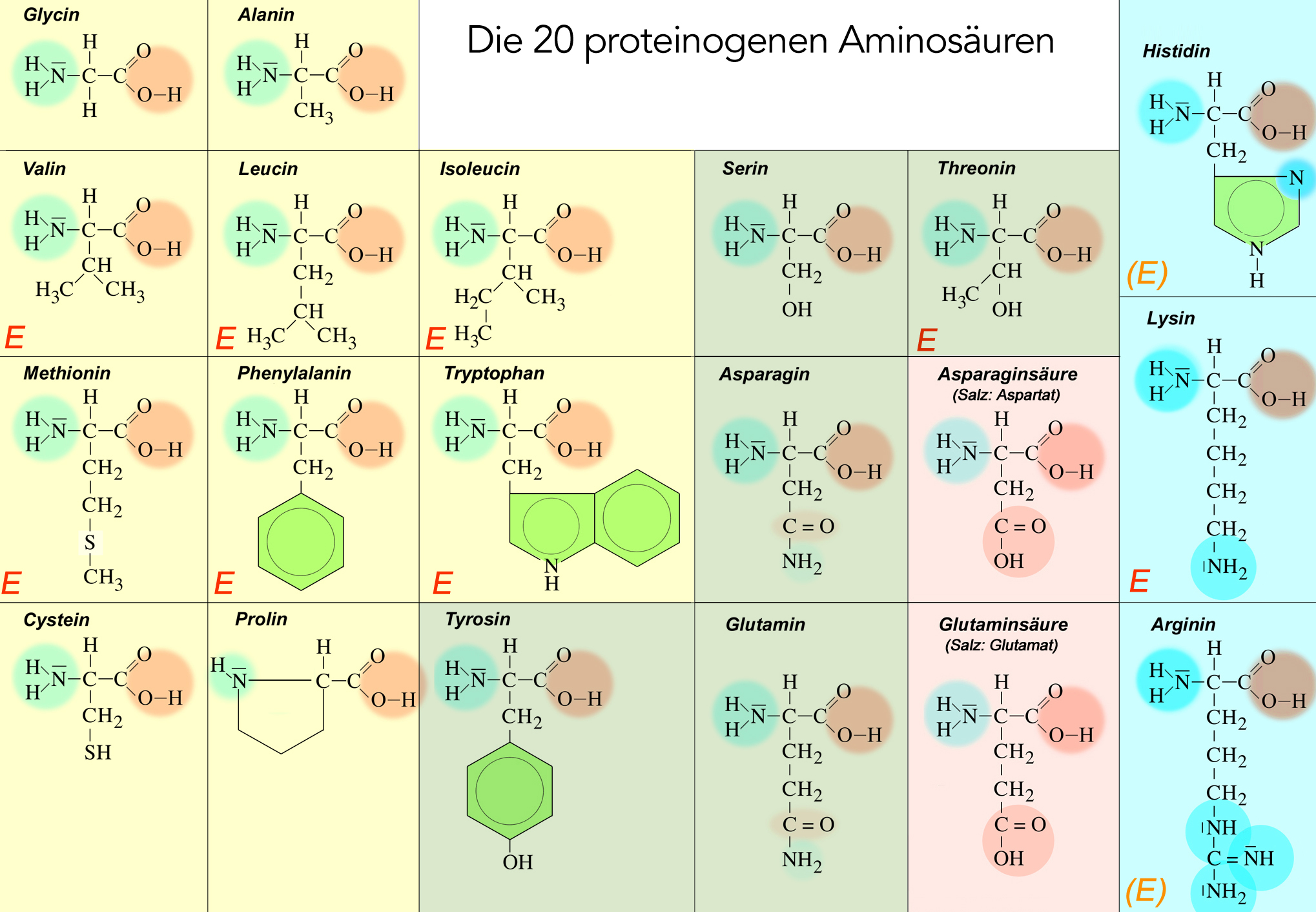
11
New cards
Welche Art von Aminosäure wird in Proteinen verwendet?
L-Aminosäuren.
12
New cards
Benenne den Unterschied zwischen Purin- und Pyrimidinbasen.
Pyrimidinbasen haben lediglich einen Pyrimidinring, Purinbasen haben jedoch einen Pyrimidinring und einen kondensierten Imidayolring, Purine haben mehr intermolekulare Wechselwirkungen und haben somit einen viel höheren Schmelz- und Siedepunkt.
13
New cards
Auf wie vielen Ebenen wird der Proteinaufbau unterschieden?
Der Proteinaufbau wird auf vier Ebenen unterschieden.
14
New cards
Was bestimmt den Aufbau der Proteine´?
Die Eigenschaften der Peptidbindungen bestimmen den Aufbau.
15
New cards
Was sind die beiden wichtigsten Sekundärstrukturen?
Die wichtigsten Strukturen sind α-Helix und β-Faltblatt.
16
New cards
Wie falten sich Proteine?
Proteine falten sich kooperativ.
17
New cards
Wieso wird der Aufbau von Proteinen in vier Ebenen unterschieden?
Weil scih Proteine in diesen vier Aufbaustadien befinden.
Die Primärstruktur ist eldiglich eine Aneinadnerreihung von Aminosäuren
Die Sekundärstruktur, welche in zwei Arten unterschieden wird ist eine in Form gebrachte Primärstruktur
Die Tertiärstruktur sind gefaltete Sekundärstrukturen
Die Quartärstruktur sind Strukturen, welche aus mehreren (min. 4) Tertiärstrukturen bestehen.
Die Primärstruktur ist eldiglich eine Aneinadnerreihung von Aminosäuren
Die Sekundärstruktur, welche in zwei Arten unterschieden wird ist eine in Form gebrachte Primärstruktur
Die Tertiärstruktur sind gefaltete Sekundärstrukturen
Die Quartärstruktur sind Strukturen, welche aus mehreren (min. 4) Tertiärstrukturen bestehen.
18
New cards
Wie sieht eine Peptidbindung aus´?
Eine Peptidbindung ist die Bidnugn zwischen zwei Aminosäuren. z.B.
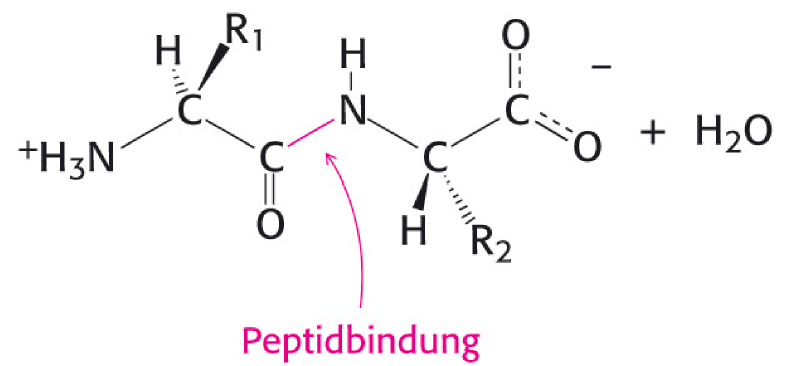
19
New cards
Bennene zwei Sekundärstrukturen und erläutere ihre Unterschiede im Aufbau.
Dei zwei Sekundärstrukturen sind α-Helix und β-Faltblatt.
Dabei ist die α-Helix als eben solche Helix angeordnet und wird durch regelmäßige Wiederholungen mit Wasserstoffbrückenbindungen stabilisiert.
\
Das β-Faltblatt kann entweder parallel oder antiparallel vorliegen, hierbei sind die Peptidbindungen in lang gestreckten Ketten vorhanden.
Dabei ist die α-Helix als eben solche Helix angeordnet und wird durch regelmäßige Wiederholungen mit Wasserstoffbrückenbindungen stabilisiert.
\
Das β-Faltblatt kann entweder parallel oder antiparallel vorliegen, hierbei sind die Peptidbindungen in lang gestreckten Ketten vorhanden.
20
New cards
Inwiefern unterscheidet sich ein gefaltetes von einem ungefaltetem Protein´?
Die Faltung eines Proteins ermöglicht dessen geplante Funktion. Ein denaturiertes, entfaltetes Protein verliert seine bisherige Funktion zusammen mit dem Formverlust.
21
New cards
Was ist ein Enzym?
Ein Enzym ist ein Protein, welches im Organismus als Biokatalysator wirkt. Es beschleunigt Reaktionen.
22
New cards
Was können Enzyme nicht?
Enzyme haben keinen Einfluss auf die Lage des Substrat - Produkt Gleichgewichts.
23
New cards
Benenne den elementaren, allgemeinen Mechanismus von Enzymen.
Enzyme binden Substrate, dann regaiert das Enzym-Substrat-Kompelx miteinander, das Produkt der Reaktion ist das Enzym-Produkt-Komplex,w elches zuletzt in die Lösung dissoziiert.
24
New cards
Beschreibe die Rolle der aktiven Zentren in der Enzymwirkung.
Das aktive Zentrum ist die Bindungsstelle für das Substrat. Hier bindet sich das Substrat und wird zu eionem Produkt umgesetzt.
25
New cards
Erkläre wieso die Enzymkinetik eine Substratsättigungskurve enthält.
Ab einer gewissen Substratkonzentration liegen alle Enzyme in Enzym- Substrat-Komplexen vor und damit ist die maximale Geschwindigkeit vmax für diese Enzymmenge erreicht.
26
New cards
Benenne Möglichkeiten der Regulation der Enzymaktivität.
Regulatorische Wechselwirkungen innerhalb der Enzyme (Kooperativität), regulatorisch-wirkende Moleküle (Allosterie),
Reversible kovalente Modifikationen, z.B. Phosphorylierung,
Aktivierender Spaltung (proteolztische Aktivierung),
Enzymmenge - Trankription, Translation, Enzymabbau.
Reversible kovalente Modifikationen, z.B. Phosphorylierung,
Aktivierender Spaltung (proteolztische Aktivierung),
Enzymmenge - Trankription, Translation, Enzymabbau.
27
New cards
Was sind Katalysatoren?
Katalysatoren beschleunigen die Einstellung von Reaktionsgleichgewichten.
28
New cards
Was sind aktive Zentren?
Bestandteile von Enzymen, in welchen Substrate gebunden und umgesetzt werden.
29
New cards
Wie können Größen, welche für die einzelnen Enzyme charakteristisch sind festgestellt werden?
Durch Messung der konzentrationsunabhängigen Reaktionsgeschwindigkeit.
30
New cards
Wodurch kann die Aktivität der Enzyme reguliert werden?
Regulatorische Wechselwirkungen innerhalb der Enzyme (Kooperativität), regulatorisch-wirkende Moleküle (Allosterie),
Reversible kovalente Modifikationen, z.B. Phosphorylierung,
Aktivierender Spaltung (proteolztische Aktivierung),
Enzymmenge - Trankription, Translation, Enzymabbau.
Reversible kovalente Modifikationen, z.B. Phosphorylierung,
Aktivierender Spaltung (proteolztische Aktivierung),
Enzymmenge - Trankription, Translation, Enzymabbau.
31
New cards
Beschreibe den Unterschied zwischen katabolen und anabolen Stoffwechselwegen.
Katabole Stoffwechselwege beschreiben Vorgänge, bei welchen aus Brennstoffen CO2, H2O und nutzbare Energie erzeugt wird.
\
Anabole Stoffwechselwege erzeugen im Gegensatz dazu komplexe Moleküle (z.B. Synthese von Glucose, Fett, etc.)
\
Demnach ist das Produkt der beiden Stoffwechselwege grundlegend verschiedend.
\
Anabole Stoffwechselwege erzeugen im Gegensatz dazu komplexe Moleküle (z.B. Synthese von Glucose, Fett, etc.)
\
Demnach ist das Produkt der beiden Stoffwechselwege grundlegend verschiedend.
32
New cards
Erläutere woher die Energie zum Aufbau von ATP in den Zellen kommt.
Die Energie wird entweder durch Zellatmung, Photosynthese, oder durch Zersetzung von energiereichen Molekülen gewonnen.
33
New cards
Warum kann der Metabolismus als die Geschichte der Wanderung von Elektronen erzählt werden?
Jeder Vorgang des Metabolismus benötigt irgendeine Form von Energie oder gibt Eenrgie ab. Somit kannd er Metabolismus ohne Elektronen gar nicht aufrecht erhalten werden. Da sie aber von einem Metabolismus Vorgang zum anderen geschickt werden, simbolysiert dies alles eine Art Wanderung.
34
New cards
Welche drei wiederkehrende Muster der verschiedenen Stoffwechselwege gibt es?
1. Aktivierte Trägermoleküle (NAD(P)H, FAD, Coenzym A)
2. Ähnliche bzw. gleiche Schlüsselreaktionen wiederholen sich im Stoffwechsel
3. Drei Arten der Regulation von Stoffwechselwegen
35
New cards
Was ist ein kataboler Stoffwechselweg?
Ein kataboler Stoffwechselweg erzeugt Energie in verschiedenen Energiestoffwechselschritten.
36
New cards
Was ist ein amphiboler Stoffwechselweg?
Ein amphiboler Stoffwechselweg läuft in einigen Schritten katabol und in adneren anabol ab.
37
New cards
Was ist der Unterschied zwischen katabolen und amphibolen Stoffwechselwegen?
Katabole Stoffwechselwege sind reine Energiestoffwechselwege, amphibole Stoffwechselwege sind jedoch katabol, sowie anabol.
38
New cards
Was ist ein phototropher Organismus?
Organismen, welche Sonnenlicht als Energiequelle nutzen.
39
New cards
Was ist ein chemotropher Organismus?
Organismen, welche oxidierbare Verbindungen als Energiequelle nutzen.
40
New cards
Beschreibe den Dreistufen-Prozess der Energiegewinnung.
1. Größere Moleküle werden abgebaut
2. Zentrale Metabolite werden gebildet
3. ATP-Bildung aus Acetyl-CoA.
41
New cards
Nenne die sechs Reaktionstypen.
Oxidreduktase,
Transferase,
Hydrolase,
Lyase,
Isomerase,
Ligase.
Transferase,
Hydrolase,
Lyase,
Isomerase,
Ligase.
42
New cards
Benenne Möglichkeiten der Regulation von Stoffwechselwegen.
Kontrolle der Enzymmenge,
Kontrolle der Enzymaktivität,
Verfügbarkeit von Substraten,
Energieladung von Zellen.
Kontrolle der Enzymaktivität,
Verfügbarkeit von Substraten,
Energieladung von Zellen.
43
New cards
Ws waren die entwicklungsgeschichtlich ersten Lebewesen?
Die ersten entwicklungsgeschichtlcihen lebewesen waren die Prokaryoten
44
New cards
Was umfasst die Gruppe der Prokaryoten?
Bacteria,
Archaea.
Archaea.
45
New cards
Im Vergleich mit eukaryotischen Zellen ist die Kompartimentierunf prokaryotischer Zellen … ausgeprägt.
geringer
46
New cards
Wo finden Atmungstoffwechsel und photosynthetische Lichtreaktionen bei Prokaryoten statt?
In der Cytoplasmamembran oder davon abgeleiteten Einfaltungen, bzw. Abschnürungen.
47
New cards
Wie groß sind Prokaryotische Zellen im Durschschnitt?
DIe Größe bewegt sich im Mikrometer Bereich,e s gibt jedoch auch größere und kleinere Vertreter.
48
New cards
Was bilden manche Prokaryoten aus?
Spezielle Zelleinschlüsse und Organellen.
49
New cards
Welche Art von Lebewesen haben verhäuft extremophile Lebensweisen?
Prokaryoten, spezifischer, meistens Achaeen.
50
New cards
Was sind pathogene Bakterien?
Pathogene Bakterien sind Pathogene, alo Krankheitsauslöser. Sie verursachen verschiedenste Krankheiten, welche dann oftmals mit Antibiotika behandelt werden können.
51
New cards
Beschreibe die Rolle von Prokaryoten in der Natur.
DIe Rolle von Prokaryoten in der Natur ist ambivalent, sie ist sehr wichtig in Stoffkreisläufen,
Wichtig für die Vitalität von Tieren und Pflanzen,
Wichtig für Herstellung einiger Lebensmittel,
jedoch auch pathogen.
Wichtig für die Vitalität von Tieren und Pflanzen,
Wichtig für Herstellung einiger Lebensmittel,
jedoch auch pathogen.
52
New cards
Welche Beziehung besteht zwischen Prokaryoten und eukaryotischen Organismen verhäuft?
Prokaryoten stehen oftmals in vielfältigen Symbiosen mit eukaryotischen Organismen.
53
New cards
Was ist in allen lebenden Zellen der Träegr der Erbinformation?
Doppelsträngige DNA ist in allen lebenden Zellen die Trägerin der Erbinformation.
54
New cards
In welcher Form liegt die DNA in einem Prokaryoten dar?
In Form eines einzelnen zirkulären Chromosoms. (auch Bakterienchromosom)
55
New cards
Welche Aufgabe tragen Topoisomerasen im Organismus?
SIe regulieren den Überspiralisierungszustand der DNA.
56
New cards
Was erzeugt geordnete Domänenstrukturen in Chromosomen?
Nukleoid assozierte Proteine (NAPs),
Kondensine,
(Archaea - Histone).
Kondensine,
(Archaea - Histone).
57
New cards
Wie erfolgt die Replikation von DNA?
Die Repliaktion erfolgt semikonservativ und bidirektional.
58
New cards
Was ist die Aufgabe der DNA-Polymerase I?
Sie entfernt die RNA-Primer und ersetzt sie durch DNA.
59
New cards
Was trägt die DNA-Ligase zur DNA-Replikation bei?
Sie verschließt die Lücken im Zucker-Phosphat-Rückrat.
60
New cards
Was benötigen DNA-Polymerasen um DNA zu replizieren?
(RNA-)Primer
61
New cards
In welche Richtung werden Primer angesetzt?
5’-3’ Richtung.
62
New cards
Was passiert während der DNA Replikation am Leitstrang?
Innerhalb von Repliomen synthetisieren zwei Kopien der DNA-Polymerase III den Leitstrang kontinuierlich.
63
New cards
Was passiert während der DNA Replikation am Folgestrang?
Der Folgestrang wird diskontinuierlich synthetisiert mithilfe von Okazaki-Fragmenten.
64
New cards
Was geschieht während der Transkription? (grob)
Es werden Gene (DNA) in mRNA oder andere RNA-Arten umgeschrieben.
65
New cards
Woraus bestehen bakterielle RNA-Polymerasen?
Aus den Untereinheiten:
Alpha2,
Beta,
Beta’,
Omega.
(zur Promoter-Erkennung ebenfalls Sigma).
Alpha2,
Beta,
Beta’,
Omega.
(zur Promoter-Erkennung ebenfalls Sigma).
66
New cards
Woraus bestehen archaebakterielle RNA-Polymerasen?
Aus komplexen Verbindungen, sie ähneln der RNA-Polymerase II (Eukaryoten).
(Zur Promotorerkennung sich Transkriptionsfaktoren notwendig).
(Zur Promotorerkennung sich Transkriptionsfaktoren notwendig).
67
New cards
Wie wird die Termination bei Bakterien eingeleitet?
An Haarnadelschleifen + Poly-U-Folgen,
oder faktorabhängig unter Beteiligung des Rho-Proteins.
oder faktorabhängig unter Beteiligung des Rho-Proteins.
68
New cards
Wie wird die Termination bei Archaebakterien eingeleitet?
An Poly-U-Folgen,
oder an Haarnadelschleifen + Poly-U-Folgen.
oder an Haarnadelschleifen + Poly-U-Folgen.
69
New cards
Wie sind die Gene in Prokaryoten oftmals organisiert?
In Operons.
70
New cards
Was ist ein Operon?
Einheit von gemeinsam regulierten Genen.
71
New cards
Beschreibe den Ablauf der Transkription.
Bei der Transkription wird die DNA in eine mRNA umgeschrieben. Der Ausgangspunkt ist also die DNA, die Desoxyribonucleinsäure. Allerdings wird nicht die komplette DNA umgeschrieben, sondern nur ein kleiner Teil, der gerade für die Proteinherstellung benötigt wird.
72
New cards
Beschreibe den Ablauf der DNA-Repliaktion kurz.
1. Initiation
Während der Initiation wird der DNA-Doppelstrang entspiralisiert und geöffnet. Dabei spielen die Enzyme **Topoisomerase** und **Helikase** eine besondere Rolle. Zuerst entspiralisiert die Topoisomerase die DNA-Doppelhelix. Anschließend trennt das Enzym Helikase die Wasserstoffbrücken und somit die DNA-Stränge voneinander. Danach lagern sich Proteine an die getrennten Abschnitte der DNA an, um zu verhindern, dass sich die Einzelstränge wieder verbinden oder von DNA-spaltenden Enzymen angegriffen werden.
2. Elongation
Der nächste Schritt, die Elongation, beginnt mit der Synthese der **Primer** an den DNA-Einzelsträngen. Die Primer, welche aus wenigen **Nukleotiden** bestehen, werden hierbei von dem Enzym **Primase** am 3'-Ende angelagert.
Die Primer bestehen genauer gesagt aus RNA (Ribonukleinsäure). Statt der für die DNA übliche Base **Thymin** enthält die RNA die Base **Uracil**. Daher ist es wichtig, dass die Base Uracil bei der DNA-Replikation durch Thymin ausgetauscht wird.
Die Primer dienen der DNA-Polymerase als **Startsequenz**. Das Enzym lagert sich an die Primer an und verknüpft die komplementären Basen mit dem DNA-Strang in 5'-3'-Richtung (sprich: *fünf-zu-drei-Strich*). Der Ablauf der DNA-Replikation unterscheidet sich zwischen Leit- und Folgestrang.
**Elongation des Leitstrangs**
Die DNA-Polymerase wandert an den Tochtersträngen vom 5'-Ende zum 3'-Ende. Am **Leitstrang** bewegt sie sich also in die gleiche Richtung wie das Enzym Helikase. Die Ergänzung des Einzelstrangs kann daher ohne Unterbrechungen ablaufen. Die Synthese des Doppelstrang läuft somit am Leitstrang kontinuierlich ab.
**Elongation des Folgestrangs**
Beim Ergänzen des **Folgestrangs** kommt es zu einer Besonderheit, denn der DNA-Strang ist hier im Vergleich zum kontinuierlichen Strang genau entgegengesetzt (antiparallel) aufgebaut.
3. Termination.
73
New cards
Wofür sind Aminoacyl-tRNA-Syntheasen verantwortlich?
Für das Beladen der tRNAs mit AMinosäuren und für die Genauigkeit der Translation.
74
New cards
Woraus bestehen Prokaryotische (70S-)Ribosomen?
Sie bestehen aus einer 50S- und einer 30S-Untereinheit,
Beide Untereinheiten enthalten RNA und eine vielzahl von Proteinen.
Beide Untereinheiten enthalten RNA und eine vielzahl von Proteinen.
75
New cards
Was bildet den Initiationskomplex?
30S-Untereinheit des Ribosoms,
IF1-3,
mRNA,
fMet-tRNA(fMet).
IF1-3,
mRNA,
fMet-tRNA(fMet).
76
New cards
Was beinhalten Posttranslationale Prozesse?
Assistierte Faltung,
den Einbau von Kofaktoren,
die Dirigierung zum Wirkort,
kovalente Modifikation von Aminosäureseitenketten
den Einbau von Kofaktoren,
die Dirigierung zum Wirkort,
kovalente Modifikation von Aminosäureseitenketten
77
New cards
Welche Vorgänge laufen zur Elongation in der Zelle ab?
Beladene tRNAs werden mithilfe von EF-Tu an den A-Ort des Ribosomosen abgeliefert,
nach Knüpfung einer Peptidbindung wird die wachsende Peptidkette durch EF-G in den P-Ort translosziert und die freie tRNA entlassen.
nach Knüpfung einer Peptidbindung wird die wachsende Peptidkette durch EF-G in den P-Ort translosziert und die freie tRNA entlassen.
78
New cards
Was sind die drei Stoppcodons?
UAA,
UAG,
UGA.
UAG,
UGA.
79
New cards
Wozu führen die Stoppcodons zusammen mit den Freisetzungsfaktoren, sowie RRF und EF-G?
Zum Translationsstopp und zur Freisetzung der Peptidkette und zur Trennung der 30S- von der 50S-Untereinheit des Ribosoms.
80
New cards
Was wird zum Translationsstopp benötigt?
Eines der drei Stoppcodons, die Freisetzungsfaktoren RF1, RF2, RF3, RRF, EF-G.
81
New cards
Was ist die Aufgabe des lac-Operons?
Lactose-Operon, lac-Operon, das Operon, das die Gene enthält, welche für den Lactosemetabolismus von Escherichia coli erforderlich sind. Sie codieren für Enzyme, die die Aufnahme des Disaccharids in die Zelle sowie für dessen Hydrolyse verantwortlich sind.
82
New cards
Wie wird die Expression des lac-Operons reguliert?
1. Durch Repressor Lacl reprimiert (Bindung eines Induktors hebt Repressorfunktion auf),
2. Durch CRP (CAP-cAMP) aktiviert.
83
New cards
Was hebt die Repressorfunktion von Lacl auf?
die Bindung eines Induktors an Lacl hebt dessen Repressorfunktion auf.
84
New cards
Was steuert die Verfügbarkeit von Sigma-Faktoren?
Die vorhandene Menge an Anti-Sigma-Faktoren.
85
New cards
Nenne ein Beispiel für einen globalen Regulationsmechanismus unter Mangelbedingungen.
Die stringente Kontrolle.
86
New cards
Was ist die stringente Kontrolle?
Eine Anpassung des Stoffwechsels von Prokaryoten auf eine Unterversorgung mit Nährstoffen wie Kohlenstoff oder Aminosäuren.
87
New cards
Was bezeichnet man als Quorum Sensing?
Die Regulation in Abhängigkeit von der Zelldichte (z.B. Produktion des Luciferase-Systems in Leuchtbakterien).
88
New cards
Wie wird in Zwei-Komponenten-Systemen Information weitergeleitet?
Eine sensorische Histidin-Protein-Kinase phosphoryliert sich selbst und überträgt die Phosphoryl-Gruppe auf einen Antwort-Regulator.
89
New cards
Was regulieren die sRNAs auf RNA-Ebene?
Die Stabilität von mRNAs, oder die Translationsinitiation.
90
New cards
Was regulieren die Riboswitches nach Metabolitbindung auf RNA-Ebene?
Die Translationsinitiation.
91
New cards
Was reguliert Attenuation auf RNA-Ebene?
Die vorzeitige Termination der Transkription.
92
New cards
Was ist der Unterschied zwischen Gram-positiven und Gram-negativen Bakterien?
Gram-negative Bakterien sind entwicklungsgeschichtlich älter, sie haben eine äußere Membran, welche den Gram-positiven Bakterien fehlt. Weiterhin haben Gram-positive Bakteriendicken Zellwände.
93
New cards
Nenne die Grundstrukturen der bakteriellen Zellhülle.
Cytoplasmamembran,
Zellwand,
Periplasma von Gram-negativen Bakterien,
Äußere Membran von Gram- negativen Bakterien,
Kapseln und andere Oberflächenstrukturen. Kapseln und andere Oberflächenstrukturen.
Zellwand,
Periplasma von Gram-negativen Bakterien,
Äußere Membran von Gram- negativen Bakterien,
Kapseln und andere Oberflächenstrukturen. Kapseln und andere Oberflächenstrukturen.
94
New cards
Woraus besteht die Zellwand?
Aus Peptidoglykan (Peptid & Zucker) oder Murein (Schutzschicht).
95
New cards
Woraus besteht die Cytoplasmamembran?
Aus Phosphorlipiden,
negativ geladene Fettsäuren,
Kanalproteine.
(Biomembran)
negativ geladene Fettsäuren,
Kanalproteine.
(Biomembran)
96
New cards
Nenne die Funktionen der Cytoplasmamembran.
1\. Permeabilitätsbarriere,
2\. Proteinverankerung,
3\. Energiekonservierung.
2\. Proteinverankerung,
3\. Energiekonservierung.
97
New cards
Erkläre den Ausdruck „Aktiver Transport".
Der aktive Transport ist einer der Transporttypen, welche in der Biomembran abläuft. Dabei benötigt der aktive Transport zusätzliche Energie, er kann nicht passiv ablauten , der Vorteil, welcher aus dieser Einschränkung entspringt ist, dass der Transport sehr viel schnellen und effektiver abläuft. („gegen das Konzentrationsgefälle”)
98
New cards
Welche Transportsysteme gibt es in der Cytoplasmamembran?
1\. Einfacher Transport (durch Protonenmotorische Kraft (Symporter, Antiporter).
2\. Gruppentranslokation (Ziel: Glucose-Transport)
3\. ABC-System (ATP-Binding-Caset, synthetisiert ATP (Konfirmationsänderungen)
2\. Gruppentranslokation (Ziel: Glucose-Transport)
3\. ABC-System (ATP-Binding-Caset, synthetisiert ATP (Konfirmationsänderungen)
99
New cards
Was ist Murein/ Peptidoglykan?
Peptidoglykan, auch Murein genannt, ist ein Biopolymer, das aus Zuckermolekülen und Aminosäuren aufgebaut ist. Es ist der wichtigste Bestandteil der Zellwand vieler Bakterienarten (Eubakterien).
100
New cards
Was sind die primäre Funktion der bakteriellen Zellwand?
Stabilität, denn die Zellwand muss dem intrazellulären Druck standhalten können.
Formgebung, die Zellwand bestimmt die Form der Zelle.
Unbegrenztes Wachstum, mit stoffwechselaktiven Kompartiment.
Formgebung, die Zellwand bestimmt die Form der Zelle.
Unbegrenztes Wachstum, mit stoffwechselaktiven Kompartiment.