T13 | Genética Bacteriana 1
1/19
Earn XP
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
20 Terms
Diferentes mecanismos de variabilidade genética
Podem ser fenotípicas ou genotípicas
Fenotípicas
Indução enzimática → Produção de uma enzima só no meio em que é necessária.
Genotípicas
Mutações, recombinações, aquisição de genes e rearranjos intramoleculares.
Mutações
Alterações hereditárias na sequência de DNA
Podem levar a uma alteração nas propriedades de um organismo
Assim, podemos distinguir três tipos de mutações:
Benéficas: a proteína codificada pelo gene mutado resulta numa atividade benéfica
Prejudiciais: alteração num gene que codifica para uma proteína funcionalmente fundamental
Neutras/Silenciosas: alteração das bases não conduz à alteração da atividade do produto codificado pelo gene.
Nas bactérias, sendo procariontes haploides, quando possuem uma mutação esta é sempre expressa (excetuando no caso das mutações silenciosas, mas se a mutação conduzir a uma alteração fenotípica vai-se sempre manifestar, porque não há dominância de gene – só existe um).
As mutações podem ocorrer de duas formas:
Espontâneas: surgem ocasionalmente em todas as células e ocorrem sem a intervenção externa (resultam habitualmente de erros durante a replicação, de lesões no DNA ou por ação de elementos genéticos móveis).
Induzidas: são resultado da exposição a agentes físicos ou químicos, a que se dá o nome de mutagénio.
Mutação pontual: alteração em apenas um par de nucleótidos.
Deteção de mutações
Aspeto das colónias → Este método de deteção de mutações é bastante simples em que uma bactéria mutante origina colónias de diferentes formas.
Mutantes resistentes a agentes antimicrobianos → De modo a isolar bactérias resistentes a um determinado agente microbiano recorre-se a um processo seletivo. Num meio de cultura colocamos o agente microbiano e as bactérias e só as resistentes (mutadas) proliferam.
É também útil para avaliar a resistência a bacteriófagos, por mutações nos recetores, por exemplo.
Mutantes auxotróficos → Mutantes que não são capazes, por exemplo, a capacidade de sintetizar a lisina. - Experiência de Luria e Delbruck
Taxa de mutação
É o número de mutantes formados por divisão celular e é expressa como o nº de novas mutações num determinado gene /geração
A maioria das mutações pontuais resultam de erros na replicação
Durante a replicação ocorrem erros a uma frequência de 1 em cada 106 bp
As mutações espontâneas ocorrem a uma taxa 1/106-1/109
Bases moleculares das mutações
As mutações ocorrem por substituições de um par de bases (frameshift) que pode ocorrer por deleção, inserção ou inversão. As mutações ocorrem por substituições de um par de bases (frameshift) que pode ocorrer por deleção, inserção ou inversão.
Emparelhamento incorreto por tautomerização de bases
Em circunstâncias mutacionais, pode ocorrer otautomerismo de cada uma das bases. Assim:
A forma imino de C emparelha com A;
A forma imino de A emparelha com C;
A forma enólica de T emparelha com G;
A forma enólica de G emparelha com T.
Desaminação espontânea de bases
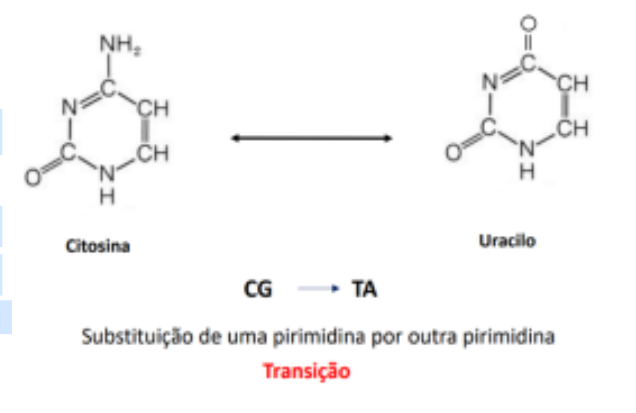
Remoção de um grupo amina) da citosina e colocação de um oxigénio transforma-a num uracilo. Este é característico do RNA e, como tal, existe uma enzima que o remove do DNA e coloca o nucleótido complementar ao que é suposto, que neste caso será uma timina. Assim se transformou um CG em TA.
Oxidação de bases
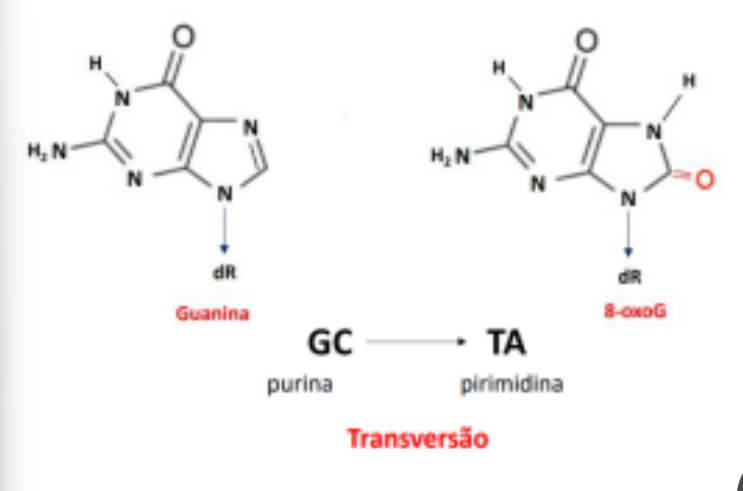
Certas agressões do meio ambiente contra o DNA alteram as bases. São as reações com ROS, ião superóxido ou OH- que lesam o DNA e favorecem a troca de bases, levando à formação do 7,8-dihidro-8-oxoguanina (8-oxoG), que emparelha com a adenina.
Mutações induzidas - Agentes mutagénicos

Agentes análogos das bases → O 5-bromo-uracilo, consoante a sua conformação pode ligar-se tanto à adenina como à guanina. Desta forma podemos trocar, quando ultrapassadas duas gerações, os nucleótidos graças à ação deste composto.
Agentes modificadores do DNA → Temos o exemplo do metil-nitrosoguanidina, um agente alquilante, que adiciona um grupo metil à guanina, fazendo com que esta passe a poder emparelhar com a timina.
Agentes intercalantes → As acridinas ou o brometo de etidium distorcem a molécula de DNA.
Agentes destruidores do DNA → O principal agente destruidor são os raios UV, pois geram dímeros de pirimidinas de duas pirimidinas consecutivas fazendo com que estas não se consigam ligar às adeninas, impedindo a leitura correta da DNA polimerase.
Consequências das mutações pontuais
Mutação silenciosa: a substituição de uma base leva a um codão que codifica para o mesmo aminoácido
Mutação missence (sentido falso): a substituição de uma base leva a um codão que codifica para outro aminoácido. O efeito na função da proteína depende do aminoácido que substitui o normal.
Mutação nonsense: a substituição da base leva a um codão nonsense (um codão stop), levando à terminação prematura da proteina, muitas vezes tornando-a não funcional.
Mutação frameshift: Qualquer adição ou deleção que aconteça num par de bases que não seja múltiplo de 3 - causa uma alteração na grelha de leitura.
Mutações e consequências
As mutações também podem ocorrer em regiões do DNA não codificantes → Podem ocorrer em sequências reguladoras: como operadores ou promotores.
Mutações
Seleção e isolamento de mutantes - Seleção
Seleção
Uma mutação capaz de ser selecionada confere uma vantagem à estirpe mutante em determinadas condições ambientais. (como a resistência a antibióticos)
Seleção e Isolamento de Mutantes - Screening
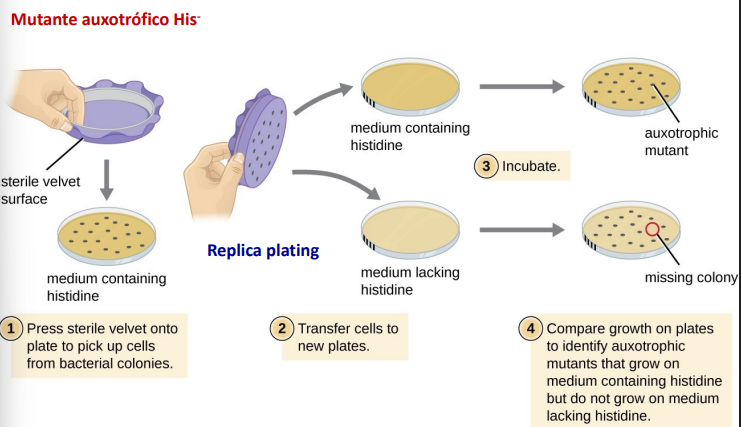
Sistemas de Reparação do DNA
A introdução de erros no DNA pode causar a paragem da replicação - as bactérias desenvolveram mecanismos para reparar esses erros.
• “Proofreading” da DNA Polimerase III
• Reparação por excisão
• Reparação de desemparelhamentos
• Reparação de bases desaminadas
• Reparação por recombinação
• Sistema de reparação SOS
Proofreading da DNA Pol III
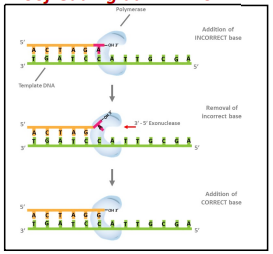
A DNA Polimerase III tem atividade exonucleásica (3’ para 5’), tendo a capacidade de remover o nucleótido que foi introduzido de forma incorreta, reparando assim esse erro.
Reparação por Excisão
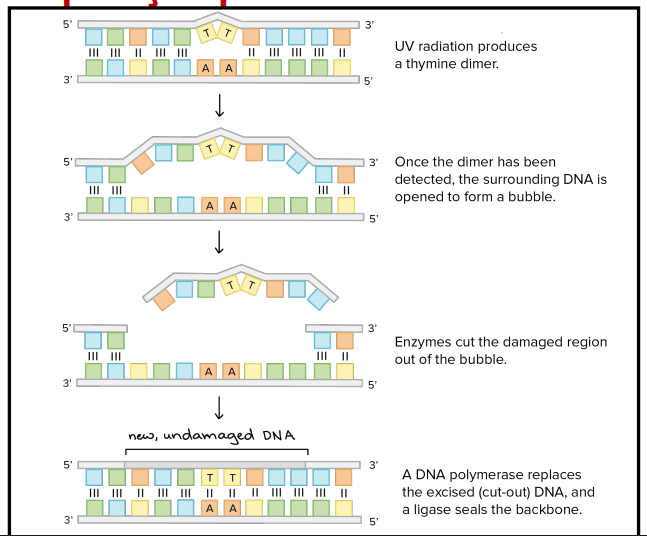
Este tipo de reparação é usado para remover os dímeros de timina. As proteínas uvrB e uvrC vão cortar o DNA no local onde está a lesão e a helicase uvrD vai remover esse pedaço. A DNApolimerase I, associada à reparação, vai fazer a cadeia normal e assim concluir a reparação e criar um DNA saudável.
Mismatch repair system
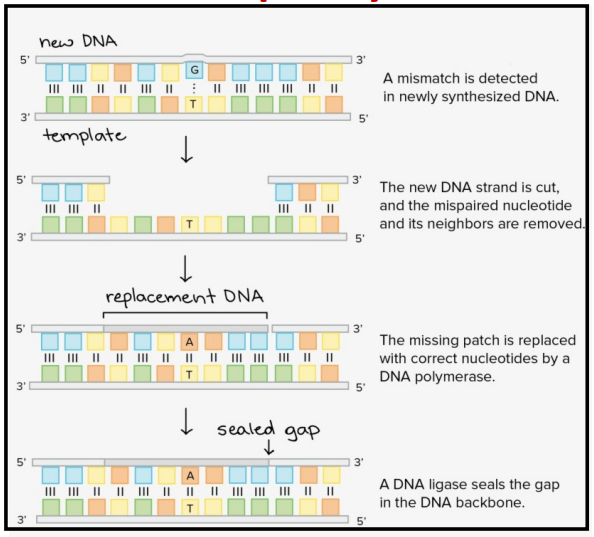
Um nucleótido errado é detetado. A cadeia de DNA é cortada e o nucleótido errado e seus nucleótidos vizinhos são removidos.
A porção da cadeia volta a ser sintetizada pela DNA polimerase de forma correta e, posteriormente, sofre a ação da DNA ligase.
Reparação de Bases Desaminadas
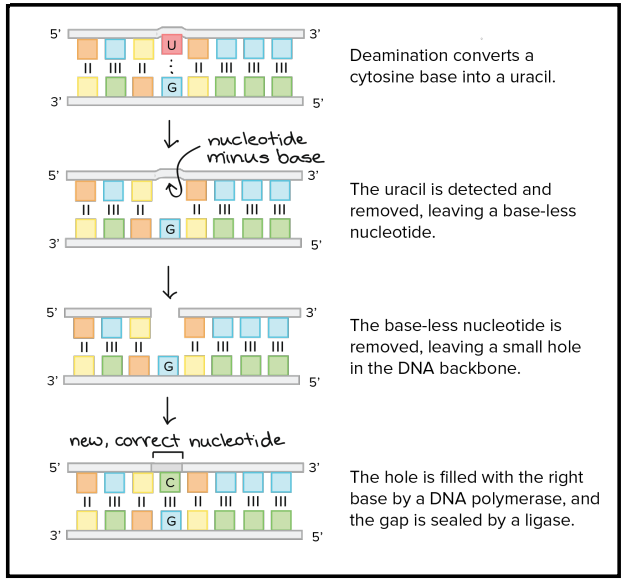
A desaminação converte a citosina num uracilo. Este último é detetado e removido, deixado o nucleótido sem a base azotada.
O nucleótido é removido deixando um “buraco” no molde de DNA, que é preenchido pela base certa com a ajuda da DNA polimerase, e é ligada aos restantes pela DNA ligase.
Sistema de reparação SOS
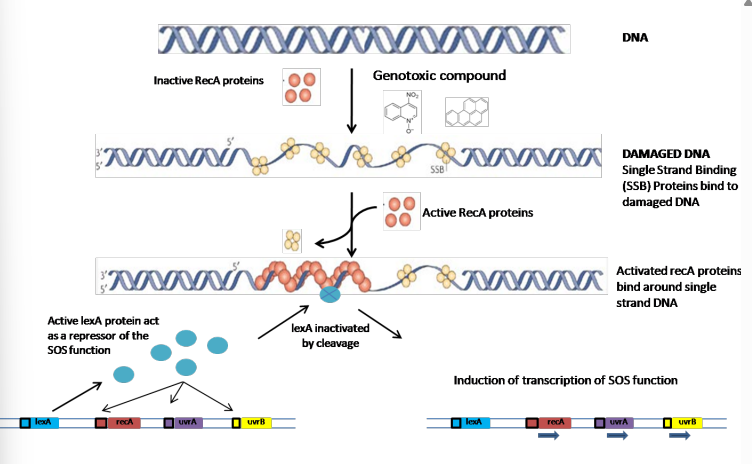
Este processo é especial porque só é usado por procariontes e merece especial atenção. Este é um alarme que a célula tem, sem o qual morre.
O regulão lexA é o regulão SOS, que é um repressor dos genes associados à reparação do DNA e ainda inibe o inibidor da FtsZ, essencial à divisão, ou seja, não inibe a divisão, sendo este um mecanismo de prevenção para que o erro não seja passado a gerações futuras.
Quando há um dímero T=T, a DNA Polimerase pára, ficando uma cadeia simples de DNA exposta, parada, sem ser transcrita. A existência desta cadeia estimula a proteína recA ̧ proteína que cliva a LexA (ativa a proteólise do repressor lexA), permitindo que ocorra expressão dos genes para a resposta SOS. Como tal, vai ser transcrito todo o regulão de reparação do DNA, no qual constam também as uvr, e vai ser inibida a divisão celular.
Ainda assim, este sistema de reparação deixa também no DNA pequenas mutações que podem induzir a evolução.