POLYMORPHISME GENETIQUE
1/24
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No analytics yet
Send a link to your students to track their progress
25 Terms
qu’est-ce que le polymorphisme génétique ?
ce sont les variations de la structure primaire de al séquence d’ADN des génomes entre individus de la même espèce
il y a un polymorphisme tout les 100 à 150 nucléotides = fréquence de variation de génome humain
ces variations sont héréditaires
Qu’est-ce que sont les marqueurs phénotypiques ?
observation de l’expression d’un gène au niveau des protéines (polymorphisme protéique)
=>l’expression observable d’un gène
=>Ils dépendent du gène, mais aussi de l’environnement, du développement, et de l’état de la cellule

Quels sont les marqueurs génotypiques
observation d’un gène au niveau de l’ADN (sa séquence)
=>Ils ne changent pas selon l’environnement : l’ADN reste le même dans toutes les cellules.

A combien de paires de bases correspond le génome humain ? combien y a il de gènes et de gènes codants ?
3,1.109 pdb → un livre de 500 000 pages
22 000 codant pour 26 000
quelle est la proportion de variants communs ? que dire sur les rares ?
1/400
les variants rares sont plus difficiles à observer, ils s’additionnent pour augmenter le polymorphisme.
Il peut etre pathologique
de combien est la variations entre deux individus non apparentés ?
0,1% ce qui représente 3-4Mi de bases
= 0,1% du génome affecté par le polymorphisme
(à 1% de diff, on change d’espèce, mais ce n’est pas du polymorphisme)
où se trouve les polymorphismes le plus souvent ? 2
dans une région non codantes, non fonctionnelle
ou dans une région codante mais silencieuse = pas de mod de la séquence peptidique
qu’est-ce que l’effet défavorable du polymorphisme ?
Il peut être dû à une mutation localisée :
- Dans une région codante avec modification de la signification du codon, ce qui entraîne la modification de la séquence en AA d’une protéine.
- Dans des régions régulatrices de l’expression/transcription d’un gène :
Promoteur.
Enhancer = région stimulatrice de la transcription. o Silencer = région répressive de la transcription.
- Sur un site d’épissage : séquence impliquée dans l’élimination des introns, entraîne une mauvaise maturation de l’ARNm, modifie la séquence protéique traduite.
=>pression de sélection négative
le polymorphisme est maintenu à une fréquence allélique faible
qu’est-ce que l’effet favorable du polymor
C’est encore plus rare que le polymorphisme défavorable. C’est une mutation dans une séquence codante. La mutation confère un avantage sélectif à un organisme dans un environnement défavorable.
Exemple : la résistance à une épidémie. Il y a donc une pression de sélection positive : le polymorphisme est amplifié dans l’espèce donnée pour la population placée dans cet environnement.
qu’est-ce qu’un locus ?
fragment ou séquence d’ADN ± longue qui contient ou non un gène (codant ou non), mais qui a une localisation chromosomique donnée, unique (locus A, locus B). On définit aussi un haplotype (ici A/B2 et A/B1)
=>l’emplacement précis d’un gène sur un K
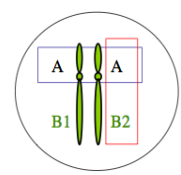
qu’est-ce qu’un allèle QCM
différentes formes moléculaires pour un même locus dans une espèce donnée (allèle B1, B2).
Exemple : le locus A est monomorphe : A, le locus B est biallélique marqueur d’un polymorphisme avec 2 allèles B1 et B2
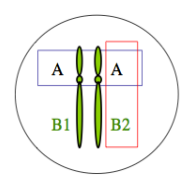
quelle différence entre monomorphe, biallélique et multiallélique ?
L’intérêt est l’étude de la région chromosomique qui porte le locus.
Une maladie héréditaire est un marqueur de polymorphisme génétique.
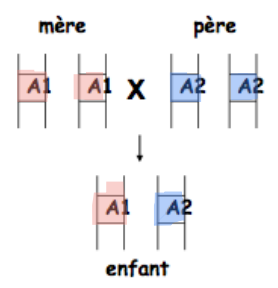
qu’est-ce que le mode de transmission mendélien codominant ?
Les marqueurs génétiques permettent d’étudier un locus polymorphe, ces marqueurs possède un mode de transmission mendélien codominant = pas de système dominant ou récessif.
Dans la transmission de Mendel, le nombre d’allèle d’un locus polymorphe ainsi que la séquence des allèles pour un locus est stable au cours des générations (aussi bien en termes de type de séquence qu’en nombre d’allèles) : donc s’il y a 5 allèles au départ, au fur et à mesure des générations, il y en a toujours 5 avec tous les phénotypes qui s’expriment (ce qui n’est pas le cas des maladies héréditaires récessives).
Il n’y a ni récessivité, ni dominance, tous les génotypes d’un individu peuvent être observés sous forme de phénotypes différents (hétérozygote et homozygote).
=>Chaque individu a un “profil” identifiable.
Type de transmission | Explication |
|---|---|
Mendélienne | 1 allèle vient du père, 1 de la mère |
Codominante | Les deux allèles s’expriment, aucun n’est masqué |
Polymorphe | Plusieurs allèles possibles dans la population |
Stable | Le nombre et la séquence des allèles restent constants au fil des générations |
quelle différence entre la femme et l’homme ?
Pour un locus biallélique :
- Locus autosomique :
o Hétérozygote (A1A2).
o Homozygote (A1A1 ; A2A2).
- Locus sur le chromosome X :
o Hémizygote (A1 ; A2) chez ♂ homme.
o Homozygotie et hétérozygotie chez ♀ femelle.
Étude familiale de la répartition des allèles au cours des générations
On retrouve l’association d’un allèle, sa transmission et la présence/absence de la maladie. Cela dépend du mode :
si codominance ou récessivité, cela complique les études familiales
On utilise la migration électrophorétique (plus le fragment est bas, plus le fragment est petit).
On suit la co-ségrégation des allèles. Ici, il y a de fortes chances pour que le marqueur soit à proximité du gène responsable de la maladie.
p5