CM2: Révisions - Transcription/traduction
1/6
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No analytics yet
Send a link to your students to track their progress
7 Terms
🔹 GÈNES DE E. coli ET ORGANISATION DU GÉNOME 🔴 Questions
1. Que peut-on dire des gènes de Escherichia coli à partir du tableau comparatif ?
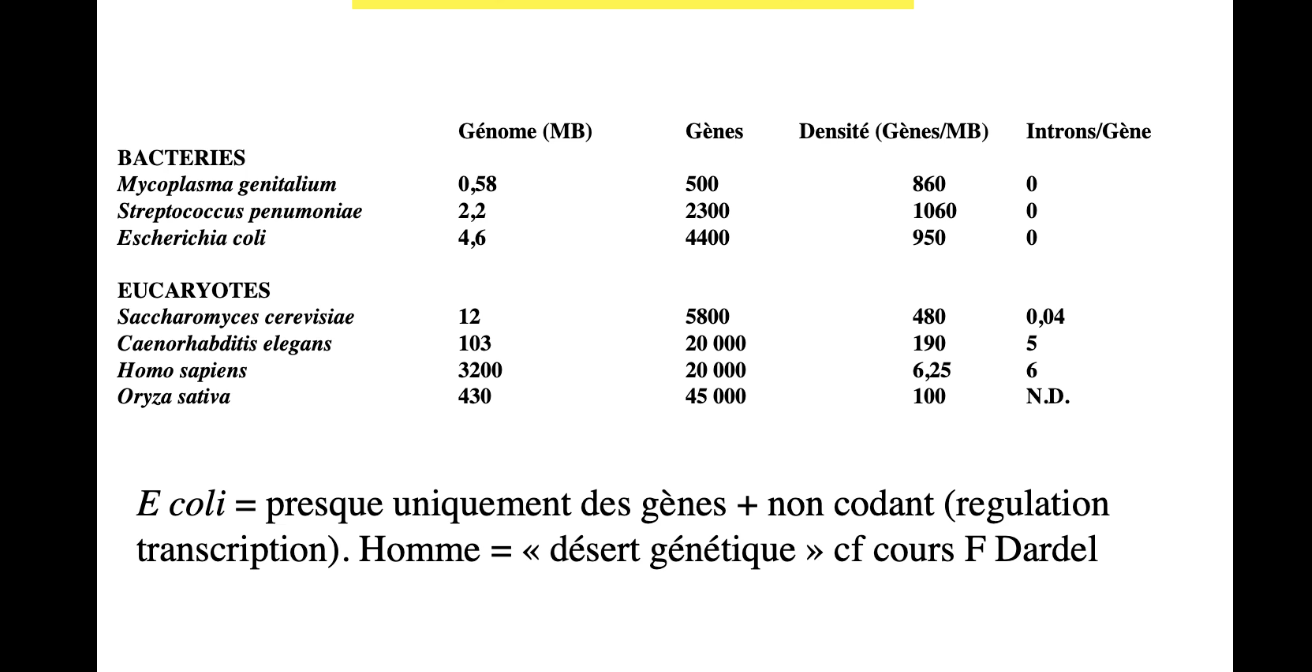
🔴 Réponse 1. Que peut-on dire des gènes de Escherichia coli ?
Quoi :
Le génome de E. coli est compact, dense en gènes et quasi entièrement codant.
Données clés (image) :
Taille du génome ≈ 4,6 Mb
Nombre de gènes ≈ 4400
Densité ≈ 950 gènes/Mb
🔴 0 intron par gène
Comment :
Les gènes sont très rapprochés
Peu de régions intergéniques
ARN non codants surtout impliqués dans la régulation transcriptionnelle
Pourquoi :
→ Optimisation maximale de l’espace génomique
→ Transcription et traduction rapides et efficaces
🟢 Comparaison implicite :
Chez l’Homme : faible densité génique → « désert génétique »
🔹 PROMOTEUR BACTÉRIEN 🔴 Questions
2. Qu’est-ce qu’un promoteur bactérien ?
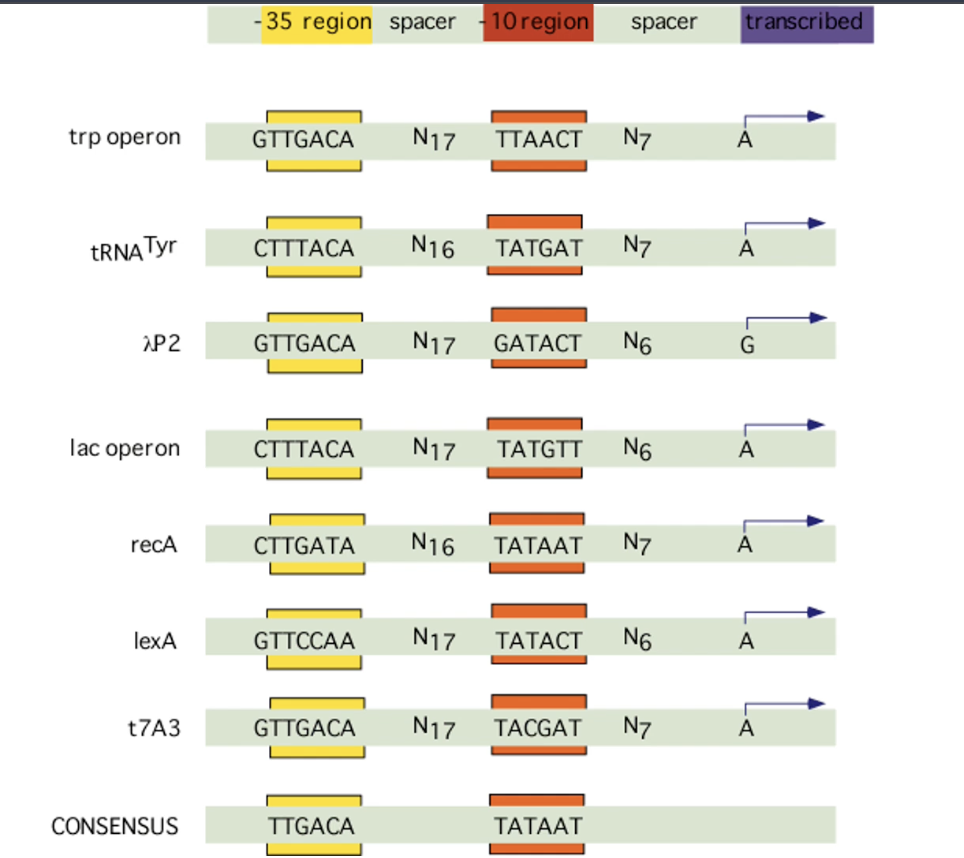
🔴 Réponse 2. Qu’est-ce qu’un promoteur bactérien ?
Quoi :
Séquence d’ADN en amont d’un gène, reconnue par l’ARN polymérase pour initier la transcription.
Où (organisation canonique) :
🔴 Région −35 : consensus TTGACA
🔴 Région −10 (boîte Pribnow) : consensus TATAAT
Espacement ≈ 17 nucléotides
+1 = premier nucléotide transcrit
Comment :
Le facteur σ reconnaît −35 et −10
La région −10 riche en AT facilite l’ouverture de l’ADN
Pourquoi :
→ Détermine :
le site de démarrage
l’efficacité de la transcription
la spécificité du gène exprimé
🔹 TRANSCRIPTION CHEZ LES PROCARYOTES 🔴 Questions
3. Quelles sont les trois étapes de la transcription chez les procaryotes ?
🔴 Réponse 3. Quelles sont les trois étapes de la transcription chez les procaryotes ?
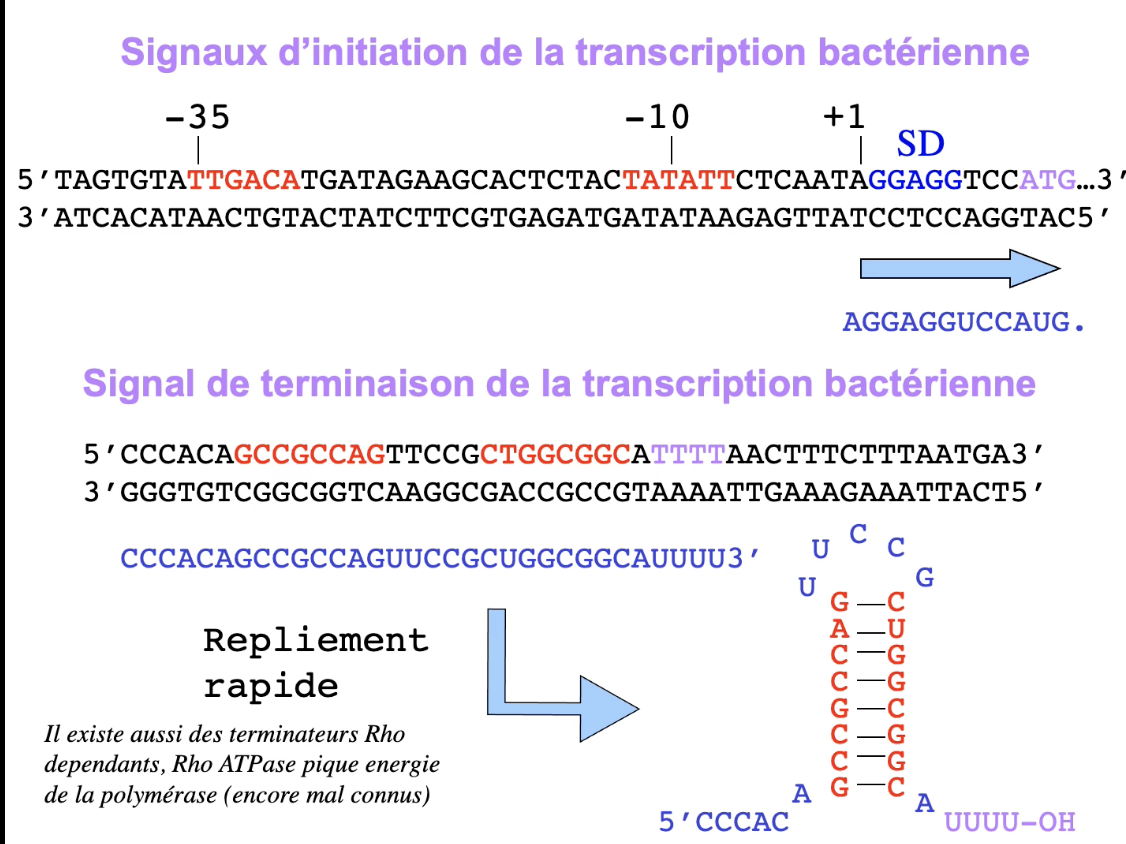
🔴 1. Initiation
Quoi :
Fixation de l’ARN polymérase sur le promoteur
Comment :
Holoenzyme = α₂ββ’σ
σ reconnaît −35 / −10
Ouverture locale de l’ADN (~15 pb puis jusqu’à ~80 pb)
Synthèse des premiers nucléotides
🔴 Quand l’ARN atteint ~12 nt → σ se détache
Pourquoi :
→ Assure un démarrage précis et contrôlé
🔴 2. Élongation
Quoi :
Synthèse de l’ARN 5’ → 3’
Comment :
β lie les ribonucléotides
β’ lit le brin matrice
ADN se referme derrière la polymérase
ARN expulsé au fur et à mesure
Pourquoi :
→ Production fidèle du transcrit
🔴 3. Terminaison
Quoi :
Arrêt de la transcription et libération de l’ARN
Comment :
🔴 ρ-indépendante :
boucle ARN GC riche + queue poly-U
repliement rapide → dissociation
🟢 ρ-dépendante :
protéine Rho (ATPase) déstabilise le complexe
Pourquoi :
→ Empêche la transcription au-delà du gène
🔹 ARNt SYNTHÉTASE ET CHARGE DES ARNt 🔴 Questions
4. Comment fonctionne une aminoacyl-tRNA synthétase ?
5. Comment se fait la charge d’un ARNt ?
🔴 Réponses 4. Comment fonctionne une aminoacyl-tRNA synthétase ?
Quoi :
Enzyme spécifique qui associe le bon acide aminé au bon ARNt.
Spécificité :
🔴 1 synthétase / acide aminé
Reconnaît :
chaîne latérale de l’aa
anticodon + structure de l’ARNt
Pourquoi :
→ Garantit la fidélité du code génétique
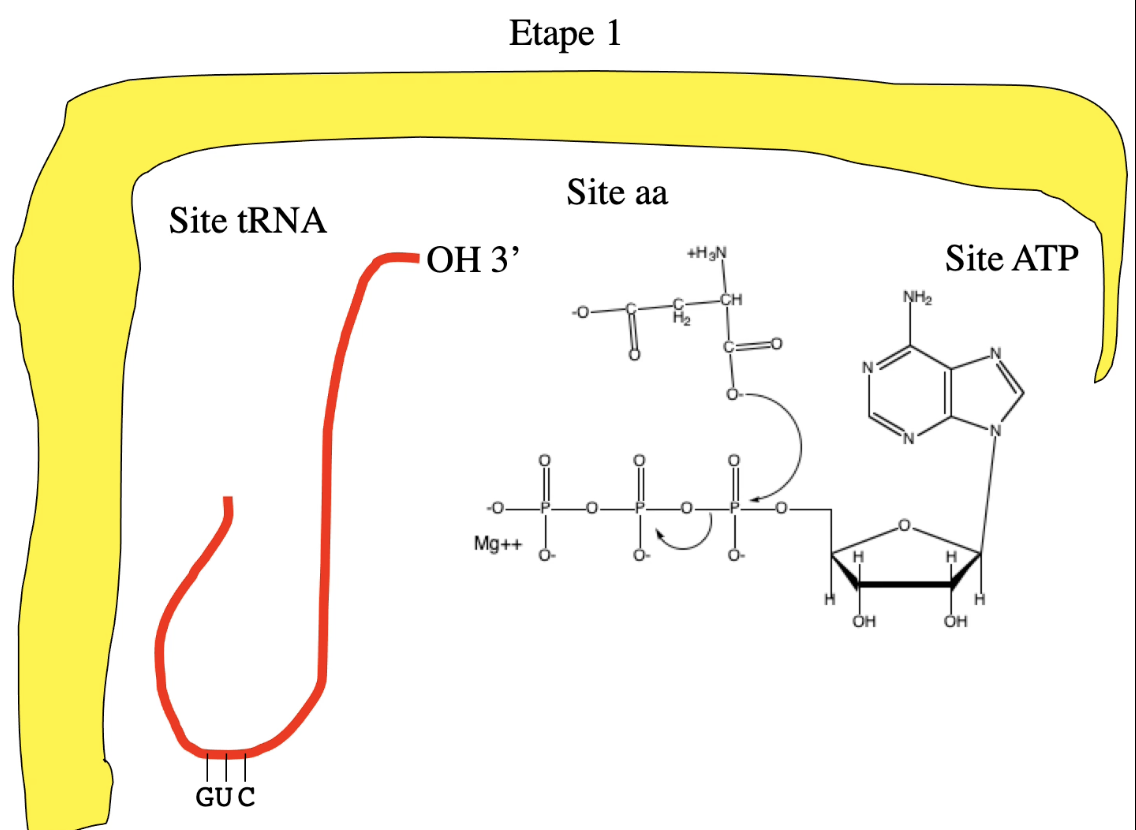
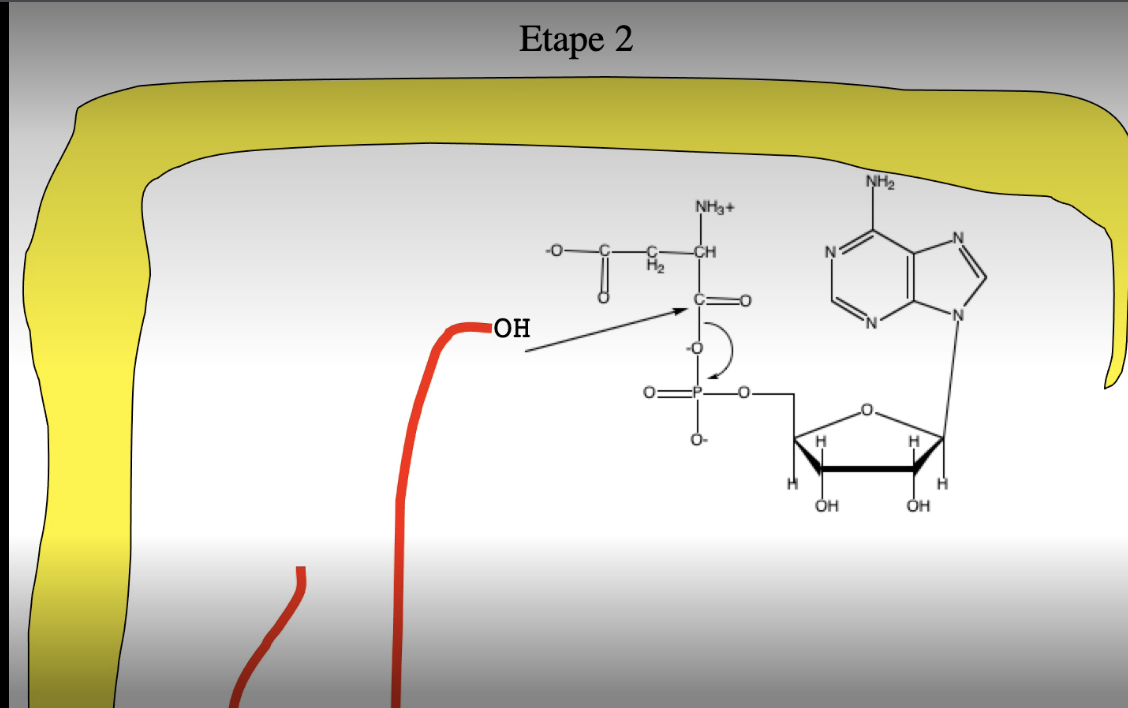
5. Comment se fait la charge d’un ARNt ? (3 étapes)
🔴 Étape 1 : activation de l’acide aminé
Acide aminé + ATP → aminoacyl-AMP
Libération PPi
Mg²⁺ indispensable
🔴 Étape 2 : transfert sur l’ARNt
L’aa est transféré sur le OH en 3’ de l’ARNt
Formation d’une liaison ester à haute énergie
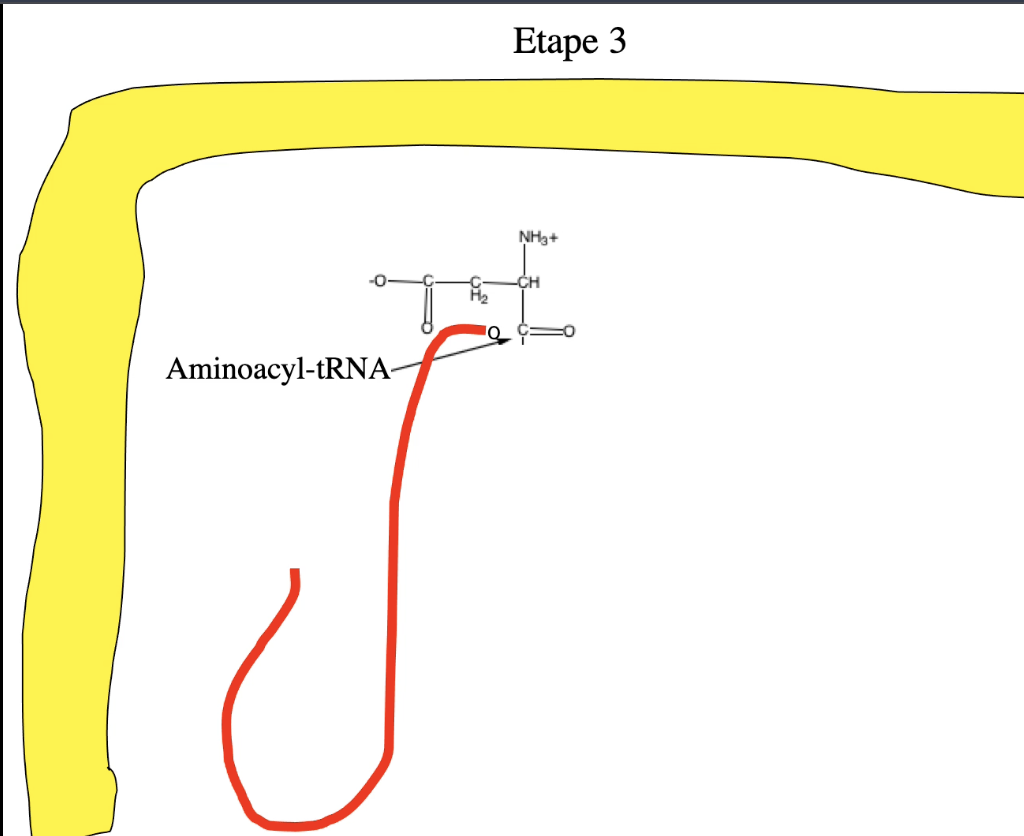
🔴 Étape 3 : ARNt chargé
Produit final : aminoacyl-tRNA
Directement utilisable par le ribosome
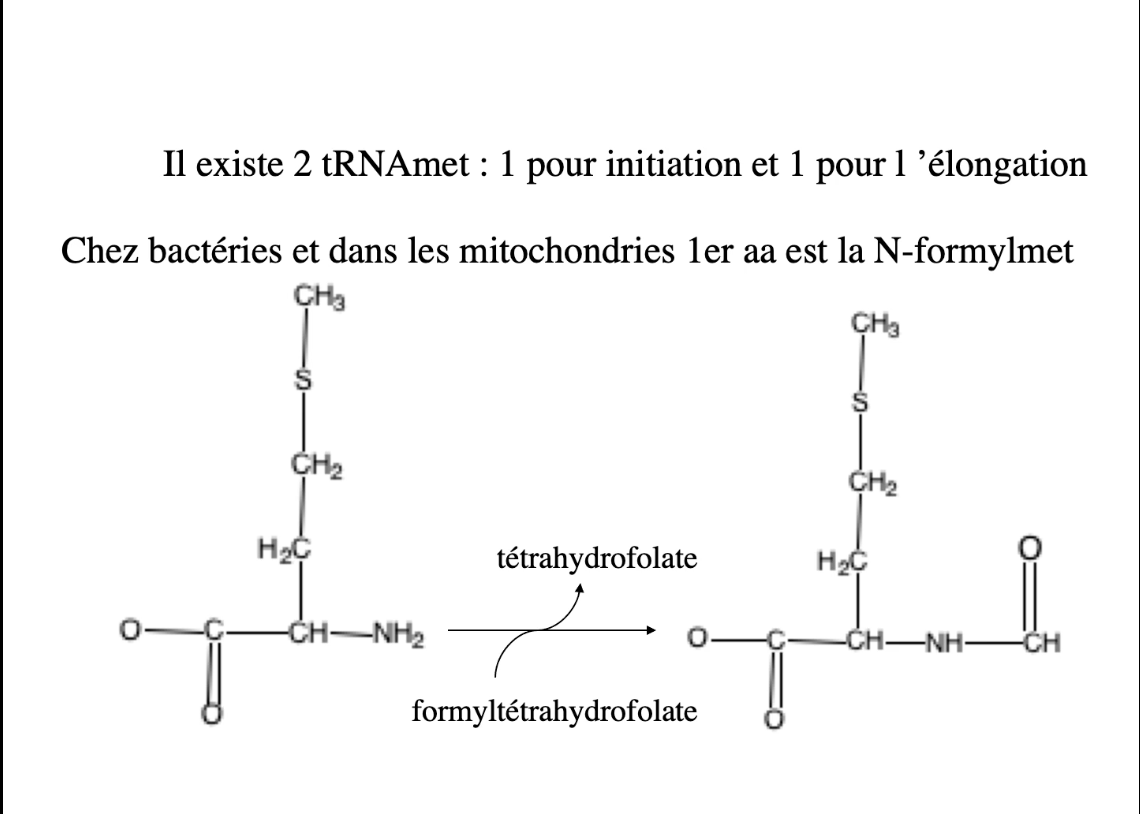
🟢 Cas particulier (image) :
Deux ARNtMet :
🔴 initiateur
🟢 élongateur
Chez bactéries (et mitochondries) :
premier aa = N-formyl-méthionine (fMet)
formylation via formyl-tétrahydrofolate
🔹 TRADUCTION CHEZ LES PROCARYOTES 🔴 Questions
1. Quelles sont les grandes caractéristiques de la traduction chez les procaryotes ?
2. Comment se déroule l’initiation de la traduction chez les procaryotes ?
3. Quelles sont les étapes de l’élongation chez les procaryotes ?
4. Comment se fait la terminaison de la traduction chez les procaryotes ?
5. Qu’est-ce que le couplage transcription–traduction chez les procaryotes ?
🔴 Réponses 1. Quelles sont les grandes caractéristiques de la traduction chez les procaryotes ?
Quoi :
Traduction rapide, cytoplasmique
ARNm polycistronique
Pas de coiffe 5’, pas de poly(A)
ARNm très instable (t½ < 2 s chez E. coli)
Où / Quand :
Dans le cytoplasme
Simultanée à la transcription
Pourquoi :
→ Réponse extrêmement rapide aux conditions environnementales
2. Comment se déroule l’initiation de la traduction chez les procaryotes ?
Quoi :
Assemblage du ribosome sur l’ARNm au codon start.
Comment (ordre exact) :
La sous-unité 30S se fixe sur la séquence Shine-Dalgarno
IF1 et IF3 empêchent l’association prématurée de la 50S
Le tRNA initiateur fMet-tRNAᶠᴹᵉᵗ arrive avec IF2-GTP
Appariement anticodon–AUG au site P
Association de la 50S
Hydrolyse du GTP → départ IF1, IF2, IF3
Pourquoi :
→ Garantit un démarrage précis et orienté de la traduction
🔴 Point clé :
Le premier acide aminé est la N-formyl-méthionine
3. Quelles sont les étapes de l’élongation chez les procaryotes ?
Quoi :
Allongement de la chaîne polypeptidique.
Comment (cycle répété) :
aa-tRNA entre au site A (protégé par EF-Tu-GTP)
Appariement codon/anticodon
Peptidyl-transférase (ARNr 23S) forme la liaison peptidique
EF-G-GTP provoque la translocation
tRNA A → P
tRNA P → E
ribosome avance d’un codon
Pourquoi :
→ Synthèse directionnelle N-ter → C-ter
4. Comment se fait la terminaison de la traduction chez les procaryotes ?
Quoi :
Arrêt de la traduction au codon stop.
Comment :
Codon stop (UAA, UAG, UGA) au site A
Fixation de RF1 ou RF2
Ajout d’H₂O (et non d’un aa)
Libération du polypeptide
RRF + EF-G dissocient le ribosome
Pourquoi :
→ Libération correcte de la protéine et recyclage du ribosome
5. Qu’est-ce que le couplage transcription–traduction chez les procaryotes ?
Quoi :
La traduction commence avant la fin de la transcription.
Comment :
Ribosomes se fixent sur l’ARNm naissant
Plusieurs ribosomes traduisent en parallèle (polysomes)
Pourquoi :
→ Gain de temps majeur
→ Exploitation d’ARNm très instables
🔹 TRADUCTION CHEZ LES EUCARYOTES 🔴 Questions
6. Quelles sont les grandes caractéristiques de la traduction chez les eucaryotes ?
7. Comment se déroule l’initiation de la traduction chez les eucaryotes ?
8. Quelles sont les différences majeures d’élongation et de terminaison avec les procaryotes ?
🔴 Réponses 6. Quelles sont les grandes caractéristiques de la traduction chez les eucaryotes ?
Quoi :
Traduction plus lente
ARNm monocistronique
Présence :
🔴 coiffe 5’ (m⁷G)
🔴 queue poly(A) 3’
🔴 épissage préalable
Où / Quand :
Traduction dans le cytoplasme
Transcription dans le noyau → pas de couplage
Pourquoi :
→ Régulation fine de l’expression génique
7. Comment se déroule l’initiation de la traduction chez les eucaryotes ?
Quoi :
Reconnaissance de la coiffe et balayage jusqu’au codon start.
Comment :
La petite sous-unité 40S + facteurs d’initiation se fixent sur la coiffe 5’
Le ribosome scanne l’ARNm
Reconnaissance du codon AUG dans le contexte de Kozak
Arrivée de la grande sous-unité 60S
Pourquoi :
→ Assure la sélection du bon AUG
🔴 Point clé :
Premier aa = méthionine non formylée
8. Quelles sont les différences majeures d’élongation et de terminaison avec les procaryotes ?
Élongation :
Mécanisme global similaire
Facteurs différents (eEF1A, eEF2)
Terminaison :
Facteurs eRF
ARNm plus stables
Ribosomes recyclés indépendamment
Pourquoi :
→ Traduction plus contrôlée, moins rapide mais plus précise
Procaryotes | Eucaryotes | |
|---|---|---|
ARNm | ||
Coiffe / polyA | ||
Initiation | ||
1er aa | ||
Couplage transcription-traduction | ||
Vitesse |
Procaryotes | Eucaryotes | |
|---|---|---|
ARNm | Polycistronique | Monocistronique |
Coiffe / polyA | ❌ | ✅ |
Initiation | Shine-Dalgarno | Kozak |
1er aa | fMet | Met |
Couplage transcription-traduction | ✅ | ❌ |
Vitesse | Très rapide | Plus lente |