molekularna izpitna pt1
1/41
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No analytics yet
Send a link to your students to track their progress
42 Terms
razlika med bakterijsko topoizomerazo 1 in 2
1: cepi verigo 1x, zvija v + smer (razvija dodatna zvitja), ne rabi ATP
2 (DNA giraza): cepi verigo 2x, zvija v - smer (uvede dodatna zvitja), rabi ATP.
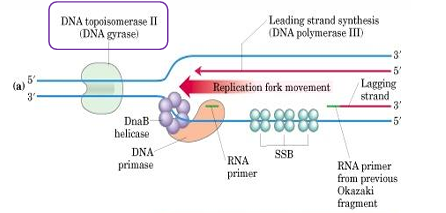
na sliki označi vodilno verigo, elemente, ki sodelujejo pri podvajanju DNA (prokarionti)
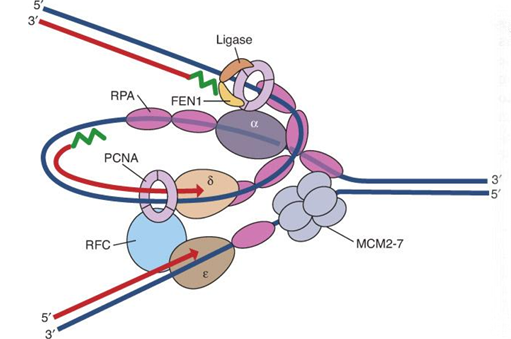
na sliki označi vodilno verigo, elemente, ki sodelujejo pri podvajanju DNA (evkarionti)
transpozon
je genetski element, ki se lahko premakne iz enega mesta v genomu na drugo. lahko prinese mutacije, lahko pa tudi antibiotik rezistenco itd. lahko inhibira gene če se ustavi notr
Bakterijski transpozoni: insercijska zaporedja (IS), na koncih imajo IR- inverzne ponovitve, vmes imajo gen za transpozazo. Sestavljeni transpozoni: ko se IS vrinejo na en ali oba konca enega ali več genov.
Evkariontski:
-tip I - retrotranspozoni, vsebujejo RNA intermediat za izvedbo transpozicije. Retrotranspozone razvrščamo v dve skupini: LTR in ne-LTR (LTR = dolge končne ponovitve)
-tip II - delujejo po načelu »samo DNA«, podobni bakterijskim.
razlika med spliceosomom in samoizrezovalnim intronom
Spliceosom: RNA-proteinski kompleks (iz snRNP-jev in prot.), rabi ATP, reže evk mRNA večinoma
Samoizrezovalni int: ne rabijo proteinov - RNA kot ribocim, ne rabijo ATP, režejo tudi v organelih, prok
skupne značilnosti intronov, evolucijska funkcija
Ne kodirajo genov, najdemo jih v DNA molekuli, imajo jih pretežno evkarionti, ponavadi so daljši od eksonov, vsebujejo 5’ in 3’ sekvence za splicing in razvejitveni nukleotid in polipirimidinski trakt
evolucijska funkcija:
alternativno spajanje: nastane iz 1 pre-mRNA več različnih kombinacij eksonov in posledično več različnih polipeptidov → evolucija genov
podpirajo RNA World hipotezo: samoizrezovalni introni mRNA ima katalitično funkcijo) so dokaz, da je verjetno na začetku razvoja življenja RNA mol nosila gensko info in tudi katalizirala biološke procese
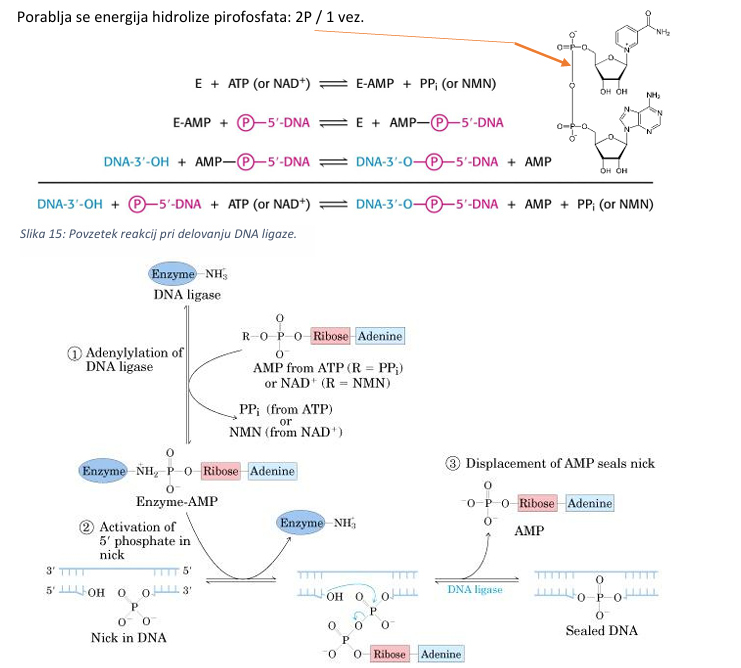
3 stopnje mehanizma ligacije
1. adenilacija - aktivacija encima (AMP vezan na Lys),
2. prenos AMP na fosfatno sk. na 5’-koncu (DNA-adenilatni kompleks) → aktivacija 5' fosfata,
3. nukleofilni napad 3’-OH skupine na aktivirani fosfor → AMP se odcepi, DNA se zlepi
mehanizem vezave aminokisline na tRNA
reakcijo kataliziraaminoacil-tRNA sintetaza:
aktivacija aminokisline (adenilacija) - v aktivno mesto se poleg ak veže ATP → aminoacil-AMP + PPi
tvori se kovalentna vez med ak in tRNA (3’ koncem, CCA zaporedje) - aminokislina je estrsko vezana na 3' (razred II) ali 2' (razred I) -OH skupini končnega adenozina
na aminoacil-tRNA se veže elongacijski faktor Tu (EFTu) s kofaktorjem GTP - sodeluje pri vnosu v ribosom in prepreči spontano hidrolizo vezi med tRNA in aminokislino.
terminacija translacije
RF-ji prepoznajo stop kodone na mRNA (UAA, UAG, UGA) v mestu A na ribosomu in se vežejo gor (1, 2, 3 pri pro, evk brez 2) - konformacijska sprememba akt. mesta - postane hidrolaza
polipeptid se hidrolitično odcepi iz tRNA v P mestu
veže se RF3 in GTP, RF1 se odcepi
hidroliza GTP v GDP → RF3 in tRNA se odcepi, vežejo se RRF (rybosome recycling factor) in EF-G → podenote oddisocirajo
psevdouracil, ribotimidin se nahajata na verigi----------, ki prenaša-----------------na aktivno mesto------------ in tvori------------------vezi.
tRNA, aminokislino, ribosoma, peptidne
opiši mehanizem odstranjevanja baze in encime, ki pri tem sodelujejo (BER)
→Popravljanje z odcepom baze (BER):
1. odcep baze in razcep verige (glikozilaza ali glikozilaza/liaza in endonukleaza) - hidroliično odstrani bazo, dobimo abazično mesto
2. zamenjava nukleotida in ligacija:
• zamenjava segmenta - DNApol delta/epsilon sint nov segment, ki izpodrine starega, flap endonukleaza ga odstrani, DNA ligaza poveže segmente
• zamenjava enega nukleotida - DNApol beta sint en nukleotid, ligaza poveže
→Popravljanje neujemanja (MMR):
Na nemetilirani (hčerinski) verigi endonukleaza zareže na obeh straneh mutacije, eksonukleaza pa odstrani vmesni del.
DNA-polimeraza in DNA-ligaza zapolnita vrzel.
→Popravljanje z izcepom nukleotida (NER):
Napako prepoznajo protini 🡪 denaturacijski mehurček; uvedba dveh zarez, odcep segmenta in popravljanje z DNA-polimerazo in DNA-ligazo
licenčni faktorji
skrbijo, da se DNA pri evk vsake cel samo enkrat na cel. cikel podvoji (S faza)
ORC (ori recognition complex) - iniciator, prvi se veže se na ori
Cdc6 (cell division cycle protein) - veže se na ORC v M7zgodnji G1 fazi, ima ATPazno aktivnost, po iniciaciji gre dol
MCM2-7 (helikaza) in Cdt1 also del predreplikacijskega sistema
celoten mehanizem sproži replikacijo, nato se odstrani - ne more priti do re-iniciacije do naslednjega cel cikla
3 motivi (oblike) , ki omogočajo vezavo na DNA
HTH - heliks-turn-helix, za gene, ki sodelujejo pri razvoju, 2 alfa heliiksa in kratek zavoj med njima - vmes se usede DNA
cinkovi prsti - v jedrnih receptorjih (TF), rabijo ligand, 2 tipa (tipI - v odsotnosti liganda v citosolu, tipII - v odsotnosti liganda v jedru vezani na DNa ampak neaktivni), notr Zn vezan na 4 Cys
levcinska zadrga - proteini, ki regulirajo cel. delitev, bazična domena heliks, ki sede v veliki žleb
nukleosom
evkarionti, kromatin navit okrog oktamera histona (1,65x → 170-240 bp, širok 110A, visok 55A). vsebuje 8 prot. kompleksov: 2x H2A, 2X H2B, 2X H3, 2X H4 plus H1 histon vezan na DNA gor - interakcije med nukleosomom in H1 → kromatosom. Dosti + nabitih ak, da vežejo DNA , ki je negativno dodatno zvita. Histoni se dodatno pakirajo v kromatinske filamente (heterokromatin)
mehanizem homologne rekombinacije
prokarionti:
endonukleaza prereže eno od molekul DNA
RecBCD (B in D helikaze, C nukleaza) kompleks se veže na prosti konec, ki nastane zaradi preloma verig in razgrajuje DNA dokler ne pride do mesta chi (crossover hotspot instigator site)
RecD aktivnost (5’→3’) se upočasni, RecB (3’→5’) pa pospeši, hidrolizira se samo še 5’, dobimo izpostavljen 3’ konec, Rec BCD oddisocira
na 3’ se veže/ovije RecA - poišče dsDNA in izpodrine starševsko verigo → D zanka, nato oddisocira
nastane Hollidayevo križišče - križna struktura DNA, RuvA in RuvB katalizirata premik HJ naprej po DNA (branch migration)
RuvC je resolvaza - rezcepi križišče, ligaza poveže proste konce
evkarionti:
namesto RecBCD so MRN kompleks (nukleaza), CtlP (5’→3’ degradacija), Bloom helikaza + DNA2 nukleaza ali EXO1 za 3’ konec exposat
Rad51 namesto RecA, sodeluje tudi BRCA2
dokaz nastanka okazakijevih fragmentov
E.coli so gojili v timidinu s tritijem, vzeli vzorce po različnih časih, iz grafa radioaktivnosti v odvisnosti od potovanja fragmentov določili razmerje daljših in krajših fragmentov ob določenem času. Zgledalo je, kot da je vsa DNA najprej v manjših frag. ki se zlepijo skupaj (zaradi popravljalnega mehanizma je zgledalo, da so obe verige tako)
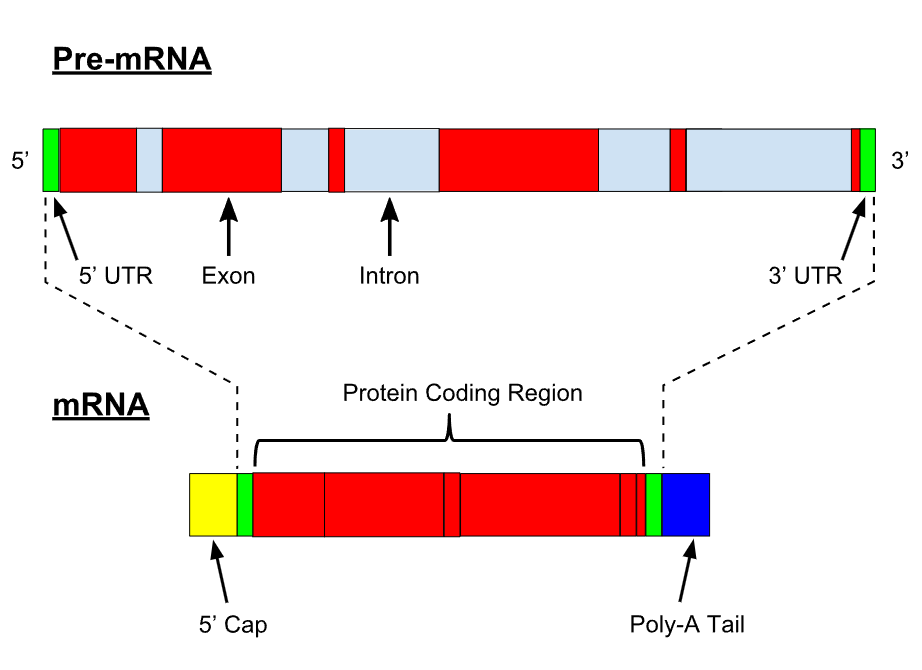
nariši primarni transkript
3 kovalentne modifikacije
formacija 5’-kape: kotranskripcijsko (ko je 20-30 nukleotidov dolg), doda se metilirana gvaninska kapa - ščiti RNA pred RNazami, pospeši translacijo, vpliva na procesiranje mRNA
acetilacija (npr. histonov): ponavadi na Lys → spremeni se neto naboj histona, DNA se odvija dol in je tako dostopna za transkripcijo - izražanje genov. Lysine acetyltransferases katalizirajo
metilacija (npr. DNA): ponavadi C-ji → transkripcijski faktorji se ne morejo vezat na DNA → utišanje genov. DNA methyltransferases katalizirajo (S-adenosylmethionine (SAM) je donor metilne skupine)
O-glikozilacija, N-glikozilacija
interferenčna RNA- 2 možna zaključka
inhibira sintezo proteinov (mikro RNA): se veže na mRNA - ji je komplementarna → ne morejo se vezati mol. za translacijo
utišanje genov (siRNA): veže se na mRNA again, ne pride do translacije, gen se ne izraža, lahko tudi uniči mRNA, ker jo cel s siRNA prepozna kot tujo
značilnosti bakt. promotorja
prinbow-ova škatla (-10) -TATAAT
-35 škatla - TTGACA
sigma faktor prepozna promotorsko zaporedje (škatle) → gor se veže RNApol → konformacijska sprememba DNA (se ovije okrog transkrippcijske mašinerije, -12 do +2 se odvije)
UP element - navzgor od -35 škatle, pospeši biosintezo RNA
v čem se telomeraza razlikuje od polimeraze
polimeraza: DNA matrica za sint DNA, sinteza cele DNA, rabi RNA primer, lahko kontrolno branje in popravljanje napak, vse celice
telomeraza: RNA kot matrica, deluje le na 3’ koncih kromosomov (preprečuje njihovo krajšanje), podaljša obstoječi 3’ previs(ne rabi primerja), dodaja ponovitve sekvenc, ni kontrolnega branja, imajo jo le nekatere celice (zarodne, rakave nekatere), podaljša le eno verigo (drugo polimeraza)
transkripcija RNA- virusov
(+) RNA: genomska RNA lahko deluje kot mRNA, lahko direkt sint. prot - tudi za replikazo da se genom podvaja lahko (prek RNA (-))
(-) RNA: iz nje se prepiše mRNA - rabijo met encime za to že sint. v virusu (replikaza, encimi za 5’ kapo in 3’ rep)
dsRNA: se prepiše z replikazo v mRNA
za podvajanje majo od RNA odvisno RNA polimerazo (replikazo)
retrovirusi: reverzna transkriptaza prepiše RNA v DNA, celica nato prepiše virusno DNA v v mRNA in novo genomsko RNA
posledice tavtomerije
nepravilna parjenja baz - npr. na A amino skupina tavtomerizira v imino in A se spari s C; lahko tudi nastanejo analogi baz ki se parijo z bazami
tranzicije - menjava purina s purinom ali pirimidina s pirimidinom
mutacije: pri podvojevanju dna je ena baza napačna (npr C se je paril z A namesto T) → se poveže napačna baza s sebi pravo → nastane drug bp kot bi moral biti
vpliv araC na transkripcijo
araC kodira za AraC protein, ki v odsotnosti arabinoze deluje kot represor (vezan na I1 in O2 → nastane zanka na DNA, RNA polimeraza se nemore vezat. Ko je arabinoza prisotna se veže na AraC in ga konf. spremeni v aktivator (veže se na I1 in I2), RNApol se lahko veže in prepiše gene za metabolizem arabinoze (araBAD)
protoonkogen, onkogen
protoonkogen: kodira proteine esencialne za normalno cel rast in delitev
onkogen: nastane ko protoonkogen mutira → sint. bolj aktivne oblike prot ali pa preveč prot. → povzroči rakave spremembe v celici
ponovitve trinukleotidov
so nestabilni, lahko povzročajo bolezni/mutacije - med replikacijo se lahko oblikujejo v zanko → DNApol zdrsne
ponovitev na hčerinski verigi - zanka postane del nove verige - več ponovitev, ta se podaljša
matrična veriga- zanka se ne prepiše, nova veriga je krajša, manjkajo nukleotidi
antibiotiki- možnost vpliva na transkripcijo
Streptomicin (aminoglikozidi) 🡪 inhibira iniciacijo in povzroči napačno branje mRNA
Tetraciklin 🡪 veže se na 30S podenoto ribosoma in inhibira vezavo aminoacil-tRNA
Kloramfenikol 🡪 inhibira peptidilno transferazno aktivnost 50S podenote (vsi prokarionti)
Cikloheksimid 🡪 inhibira peptidilno transferazno aktivnost 60S podenote (veže na aktivno mesto) (evkarionti)
Eritromicin 🡪 veže na 50S podenoto in inhibira translokacijo (prokarionti)
Puromicin 🡪 povzroči prezgodnjo terminacijo. Deluje kot analog aminoacil-tRNA (prokarionti in evkarionti)
razlika med DNA polimerazo 1 in 3
1: odstranjuje RNA primerje, in namesto primerja sint. DNA, 3’→5’ in 5’→3’ eksonukleaza, počasnejša, ne rabi ATP, občutljiva na visoko ionsko moč, p-kloromerkuribenzoat
3: sintetizira verigo DNA, 3’→5’ eksonukleaza, hitrejša, rabi ATP za naložit drsno vponko
vloga represorja pri arabinoznem operonu
represor je protein AraC - dimer, v odsotnosti arabinoze je vezan na I1 in O2 mesti na promotorju → nastane zanka na DNA, RNApol se ne more vezati, geni se ne prepišejo. Če je arabinoza prisotna, se veže na AraC in ga spremeni iz represorja v aktivator.
kaj je tumorski supresor
gen, ki kodira proteine, ki upočasnijo replikacijo DNA in delitev celic ter tako poskrbijo, da se cel ne nekontrolirano delijo in rastejo.
razlika med replikacijskimi vilicami in repl. mehurčkom
mehurček: nastane na DNA - začne se na mestu ori (lahko jih je več) - odvita DNA, helikaza jo širi v obe smeri, dokler se mehurčki ne srečajo
vilice: aktivno mesto na koncu mehurčka, kjer se DNA razvija in nova DNA sintetizira (1 mehurček ima 2 vilice)
kaj nardi deaminacija baze
deaminirana baza se lahko poveže z napačno → tranzicijske mutacije
mutacije pri replikaciji - DNApol se ne more vezat gor
lahko premakne bralni okvir
kolk procentov ponovitev ima v genomu človek in kakšnih zaporedij ima največ
60% genoma so ponovitve, največ je intronov (90%), največ ponovitev je ALU
kaj je resolvosom
proteinski komnpleks, ki sodeluje pri popravljanju DNA - razreši Hollidayevo križišče.
(pri E.coli) je iz RuvABC: RuvA veže RuvB na DNA, B je helikaza, ki premika HJ po DNA, RuvC je nukleaza, ki cepi HJ.
opis samoizreza introna
-OH skupina prostega G napade fosfat na 5’ koncu introna
na 3’ koncu prvega eksona je zdaj prosta -OH skupina → napade fosfat na 5’koncu drugega eksona
dobimo odcepljeni intron in spojena eksona
3’ -OH skupina na intronu napade razvejitveno mesto introna → dobimo ciklični intron + 5’-končno zaporedje introna
lahko pa ne poteka prek prostega G, ampak prek vgrajenega A
mehanizem popravljanja napačno vgrajene baze pri DNA polimerazi 1
konformacijska sprememba encima - prsti encima ne oblikujejo žepa za bp, če je ta napačen
3’ konec z napačnim nukleotidom je premaknjen iz aktivnega mesta polimeraze (D3) v 3’→5’ eksonukleazno aktivno mesto (D2), pri tem se prekine več H vezi
poteče nukleofilni napad na 5’-fosfat napačnega nukleotida, odcepi se tudi par ostalih nukleotidov pred njim
3’-OH konec nato gre nazaj v aktivno mesto polimeraze, sinteza se nadaljuje
princip delovanja telomeraze
je ribonukleoprotein, sint. DNA po RNA matrici (reverzna transkriptaza)
po replikaciji DNA nastanejo topi konci, tega ne more daljšati → deluje 5’→3’ eksonukleaza da dobimo 3’ previs
telomerazna RNA matrika se veže na 3’ previs, telomeraza po njej sintetizira DNA - podaljša 3’ previs
primaza sintetizira RNA primer na drugi verigi - obnovi 5’konec, skupaj spet poveže primaza
mRNA po sintezi njenih koncev

kakšni mehanizmi omogočajo vgradnjo prave ak na tRNA
vsaka ak ima specifično tRNA
also vsaka ima svojo aminoacil-tRNA sintetazo, ki katalizira vezavo ak na tRNA - prepoznajo tRNA po prepoznavnih zaporedjih- akceptorsko mesto in antikodonska zanka
aminoacil-tRNA sintetaza ima tudi sposobnost kontrolnega branja
mehanizem dvojnega sita: 1) aktivacijsko mesto - grobo sito → omogoči vezavo prave ak in še nekaterih manjših, večjih pa ne 2) editing mesto - fino sito →če je vezana napačna ak jo odstrani
EF-Tu preverja, če je prava ak gor
katere sestavine potrebujemo za iniciacijo sinteze proteina,
prokarionti:
podenote ribosoma (mala - 30S in pozneje še velika - 50S)
iniciacijski faktorji: IF1 se veže na 30S podenoto (A mesto), IF2 hidrolizira GTP in se veže s fMet-tRNA^Met, IF3 se veže na 30S in prepreči ponovno vezavo s 50S
iniciatorska tRNA: fMet-tRNA^fMet - nosi začetno ak
mRNA
evkarionti:
40S in 60S podenoti
eIF-ji + GTP (eIF2 prinese Met-tRNA)
tRNA: Met-tRNA^Met (ni formiliran Met)
mRNA (s 5’-G kapo in Poly-A repom)
opiši vse tri elongacijske faktorje pri bakterijah- kateri so in vloga
EF-Tu: ko veže GTP lahko tvori kompleks z aminoacil-tRNA in jo prinese na A mesto na ribosomu + preprečuje prekinitev esterske vezi med ak in tRNA
EF-Ts: izmenjuje GDP-GTP → regenerira EF-Tu (veže GTP gor namesto GDP in EF-Tu lahko spet deluje)
EF-G: premik ribosoma po RNA - vstopi z GTP-jem na A mesto, hidroliza GTP povzroči konf spremembo → mRNA in tRNA so potisnjene za en kodon naprej, peptidil-tRNA se premakne iz A na P mesto
kako je sestavljen genom SV40
1 krožna dsDNA dolga 5243bp
ima histone
regulatorna regija: 400bp, mesto ori (gor se veže antigen T), signali, ki regulirajo transkripcijo
zgodnja regija: zapisi za veliki antigen T, mali antigen t in protein 17kT
pozna regija: zapis za proteine kapside (VP 1, 2 in 3)