Transcription, maturation et lecture de l'ARN
1/24
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No study sessions yet.
25 Terms
Quel type d’ARN polymérase existe ?
Dans quelle direction est lue l’ADN et dans quelle direction est-elle synthétisée.
Il existe de type I (se trouve dans le nucléole) , II (principale) , et III.
Pol I : synthéthise l’ARN ribosomaux
Pol II : Produit l’ARNm
Pol III : transcrit les ARNt, les snRNA et miRNA pour la traduction.
L’ADN est lu de 3’ à 5’
Synthétise l’ARN de 5’ en 3’ directionnelle
Fonction de l’ARN messager ?
Copie temporaire et sert de modèle pour la traduction (synthèse des protéines)
Transfère l’information génétique du noyau vers les ribosomes du cytoplasme
Par quelle polymérase est-ce que l’ARNm est synthétisé?
Quand est-ce que l’ARNm est formé?
Synthétisé par la polymérase II
Formé lors de la transcription
Quelle modification subit l’ARNm après la transcription?
Ajout d’une coiffe 5’
Epissage
Ajout d’une queue poly(A) 3’
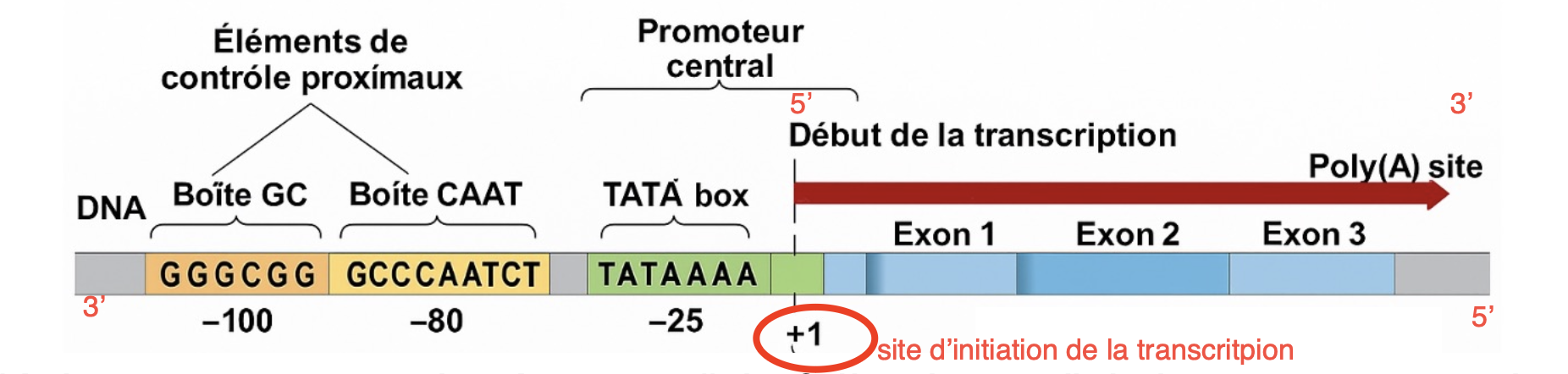
Qu’est-ce qu’un promoteur et à quoi elle sert?
C’est une séquence particulière indiquant à l’ARN polymérase II où commence la transcription avec les facteurs nécessaire.
Exemple de promoteurs
Boite TATA : séquence riche en T et A reconnu par le facteur TBP
Boite CAAT : augmente l’efficacité de la transcription
Elements GC : Régule l’intensité de la transcription
Site d’initiation +1 : Endroit où commence la synthèse d’ARN
Rôle du facteur TFIID
C’est un complexe d’initiation de base pour le démarrage de la trancription :
Se lie à la boite TATA permettant à d’autres facteurs de recruter l’ARN polymérase II sur le promoteur
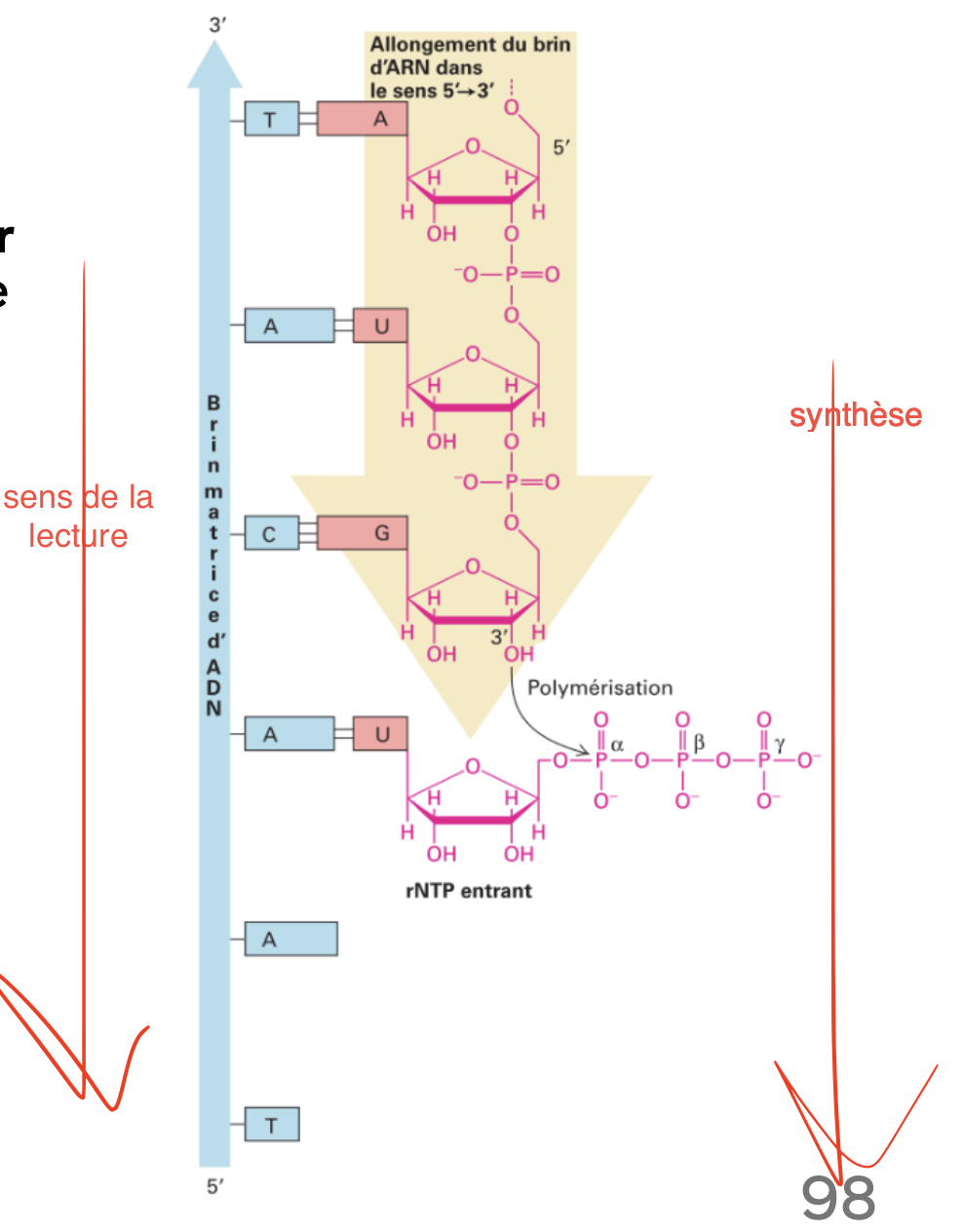
Mécanisme d’élongation de la transcription
l’ARN polymérase II ouvre la double hélice sans l’aide d’hélicase
l’ARNm progresse le long du brin matrice de 3’ à 5’ mais les nucléotides s’assemblent de 5’ à 3’ créant une hélice hybride ADN-ARN transitoire
Est-ce qu’il est possible que la transcription se fasse sur 2 bases complémentaires au même endroit ?
Très très rare car cela signifie qu’il y ait un promoteur pile au bon endroit entre 2 bases complémentaires.
Formation de la liaison phosphodiester par l’ARN polymérase :
ARN polymérase ajoute les ribonucléotides avec une liaison phosphodiester entre le OH 3’ le prochain ribonucléotide dans la direction 5’ à 3’
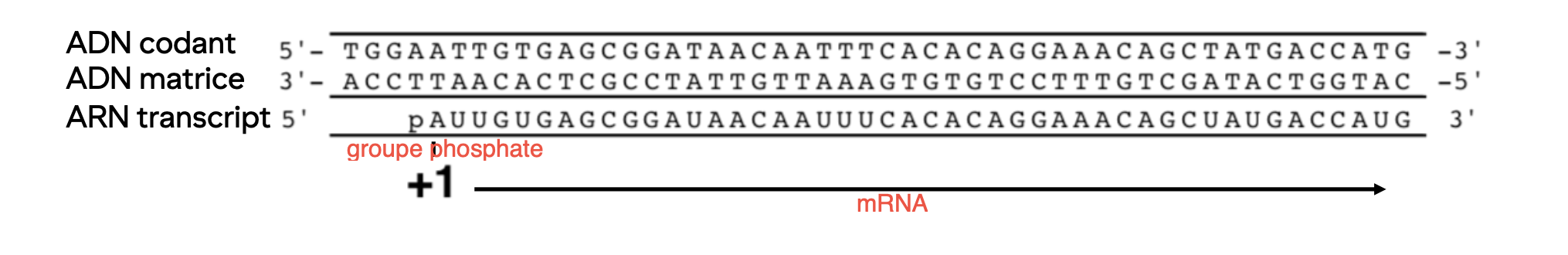
Relation entre le brin
codant
matrice
transcript
La séquence transcript est identique au codant à l’exception de T→ U
Le site +1 est le point du début de la transcription
Les 3 étapes du pré-ARN à l’ARNm mature
Ajout d’une coiffe 5’
Polyadénylation en 3’
Epissage
Qu’est-ce qui constitue la coiffe 5’ et sa fonction ?
Ajout du 7-méthylguanosine reliant une liaison 5’ du triphosphate au 5’ du ribonucléotide.
Le ribonucléotide est méthylé en 2’
Protège de la dégradation
Sert de signal de reconnaissance pour le ribosome dans la traduction
Stabilise de l’ARNm et sa reconaissance
Mécanisme d’ajout de la coiffe 5’
Phosphohydrolase : On enlève un phosphate à l’extrémité 5’ du pré-ARNm
Guanylyl transférase : GTP perd 2 phosphates et devient un GMP qui fait une liaison 5’-5’ triphosphate P +PP = PPP
Méthyltransférase : ajout de groupe méthyles sur la guanine en 7’ et parfois sur le premier ribose en 2'
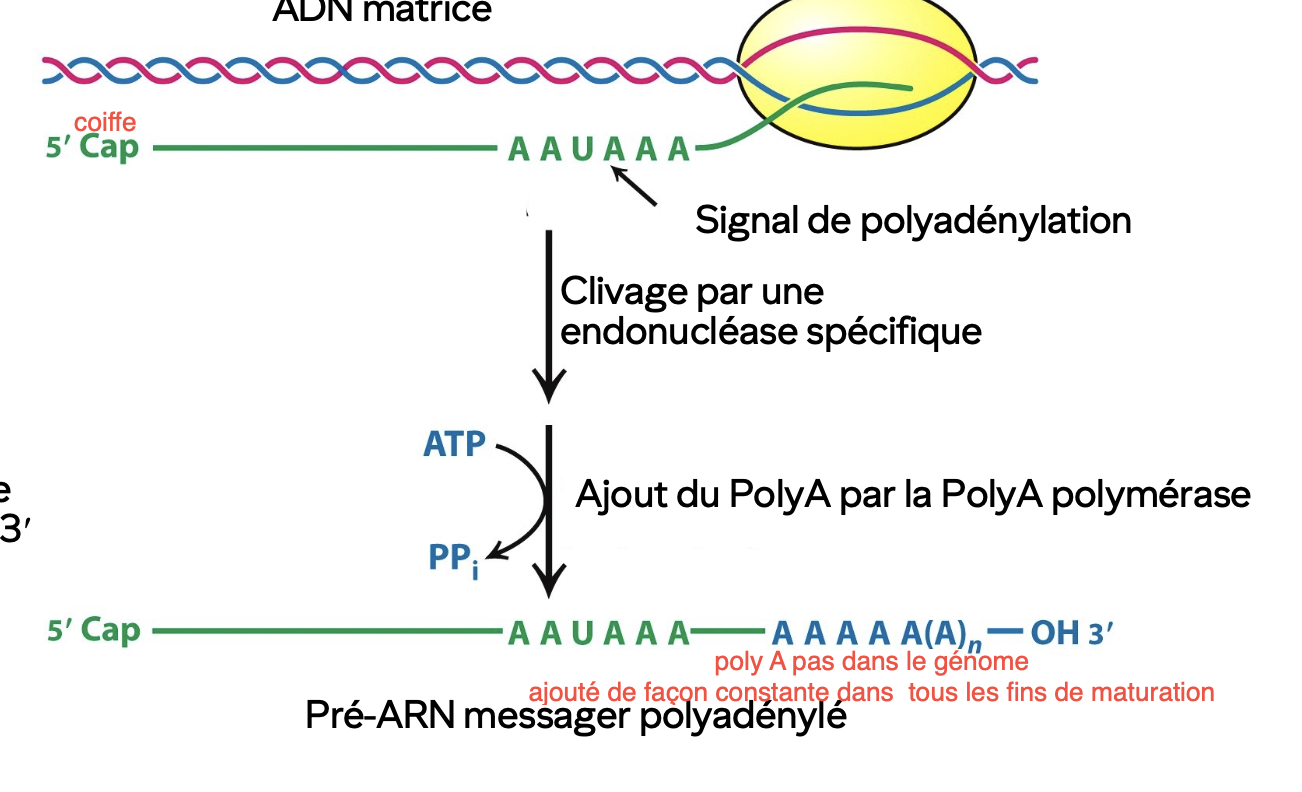
Polyadénylation en 3’
Stabilise l’ARNm
Marque la fin de la transcription
Identifiable avec le motif AAUAAA suivi d’un site de clivage 15-30 nucléotides après.
Après le clivage, il y aura une polymérisation d’adénine à l’extrémité 3’ demandant de l’ATP
Epissage
Retirement des introns pour ne garder que les exons. Les exons sont assemblés pour former l’ARNm mature.
Réalisé par le spliceosome
Augmente la diversité des protéines car peut choisir que certains exons à d’autres.
Séquence pour l’épissage
début d’intron : GU fin d’intron : AG
Point d’embranchement des 2 parties de l’intro : A formant un lasso
Nécessite des petits ARN nucléaires (ARNsn) comme U1 et U2 formant le spliceosome
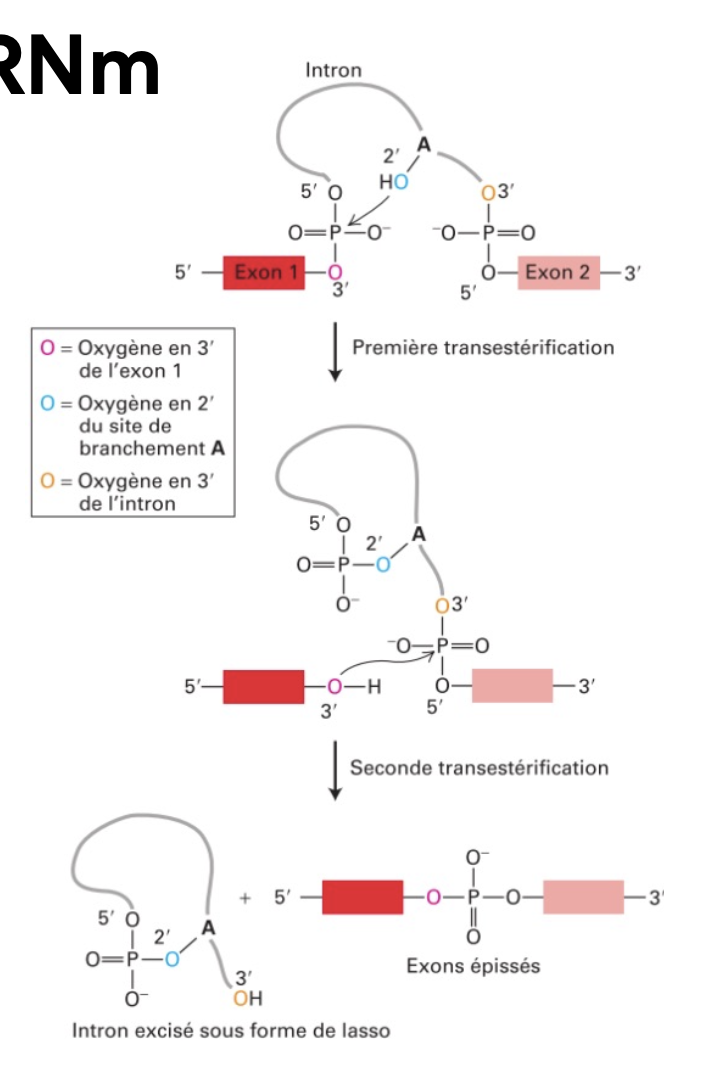
Mécanisme de l’épissage du ARNm
2 réactions de transestérification :
le 2’OH de l’adénine (point de branchement A) se lie au phosphate (groupe donneur GU en 5’)
Puis le 3’OH de l’exon 1 attaque le groupe phosphate (site accepteur AG en 3’). Ce qui relie les exons ensemble et dégage l’intron.
Structure d’un ARNm
coiffe en 5’
queue en poly(A) 3’
Régions 5’UTR et 3’UTR encadrent le codon de départ et d’arrivée.
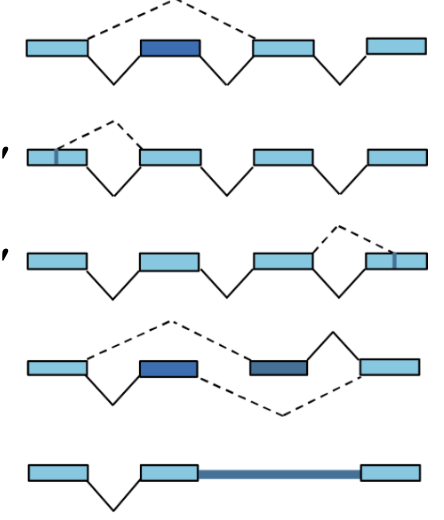
Donne le nom des épissage alternatifs sur l’image :
Forme des isoformes protéiques
Saut d’exon
Site d’épissage alternatif en 5’
Site d’épissage alternatif en 3’
Exons mutuellement exclusifs
Rétention d’exon.
Exemple de l’IgM pour l’épissage alternatif
L’épissage alternatif permet de fournir des fonctions divers à une protéine
E.g. IgM selon les exons retenu et les polyA, il peut devenir membranaire ou libérer dans la cellule (sécrétée)
Code génétique
3 bases = 1 codon = 1 acide aminé
Plusieurs codons pour 1 aa
Codon START : AUG
Codon STOP : UAA, UAG, UGA
Le décalage dans la lecture des bases
Change complétement la lecture des codons.
3 possibilités de lecture différentes
ARN de transfert :
Chaque acide aminé est lié à un ARNt avec l’aminoacyl-ARNt synthétase avec une liaison riche en énergie
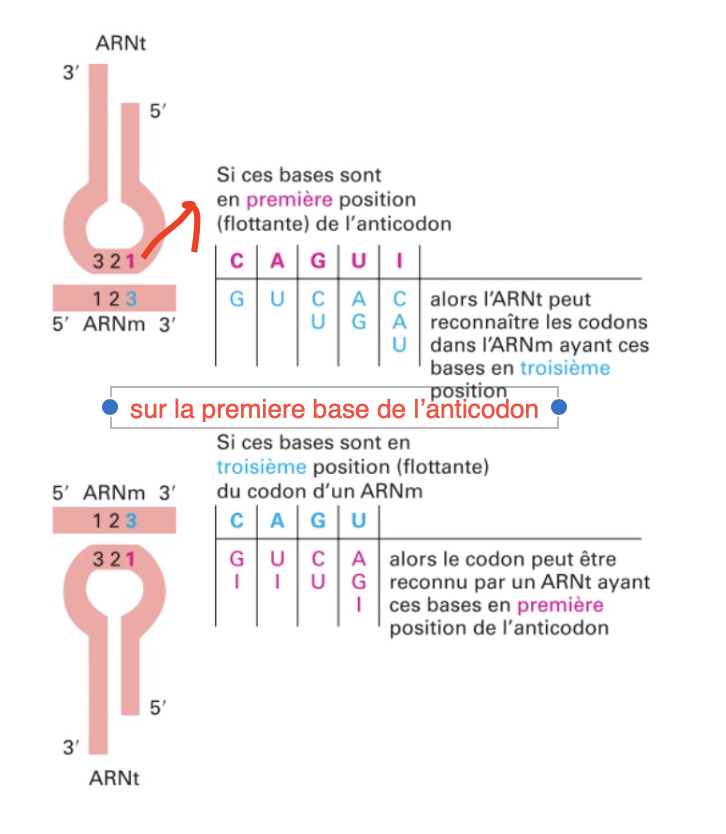
Dégéresence et flexibilité du code génétique :
La base du premier ARN de transfert (coté 5’) est souvent modifié et vu que la lecture se fait du sens 3’ à 5’ pour le codon, certains codons vont pour le même acide aminé
Résulats : Un ARNt peut reconnaitre plusieurs codons synonyme car elle est déterminé par le premiere base de son anticodon contre la troisième pour le codon.
Environ 40 ARNt pour 61 codons différents car 3 codons STOP