CM1: Structure, organisation et dynamique du génome humain
1/20
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No study sessions yet.
21 Terms
1) Encadrement réglementaire des activités de génétique en France : Convention
Que signifie bioéthique?
d’Oviedo, Loi de bioéthique, Plan France Médecine Génomique 2025
Quelle est le seul encadrement reglementaire autour de l’étude genetique bioethique? Quelle convention contraint la bioetique et de quelle loi s’inspire telle? quest ce qu un ogem (organisme gene etc)
quelle sont les 4 articles et de quoi parle il?
Est ce que cette loi est figée? (non exemple “protocoloe additionnel strasbourg 2008”)






1) Encadrement réglementaire des activités de génétique en France : Convention:
Quelles sont les trois finalités aux examens génétiques en france?
quels sont les differnets types de prelevement?
Que dit le code de la santé p!ublique, code civil, sur le consentement lors l’examen des caracteristique genetiques constitutiinelles?
Est ce que tout le monde peut faire de stest genetiques? et est ce que ca peut etre fait nimporte ou? (non les praticiens agréés et slmtdans des laboratoires de biologie médicale autorisés à cet effet par l’Agence Régionale de Santé.)
En quoi consisite examen des caractéristiques génétiques constitutionnelles et somatiques selon le code de la sante publique?
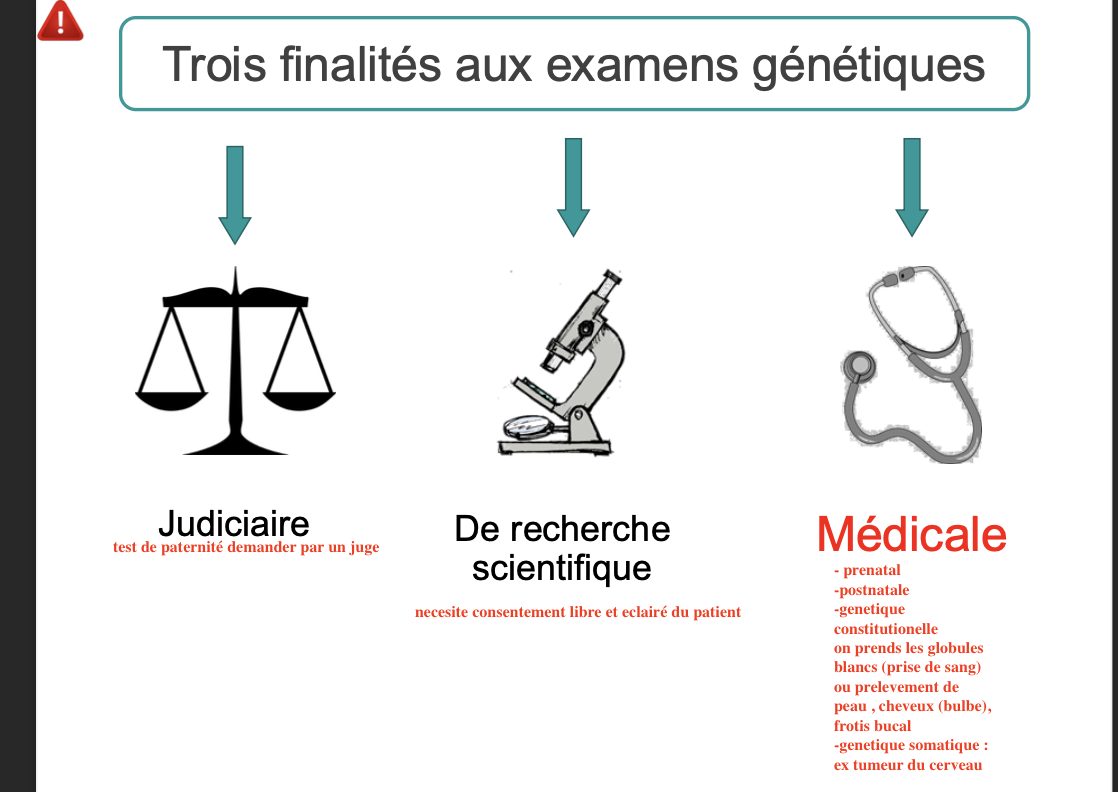

code civil: la personne par ecrit doit consentir a ce test ce consentement repose sur sa connaisance du proczessus de lexamen , sa nature, l’indication de l’examen, de son objectif , de l’apparition de données incidentes (mutations trouvee dans un autre genes, lui demender si ils veut savoir si on les trouve, cependant si il refuse de savoir ces donnees il est responsable de les communiquer ou non aux autres. Le consentement est révocable en tout ou partie, sans forme et à tout moment.

2) Rappels : structure de l’ADN, taille des génomes et valeur C
Quest ce que le genome?
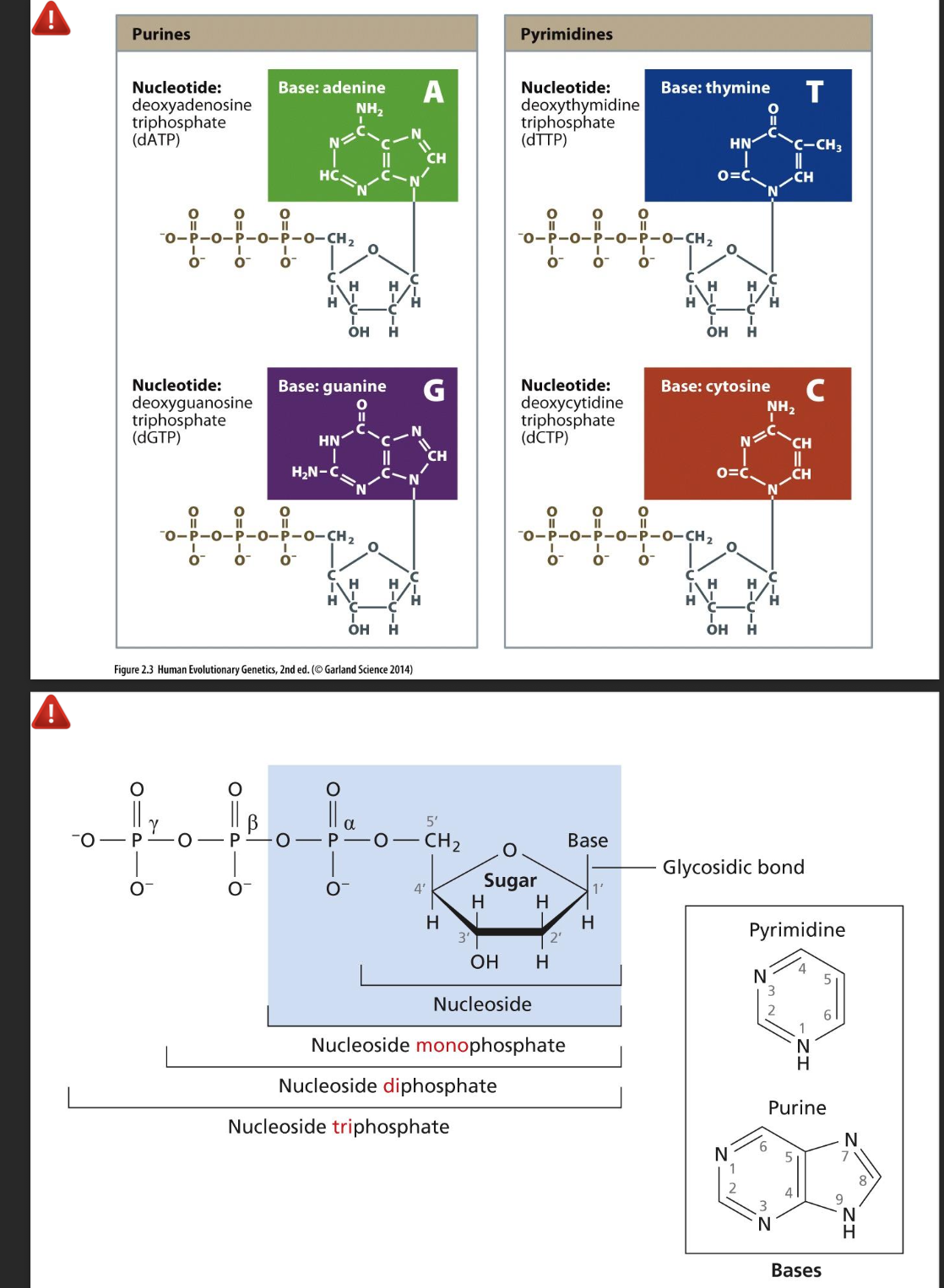
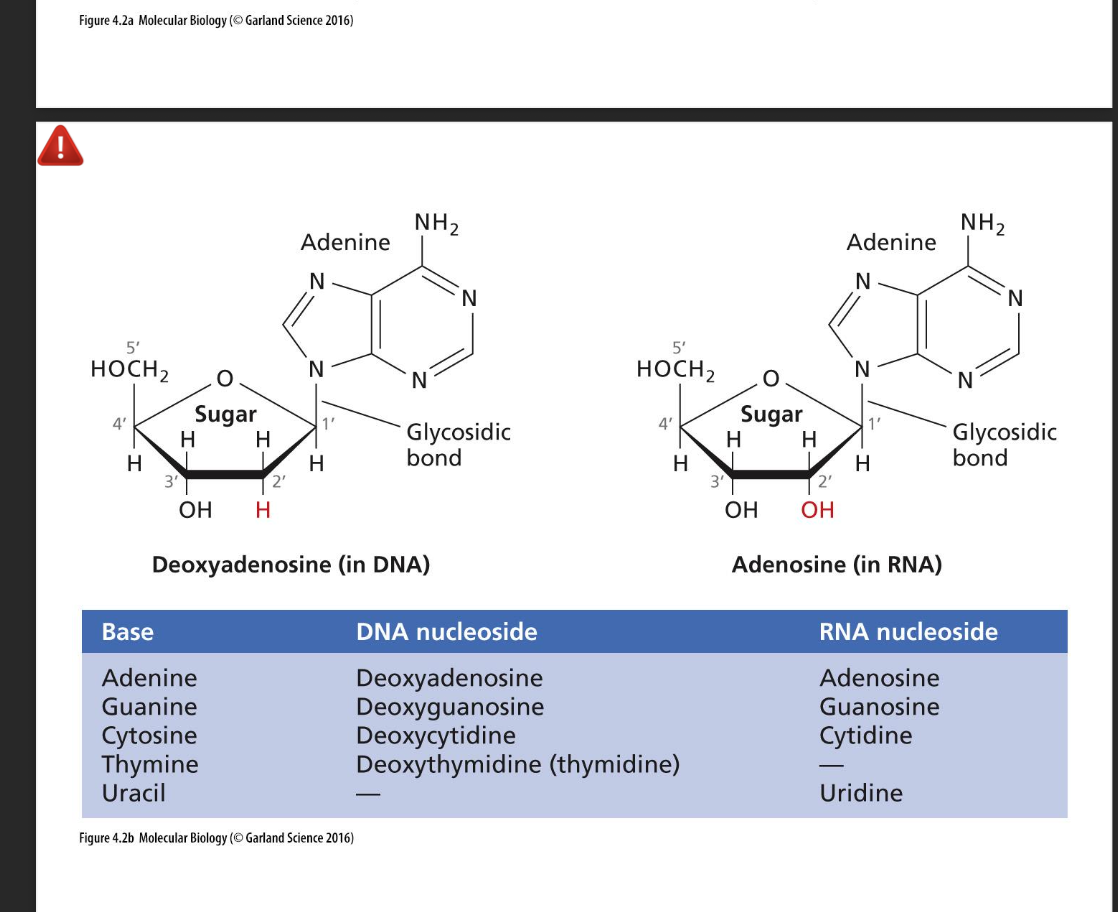
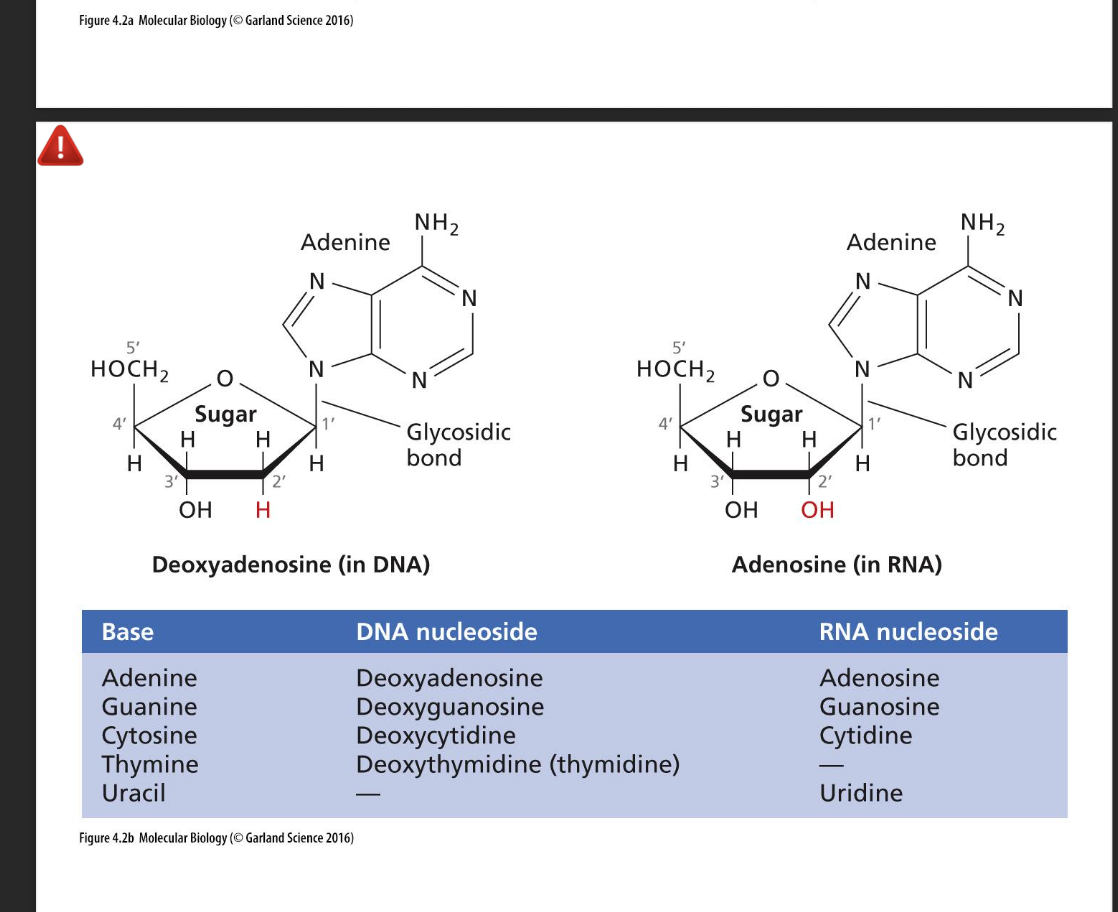
Que signifie ADN? quelle sont c composant et sa structure?
Caracteristique? (double helice, ordre de grandeur etc)
Quest ce que la valeur c est pk est ele interessante?(variation selon les especes)
Quest ce que le paradoxe d ela valeur C?
Quelle est le pourcentage d’adn codant pour les humains et pour les bacteries?(1% et 100%)
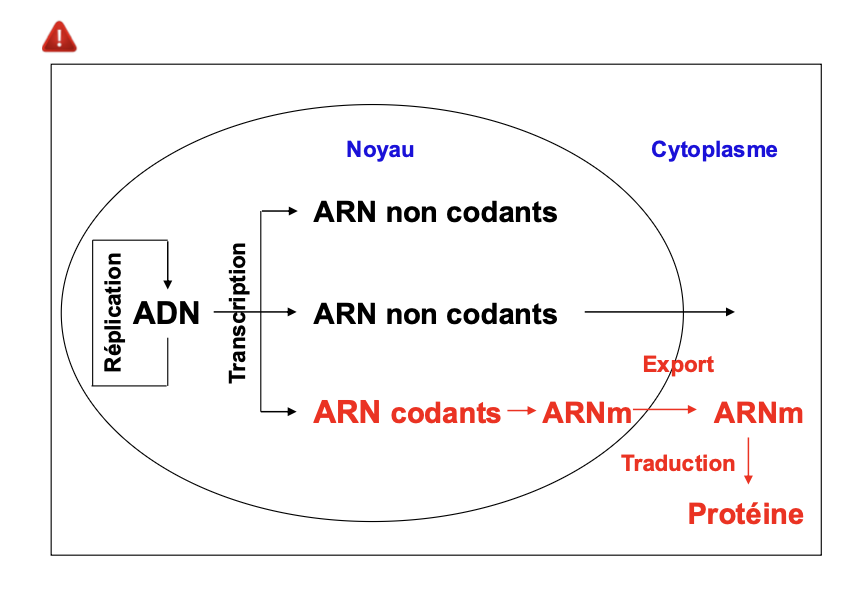
Le mot génome désigne l'ensemble de l'information héréditaire d'un organisme présente en totalité dans chaque cellule. Le support matériel de l'information génétique est l'ADN (A, T, G et C) plus rarement l’ARN(A, U, G et C)
La valeur C représente la taille d'un génome qui peut être exprimée en millions de paires de bases (Mb) ou en pico-gramme (10-12g) : 1 pg correspond à 978 Mb.
La valeur C est très variable en fonction des espèces:
VIRUS : quelques milliers
BACTERIES : quelques millions
MAMMIFERES : quelques milliards
La taille des génomes ne corrèle pas avec la
complexité des organismes. C'est le paradoxe de la
valeur C
im occlusion
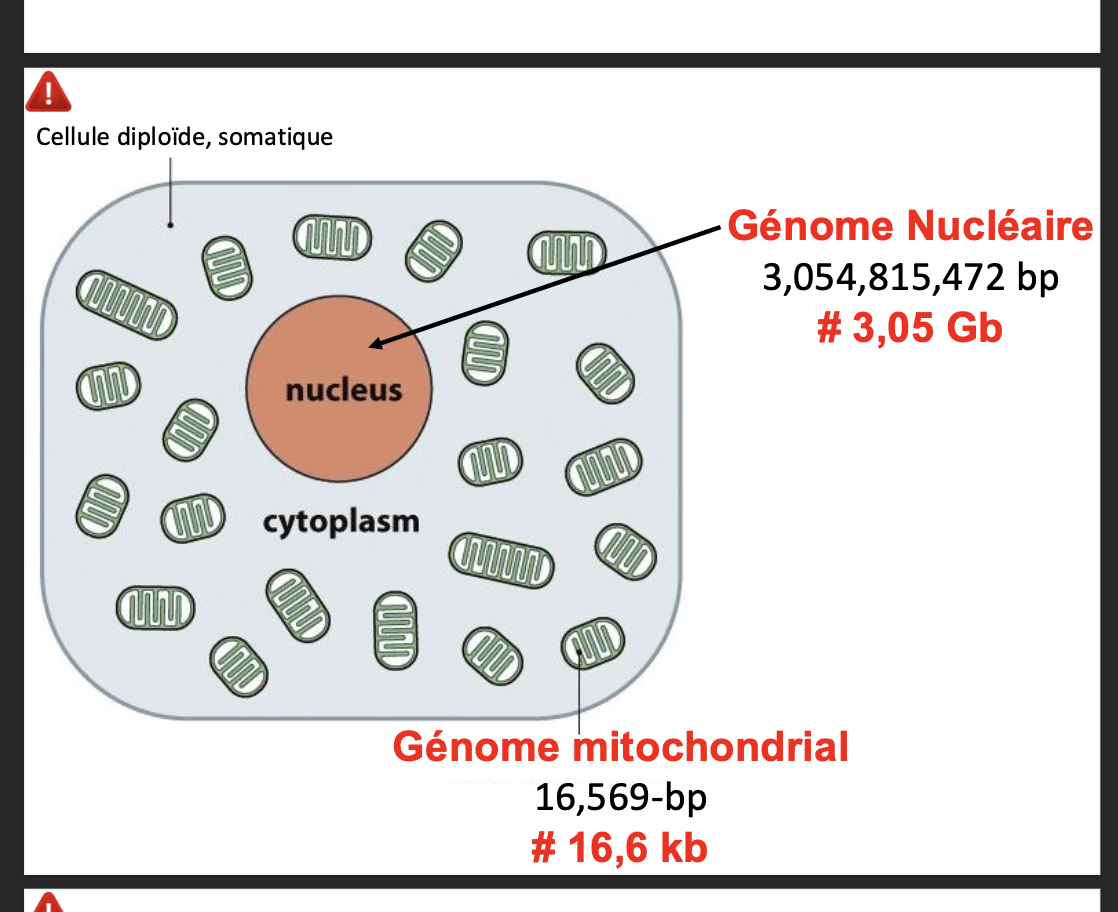
3) Présentation générale du génome humain nucléaire et du génome mitochondrial
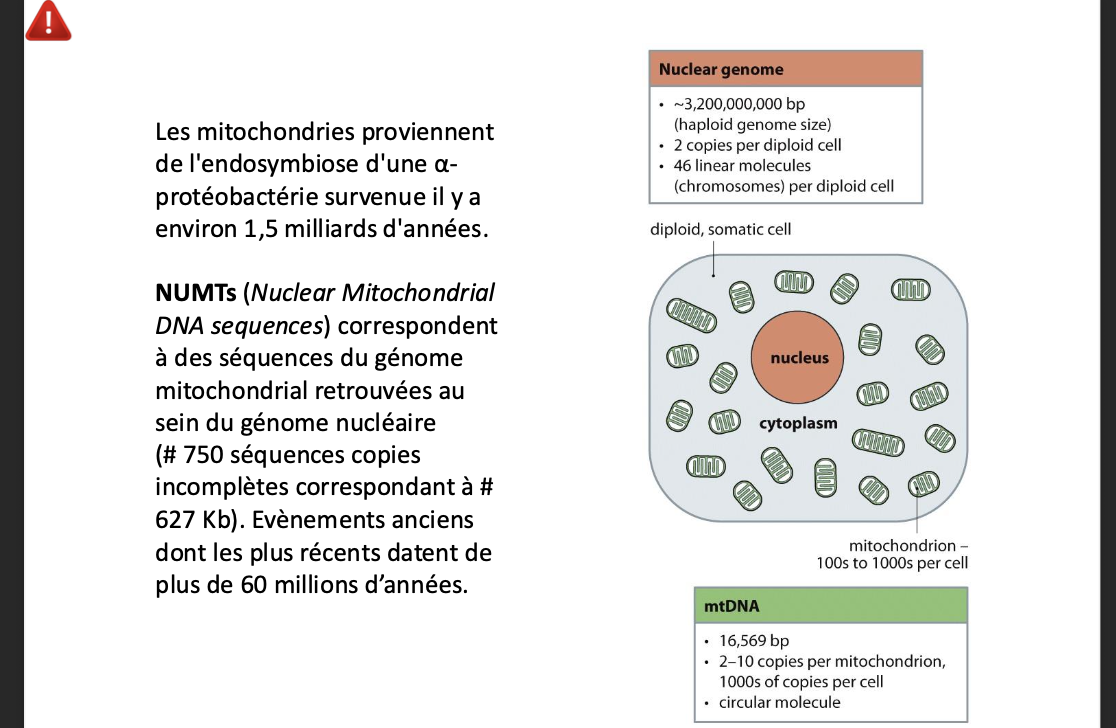
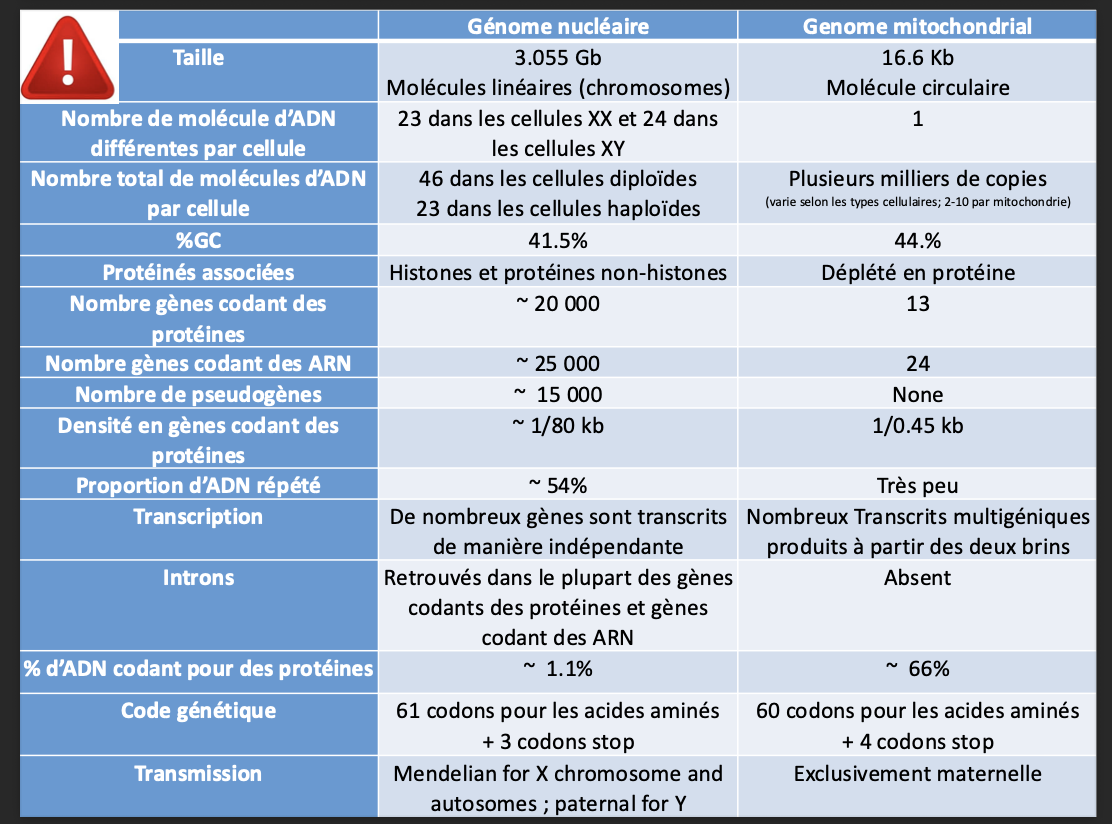
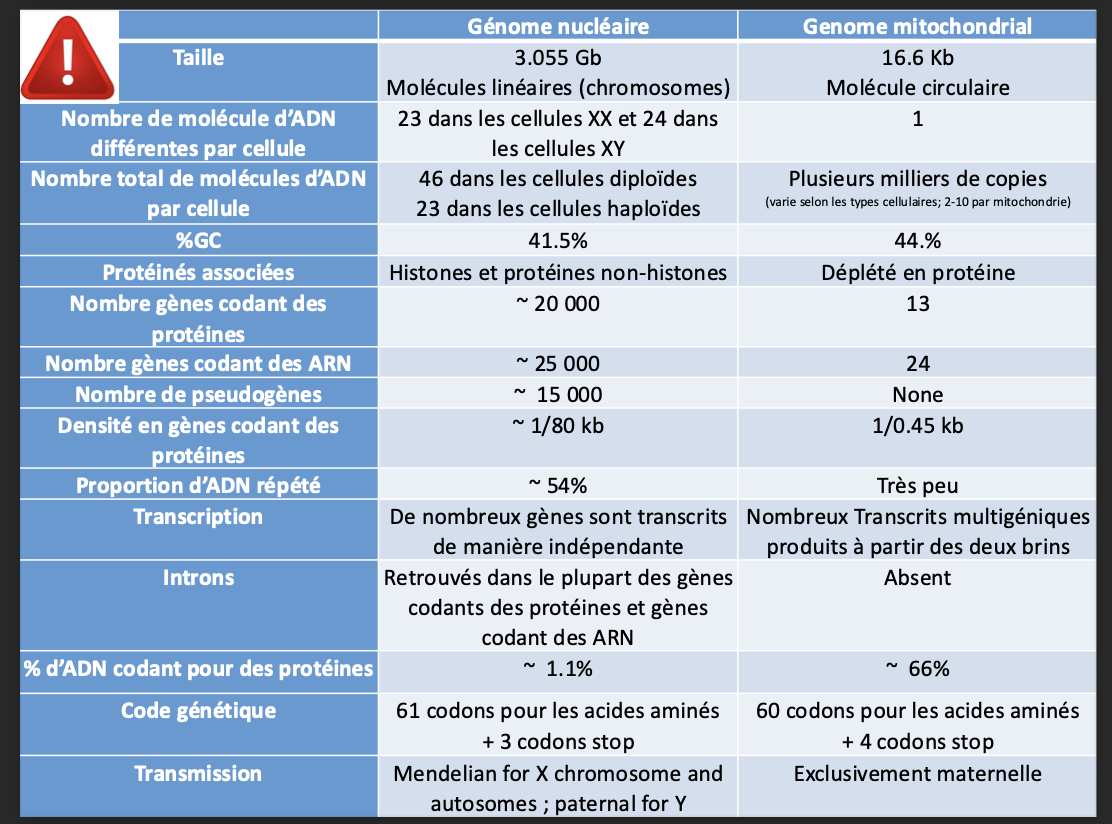
Ordre de grandeur du genome nucleaire et mitochondrial? (origine de la mitochonries?)
Quest ce que les NUMTs
combien ya til de molecule dadn diff dan sle genome nucleaire (23 chez XX et 24 chez XY)
A combien la taille du protéome est estimée? (La taille du protéome mitochondrial humain est estimée à plus d'un millier de protéines, dont environ 1% codées par le génome codées mitochondrial (13 protéines). Toutes les autres protéines sont par l e génome nucléaire (cf. adressage des protéines mitochondriales))
Quels sont les trois caracteriques du code genetique?
Quelle sont les codons stop et start dans le genome nucleaire? est ce que c les memes pour le mitoconriale? (non)
Comment est organiser un caryotype? (par ordre decroissant (sauf le 21 qui est le plu petit) Taille du plus petit et du plus grand?
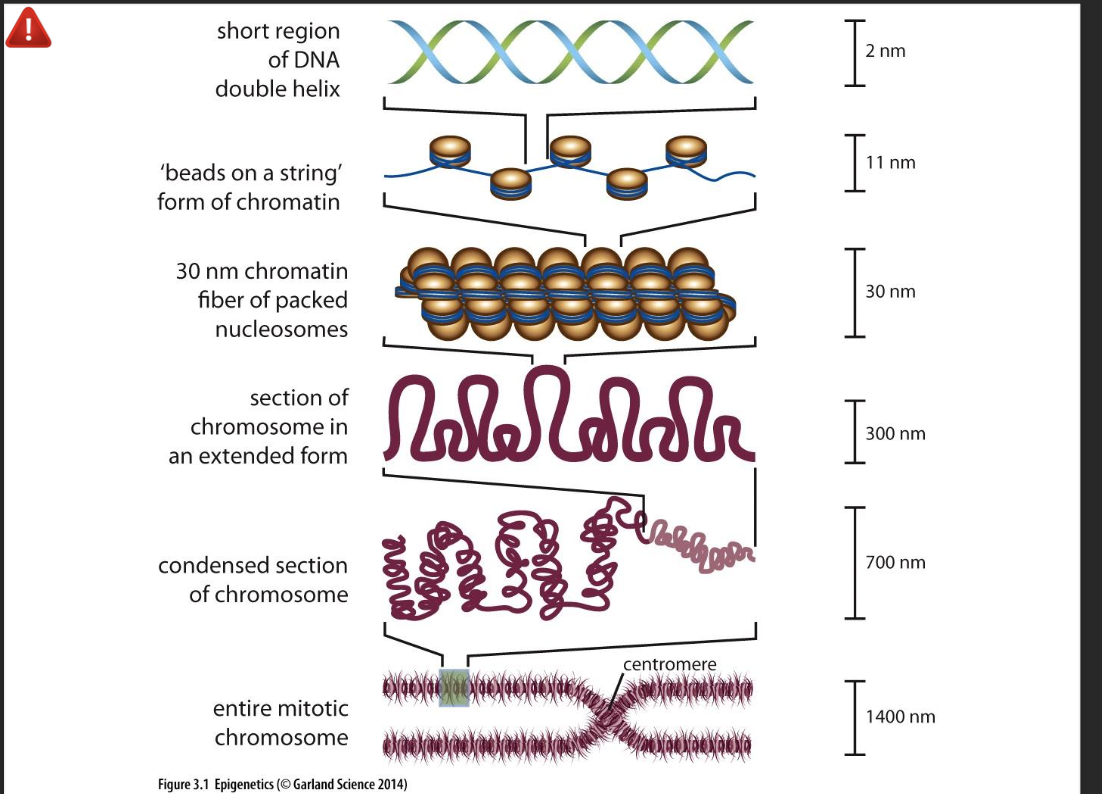
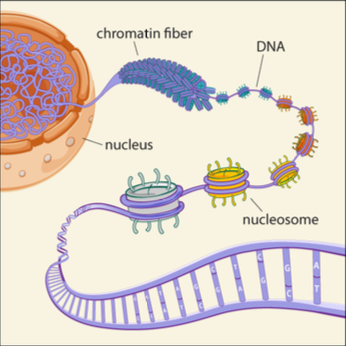
Quest ce que la chromatine et ses roles?
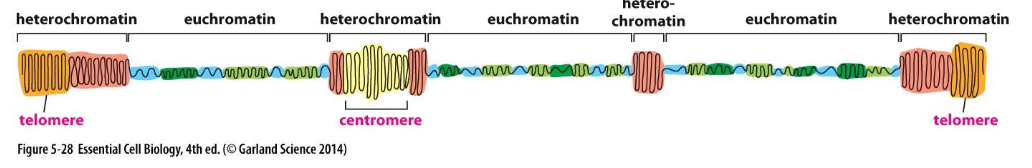
Qu’est ce aue l’euchromatine, l’heterochromatine?
Quels sont les deux types d’hetrochromatines?
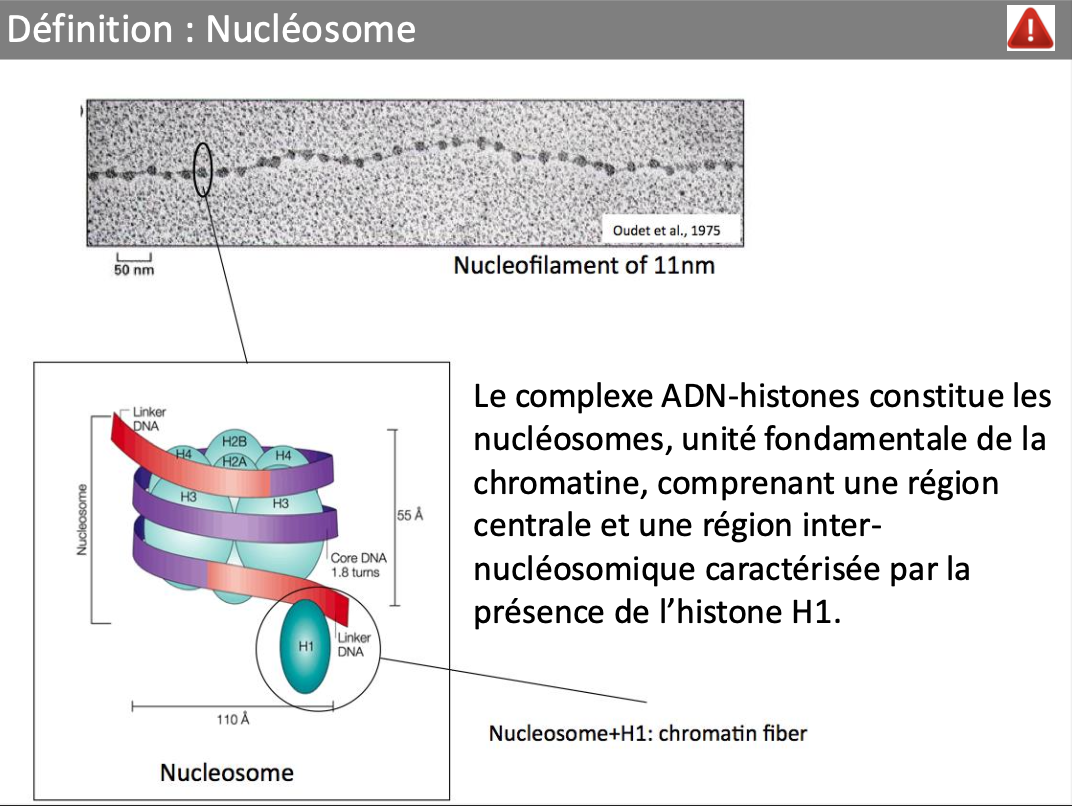
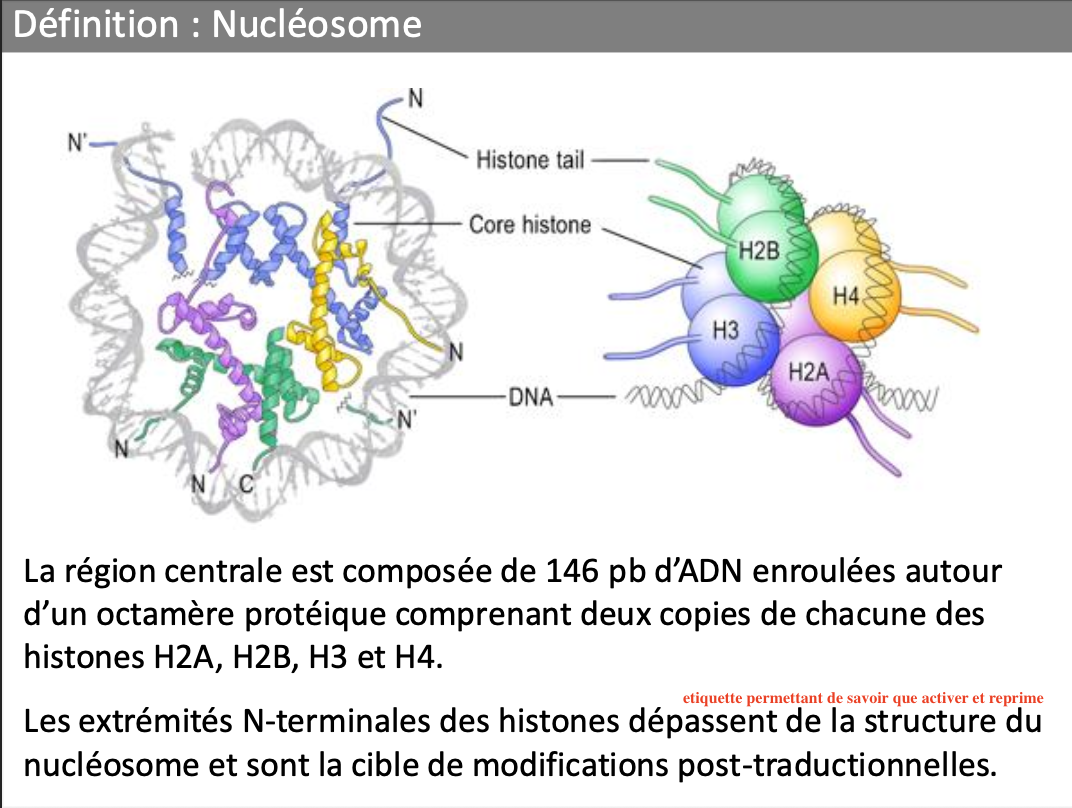
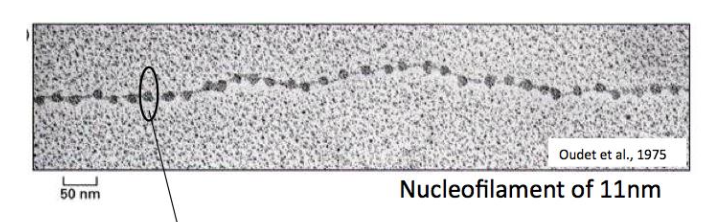
Qu’est ce que le nucleosome?
Ou se font les modif post trad sur les histones?

im occlusion
im occlusion
4) The Human Genome Project
en combien deb temps on peut sequencer le genome et combien ca coute? (72h environ)(1000$)
Quest ce que le human genome project et combien a il coute? (meme pris que pour aller sur la lune)
Mendel? Thomas Morgan? watson et crick? Rosalind Franklin? Sanger? gary ulysse? Leroy hood?
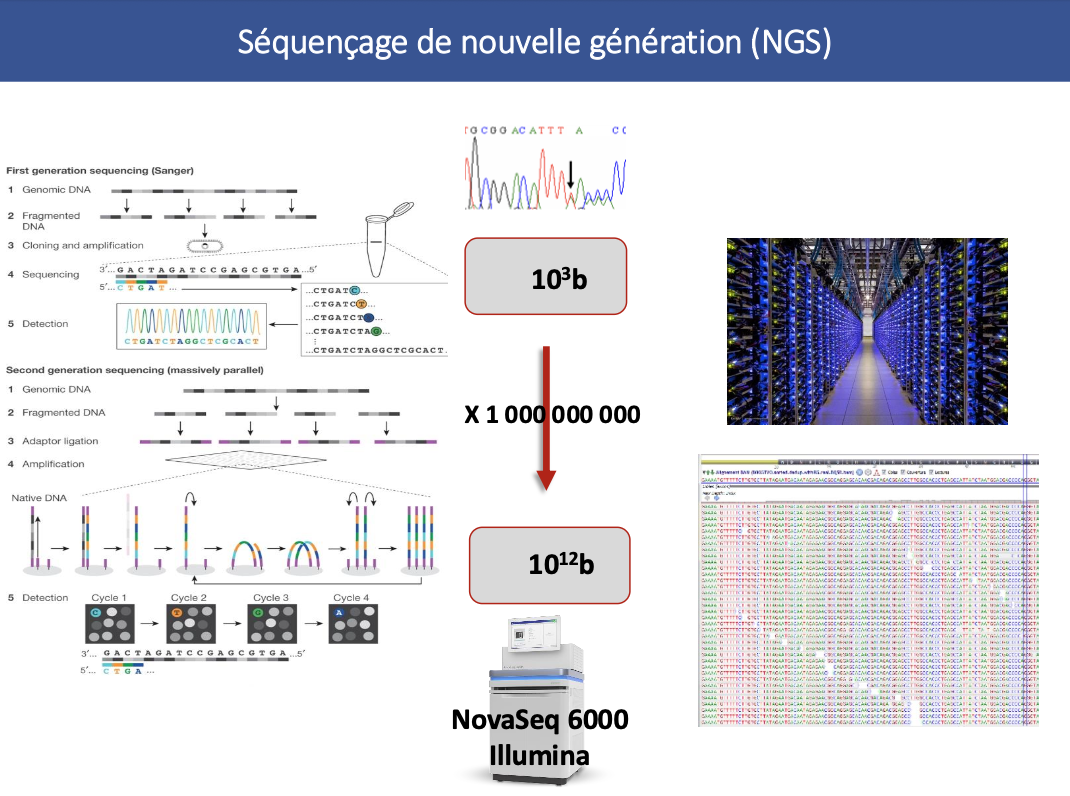
First generational sequencing?
Qu’est ce que le shotgun sequencing?
Technique de craig van
Comment se fait le NGS et quelle est la diff avec Sanger?
fastest genetic diagnosis to this day ? (- de 50mins)
Qu’est ce que le projet gnomAD? et UK Biobank?


im occlu
5) Présentation des gènes codant les protéines : définition de la notion de gène, transcription, épissage et traduction
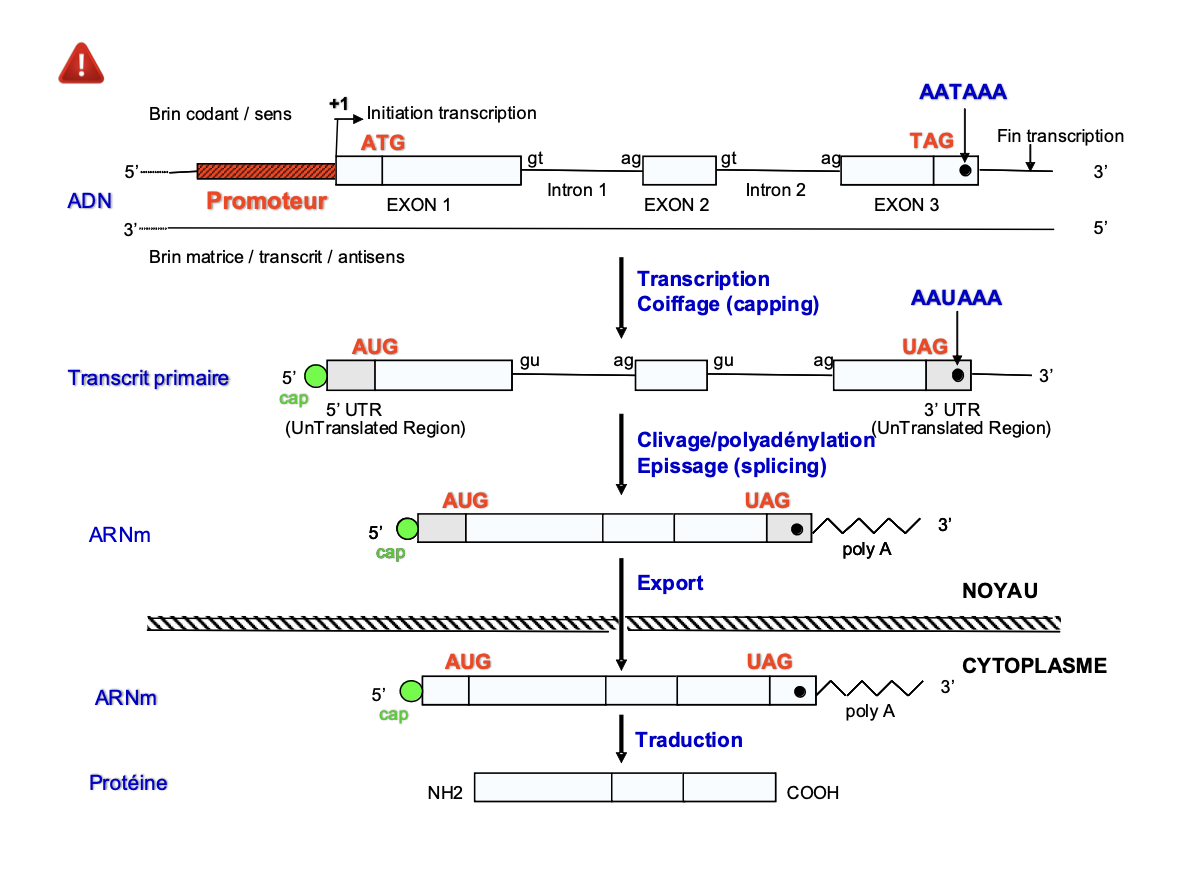
Qu’est ce qu’un gène?
Schematiser le passage d’adn a ARN non codants er Poroteine?

suite
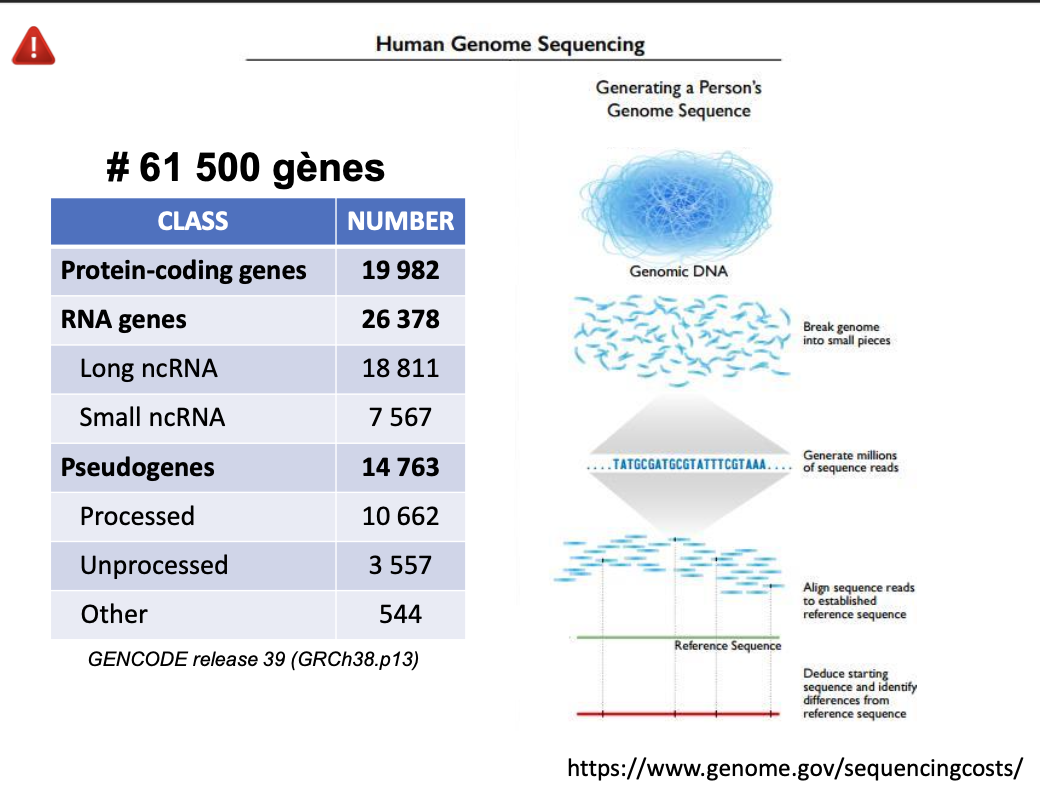
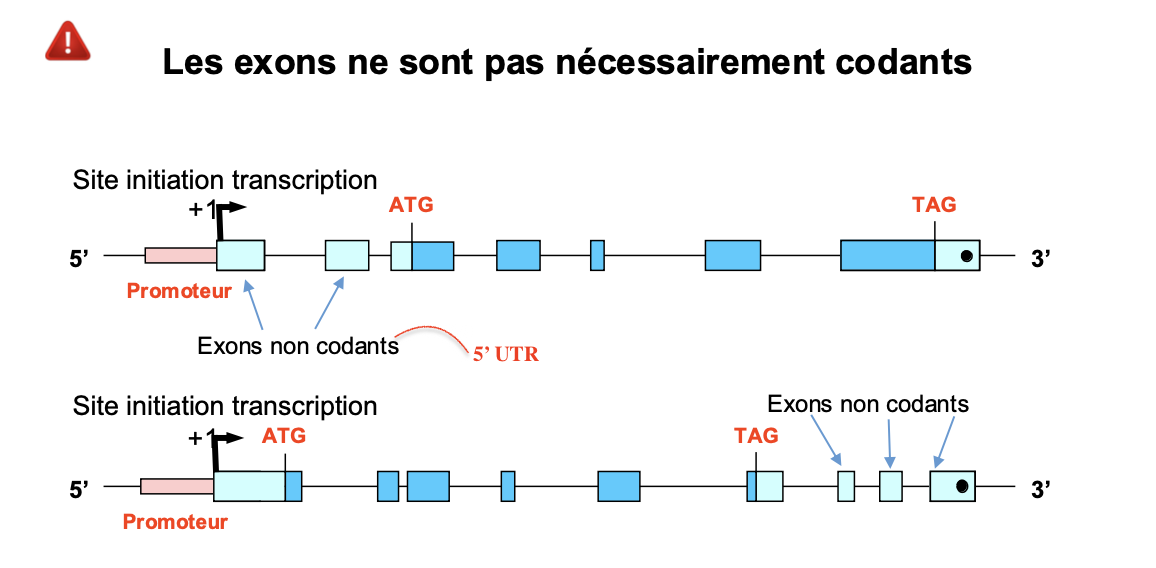
quelles proportions represente les exons dans le genome dans les gens codant? (Caractéristiques du génome humain
# 20 000 gènes codant des protéines dont les exons
occupent 1.2 % du génome)
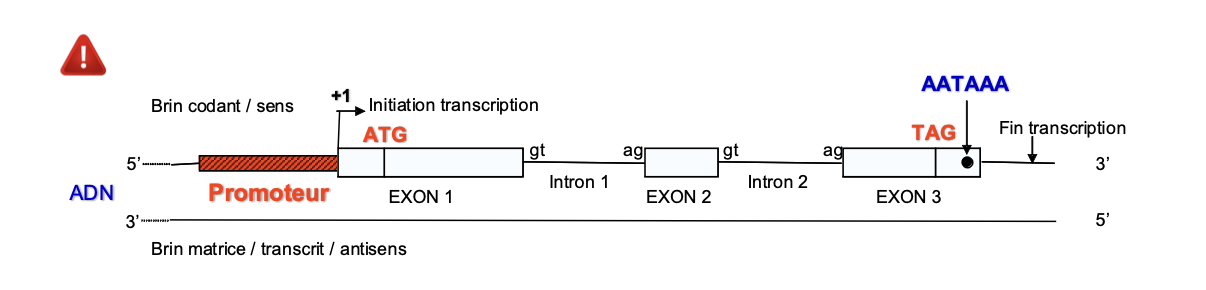
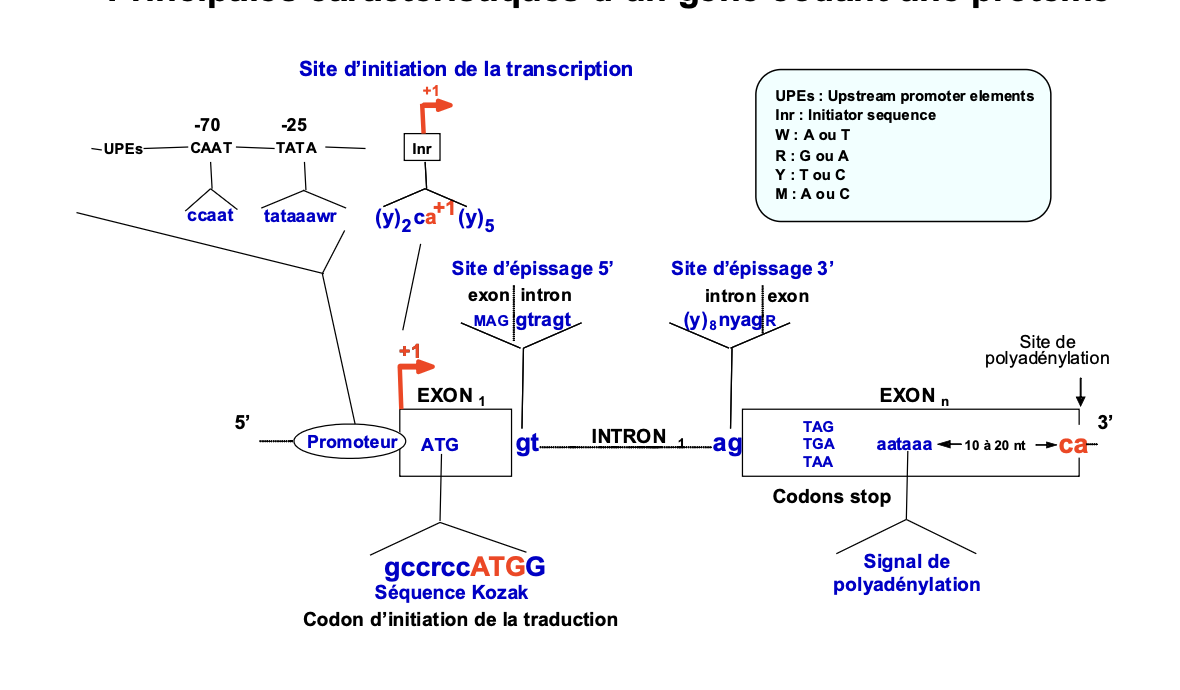
promoteur def: région de l’ADN située habituellement en amont (en 5’) d’un gène et indispensable à sa transcription en ARN. Il comporte le site d’initiation de la transcription (+1) et les sites de fixation de facteurs de transcription et de l’ARN polymérase.
ARN Poly
Facteurs de transcription
Un gene codant par une proteine commence par un exon
ATG ->Meth (AA qui initie la traduction) qui peut etre dans lexon un ou deux (dans ce cas il y a une UTRs→ sert a la regulation et stabilite de l’arn m) la queue poly A est ajoutée lors de la maturation de l’arn messager.
quels sont les difernents type d’arn pol?

Les deux types de genes implique dans la differenciation? Par convention, on appelle +1 le 1er nucléotide à partir duquel la transcription commence et -1 le nucléotide qui précède.
Le contenu en ADN des cellules somatiques (à l’exclusion en particulier des lymphocytes et des neurones) est # identique à celui de l’œuf fécondé. C’est le profil des gènes exprimés dans les cellules qui est à l’origine de la différenciation cellulaire.
On distingue :
- les gènes exprimés dans toutes les cellules qui sont essentiels aux fonctions générales de la cellule : gènes domestiques ou «housekeeping genes»
- et les gènes exprimés spécifiquement dans certaines cellules ou certains tissus : «tissue-specific genes»
def enhancers : régions de l'ADN sur lesquelles se fixent des facteurs de transcription pour activer la transcription de gène(s). Ils agissent en cis et peuvent être situés jusqu'à 1 million de paires de bases (en amont ou en aval) des gènes régulés.
def silencer: régions de l’ADN sur lesquelles se fixent des facteurs
de transcription pour inhiber la transcription de gène(s).
Les enhancers et les silencers agissent en cis et peuvent être situés
jusqu'à 1 million de paires de bases (en amont ou en aval) des gènes
régulés.
quest ce que les region regulatrices distales ou proximales?
Comment fonctionne les enhacers ou les silencers?(ils agissnt en cis sur la meme molecule d’adn (pas le k homologue))
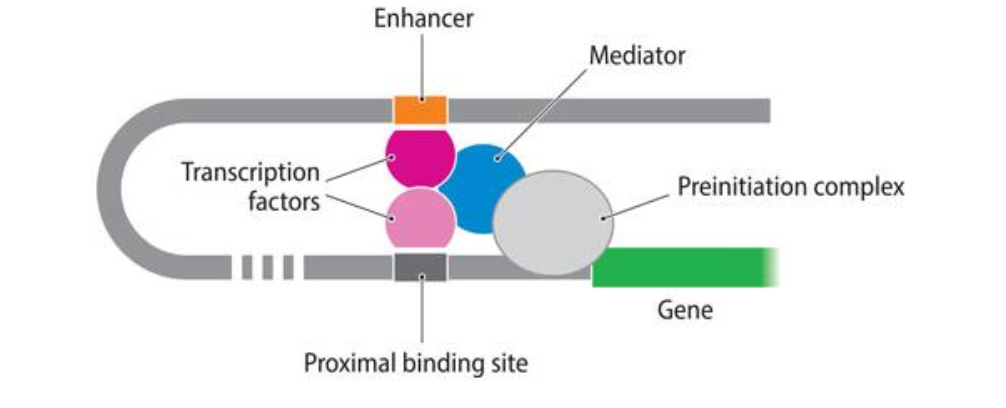
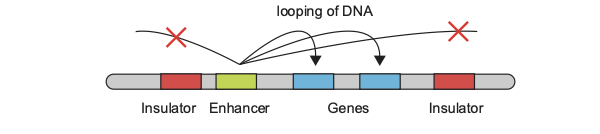
quest ce quun insulateurs? régions de l’ADN sur lesquelles se fixent des facteurs
empêchant un "enhancer" (ou un "silencer") d'activer (ou d'inhiber) la
transcription d'un autre gène voisin.
qu’ets ce qu’un exon? partie du gène qui persiste lors de la maturation (épissage) du transcrit primaire en ARNm. On dénombre plus de 500 000 exons dans les gènes codants les protéines. les exons ne sont pas particulierement des multiples de trois, des mutations atteignat l"‘epissage peuvente decaler le cadre de lecture.
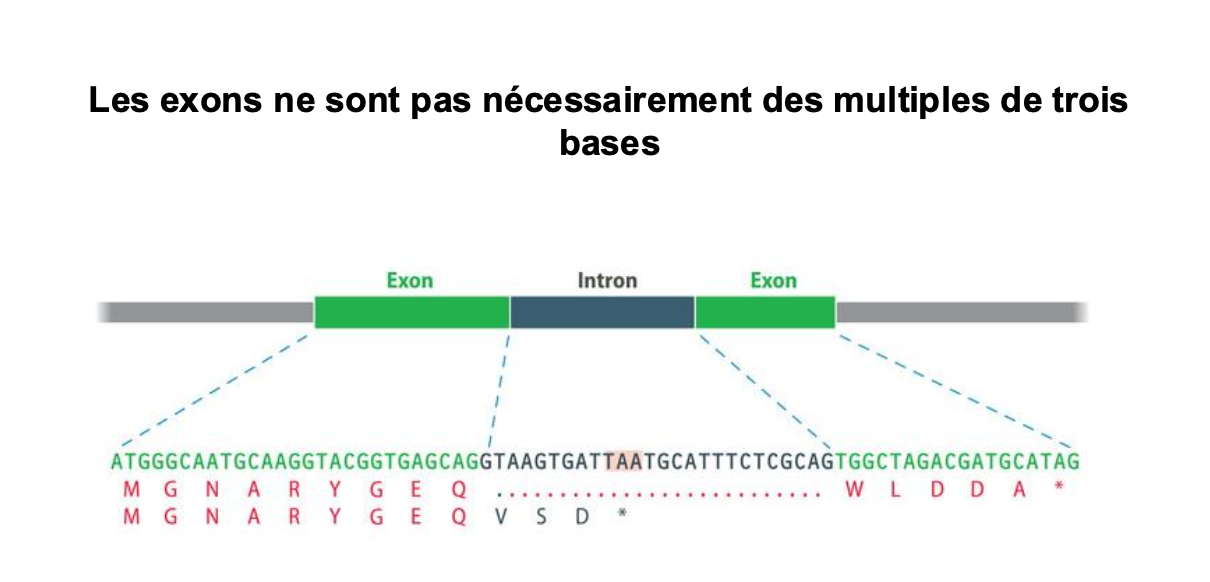
INTRON : partie du gène située entre deux exons et qui est excisée lors de la maturation (épissage) du transcrit primaire en ARNm.
UTR (Untranslated Transcribed Region) : régions transcrites non
traduites (5’UTR et 3’UTR).
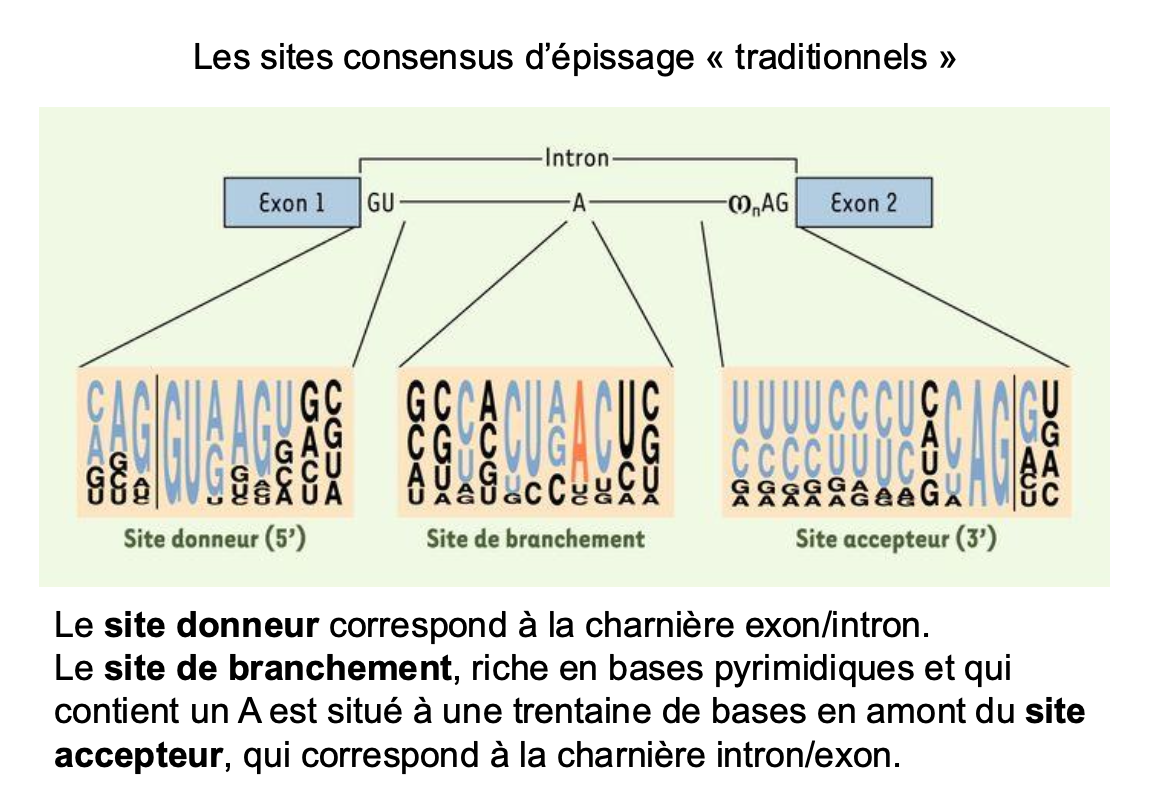
SEQUENCES CONSENSUS : séquences nucléotidiques impliquées dans des fonctions semblables et ne présentant entre elles que quelques variations de séquence nucléotidique. ex: TATA box, le gt (site d’epissage 5’), et ag (site d’epissage 3’), sequence kozak (codon dinitiation de la trad pour le ribosome)
Quels sont les sites consences d’epissage “traditionels”?
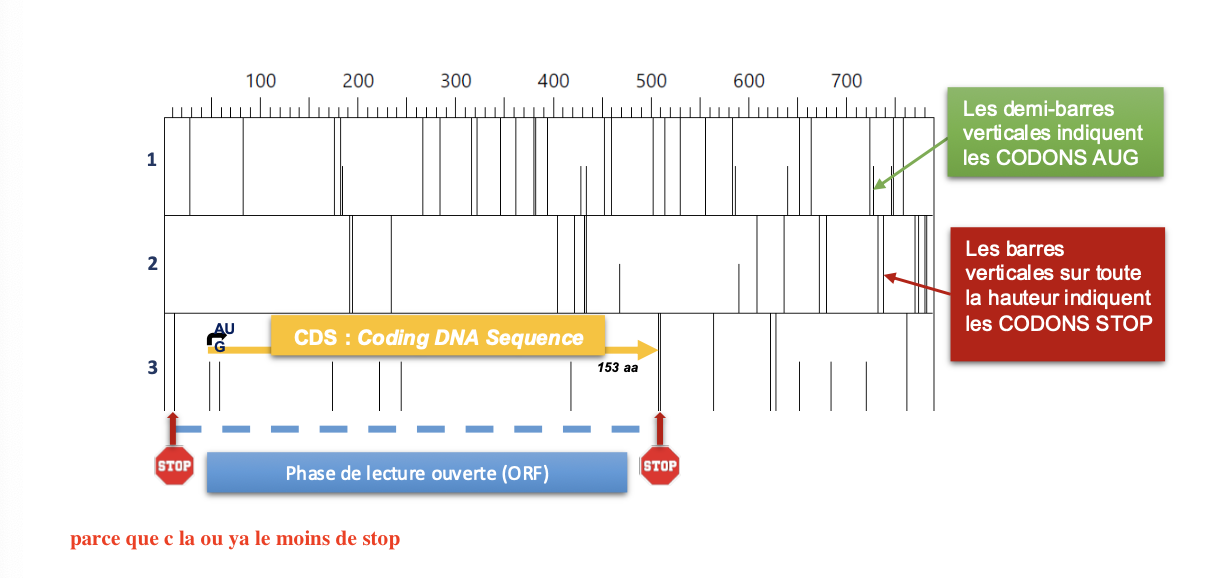
Le code genetique? Le code génétique est dégénéré (plusieurs codons peuvent
correspondre à un même acide aminé), mais non ambigu
(à un codon donné correspond un seul acide aminé)
def Phase de lecture ouverte (ORF, Open Reading Frame) : séquence nucléotidique multiple de trois bases qui sépare deux
codons STOP (TAA, TAG et TGA).
Dans une phase ouverte de lecture, on retrouve une séquence
codante (CDS, Coding Sequence) qui débute par un codon
d’initiation de la traduction ATG et se termine un codon STOP.
Par abus de langage, la CDS est parfois appelée ORF.
En théorie, chaque séquence nucléotidique a 6 phases de
lecture possibles (3 pour chaque brin) dont une seule, sauf
exception, est ouverte et contient une séquence codante.
dans une phase ouverte (entre deux codons stop, jai trois cadre de lecture
Comment retrouver la CDS? entre deux codons top bcp de start et sequence assez longue:
Les genes font ils tous la meme taille ? NON Caractéristiques du génome humain
~ 20 000 gènes codant des protéines dont les exons
occupent plus de 1 % du génome.
Taille très variable : moins de 1 kb pour le gène SRY à
plus de 2 Mb pour les gènes DMD (2,220 Mb) et
RBFOX1 (2,473 Mb).


Qu’est ce que l’epissage alternatif?
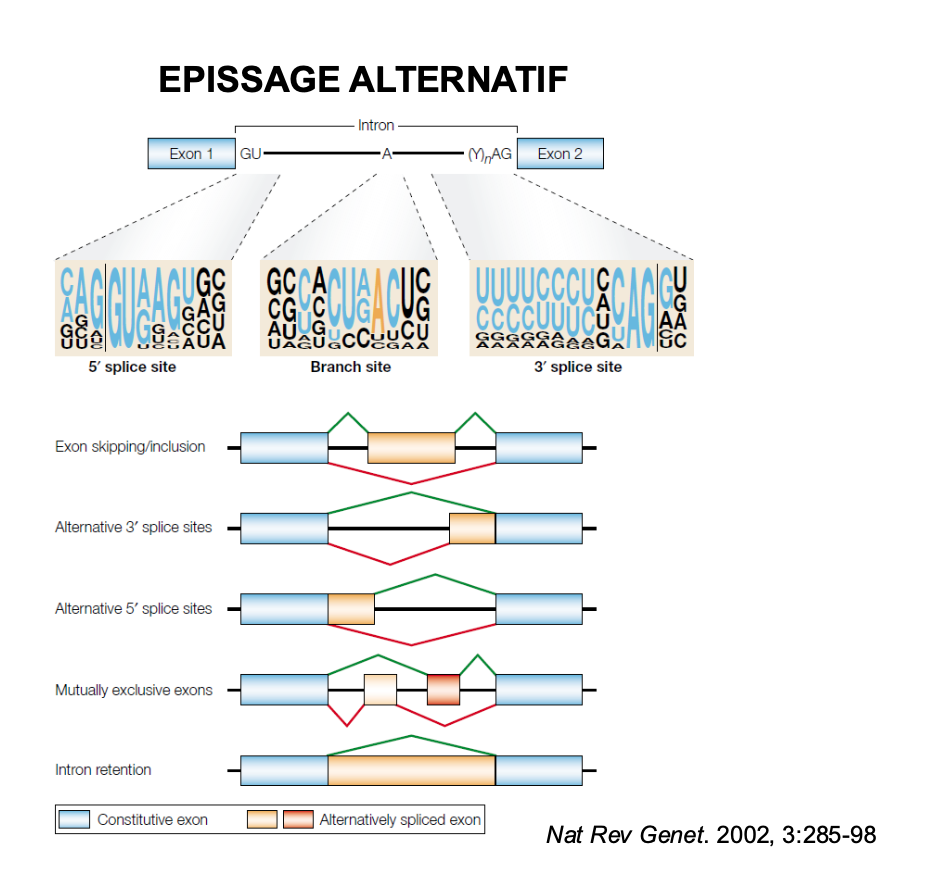
Quels sont les mecanisme de regulation de l’epissage alternatif?
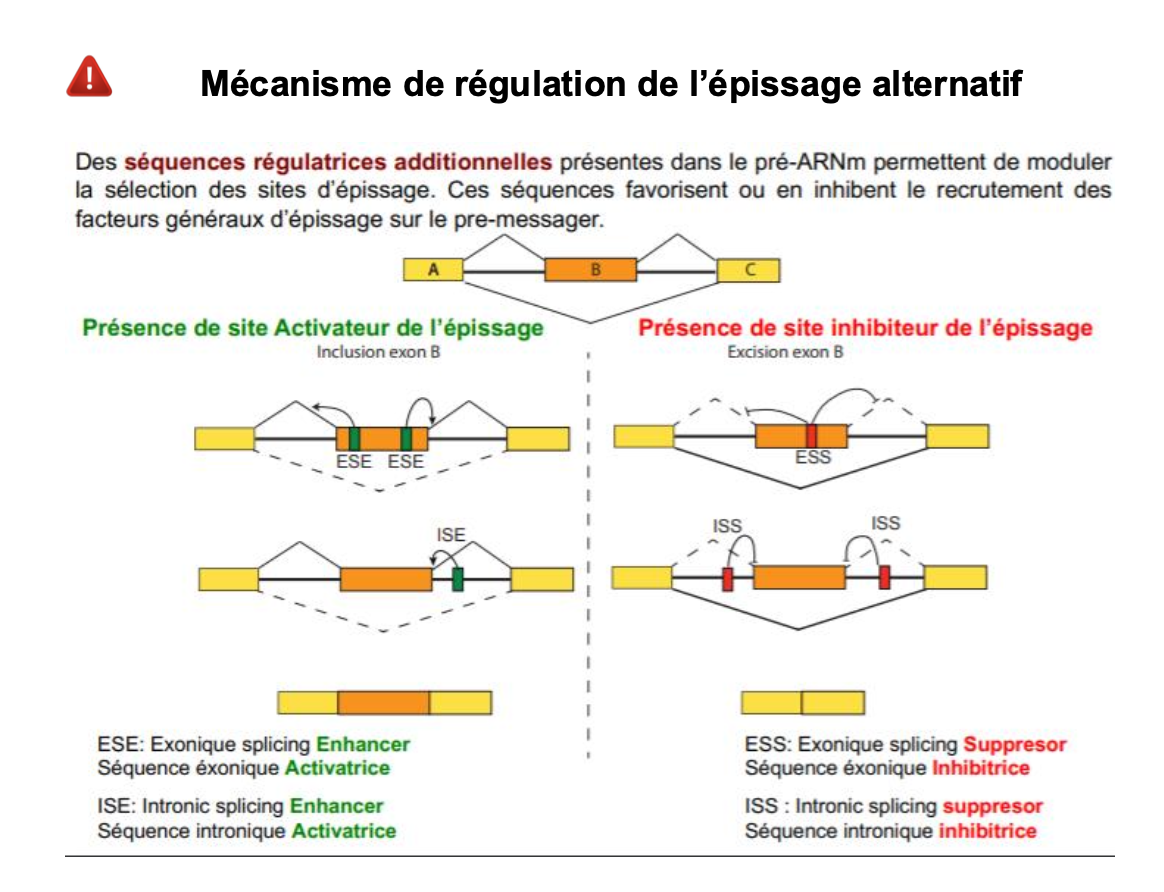
Quelles sont les proteines regulatrices de ce mecanisme? (prot SR:ESE, ISE / prot hnRNP) en gros ya des sequence regualtrice
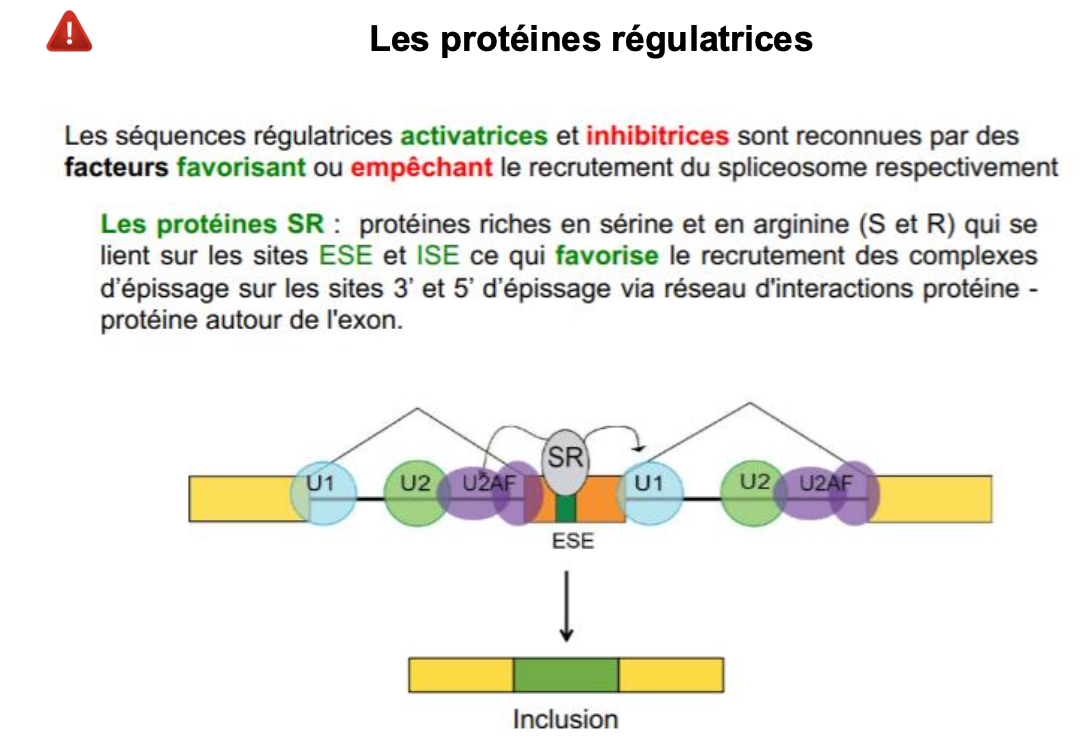
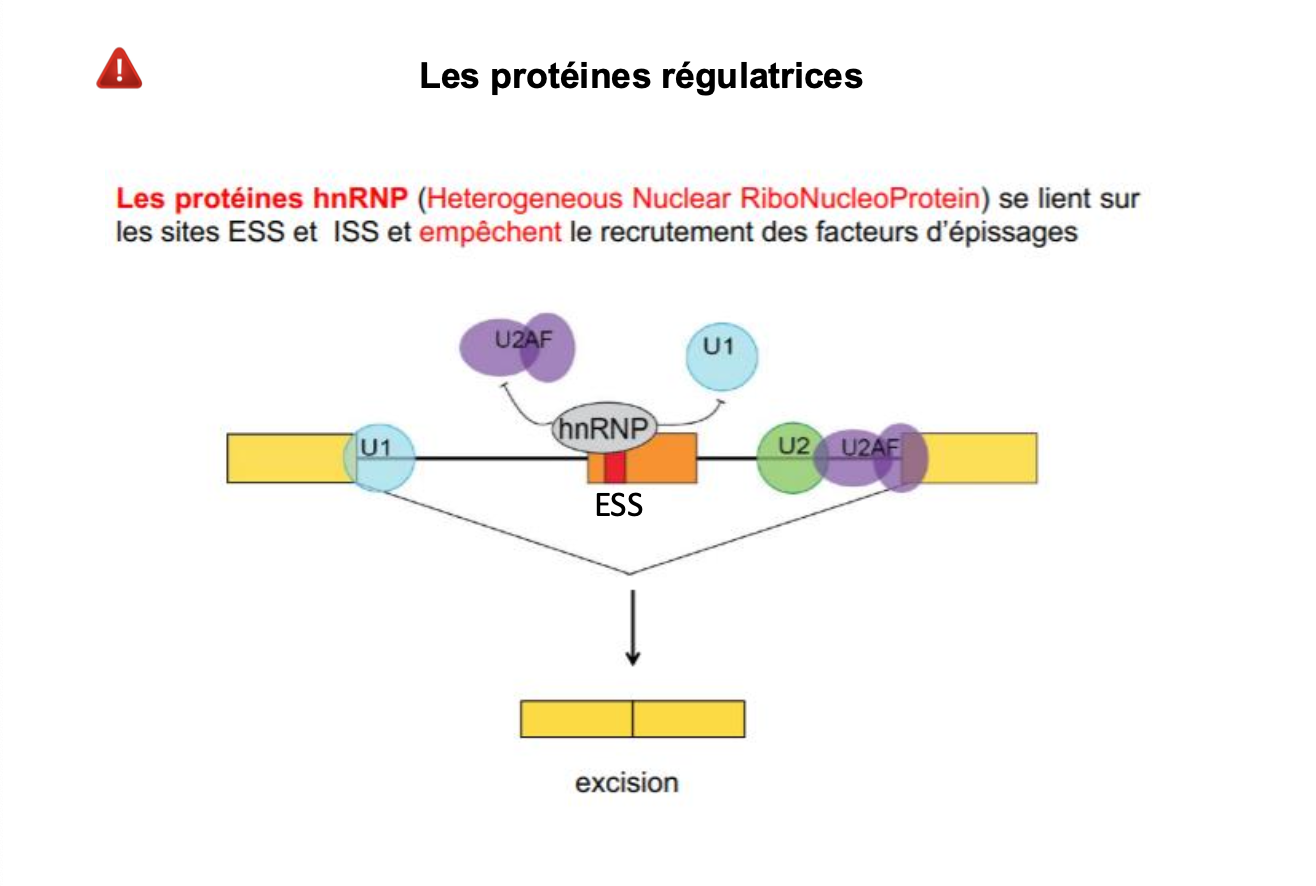
Les conséquences fonctionnelles des variations de séquence
nucléotidique siégeant dans des exons codants des gènes qui
codent les protéines ne doivent pas être interprétées en fonction
uniquement des conséquences sur la traduction des protéines
mais également en fonction des conséquences sur les motifs ESE
et ESS. Exemple : variation affectant la 3ème base d’un codon et qui par
conséquent ne change pas la nature de l’acide aminé codé peut
siéger dans un ESE et conduire à un saut d’exon par non
reconnaissance de l’exon. Les exons n’étant pas nécessairement
des multiples de trois bases, ce saut d’exon peut non seulement
déléter les acides aminés codés mais surtout introduire un
décalage du cadre de lecture.
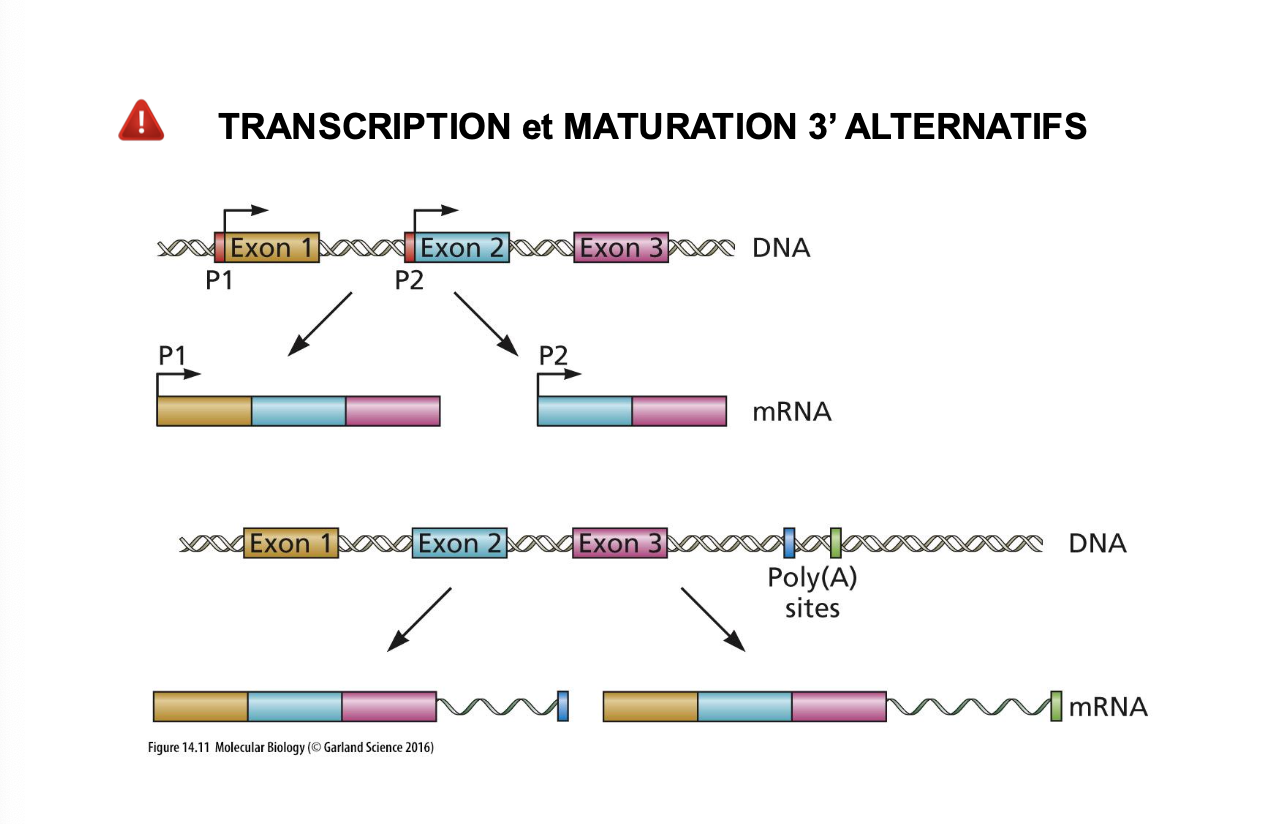
Quest ce quun promoteur alternatif? et des site de poly adenylations alternatif? pk c utile?
a apprenre ou a completer
6) Présentation des gènes produisant des ncRNA
Quest ce quun ?
ncRNA
mRNA : messenger RNA;
tRNA : transfert RNA;
rRNA : ribosomal RNA
sncRNA : small non coding RNA [
miRNA : microRNA,
siRNA : small interfering RNA,
piRNA : piwi-interacting RNA,
snRNA : small nuclear RNA;
snoRNA :
small nucleolar RNA]
lncRNA : long non coding RNA (smnt chez les euk)
Quel pourcentage de l’ARN est codant?
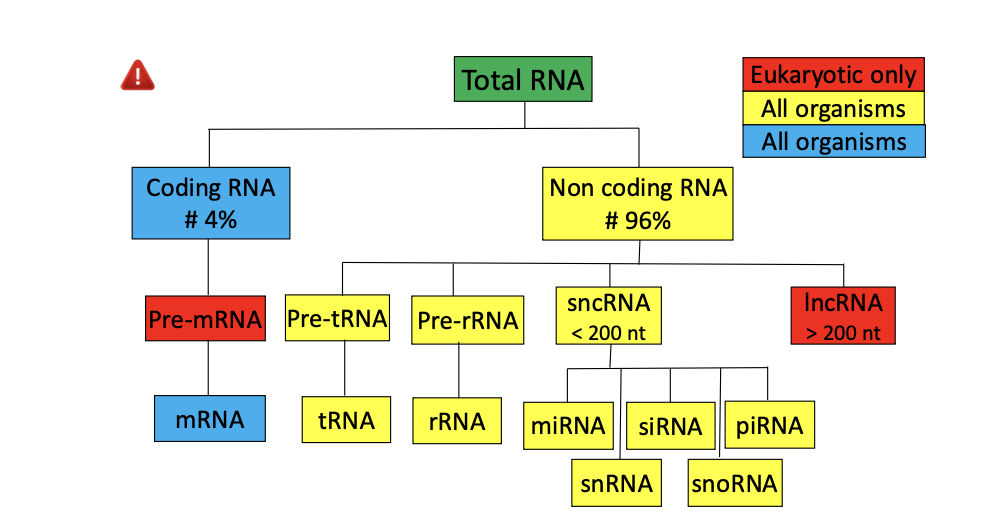
Plus de 25 000 gènes produisant des ARN non
codants
7) Présentation des pseudogènes
processed and unprocessed

Pseudogènes créés par rétrotransposition de l'ARNm d'un gène
parent fonctionnel codant pour une protéine.
- Parce que l'ADNc intégré ne possède pas les séquences
promotrices et régulatrices du gène parental, il reste généralement
silencieux sur le plan transcriptionnel, acquiert des mutations
délétères dans la séquence codante et dégénère en un processed
pseudogène ou un rétropseudogène.
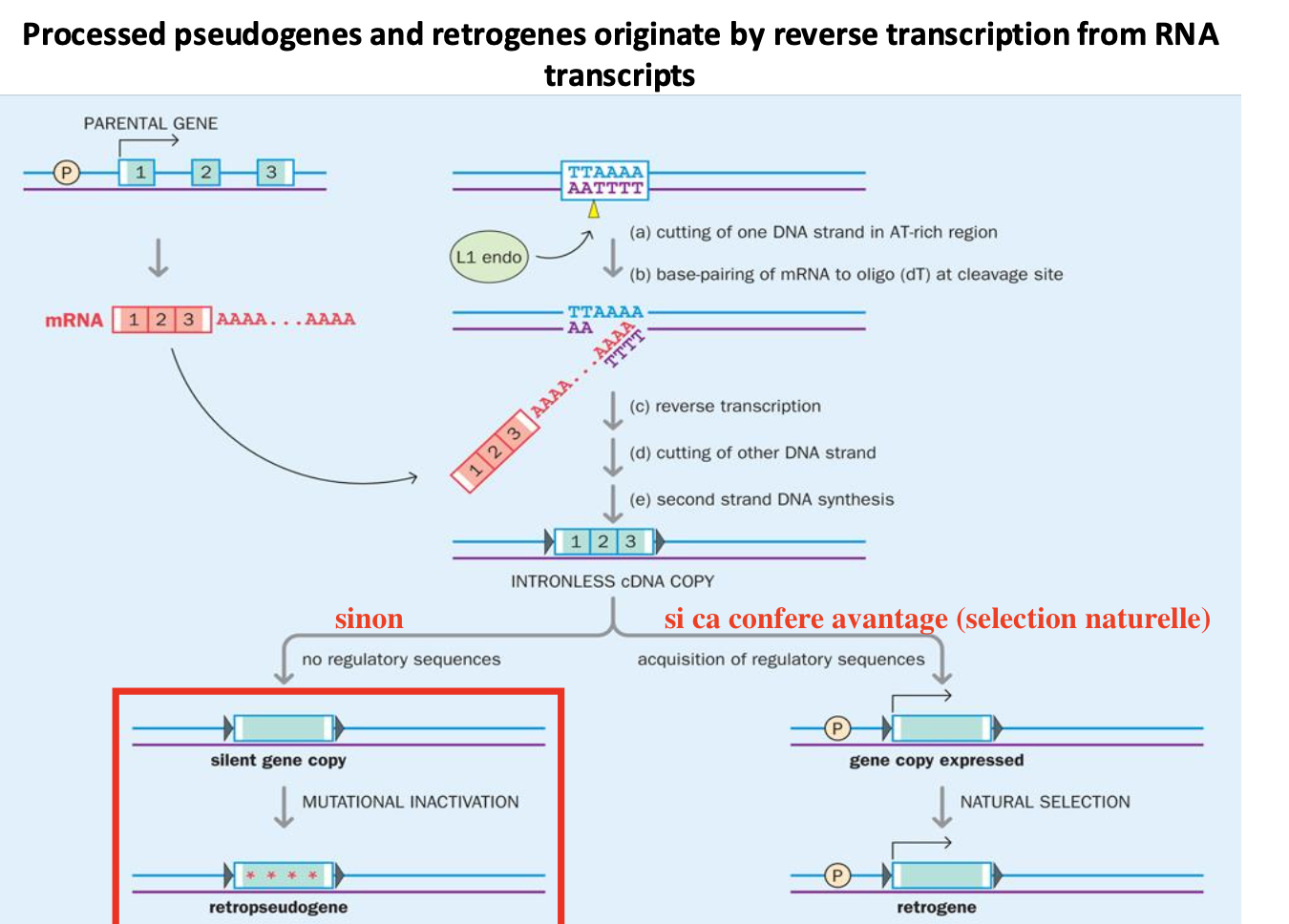
- Dans certains cas, la copie d'ADNc acquiert ou développe des
séquences régulatrices (par exemple, en l'insérant à proximité ou
dans un autre gène) qui lui permettent d'être transcrite. Si
l'expression de la copie d'ADNc est avantageuse, elle peut alors être
préservée par sélection naturelle en tant que rétrogène fonctionnel.
un autre type de pseudogene: PSEUDOGENES : Unprocessed or Duplicates pseudogenes
Pseudogène créé par duplication génomique d'un gène parent fonctionnel codant pour une protéine, suivie d'une accumulation de mutations invalidantes.
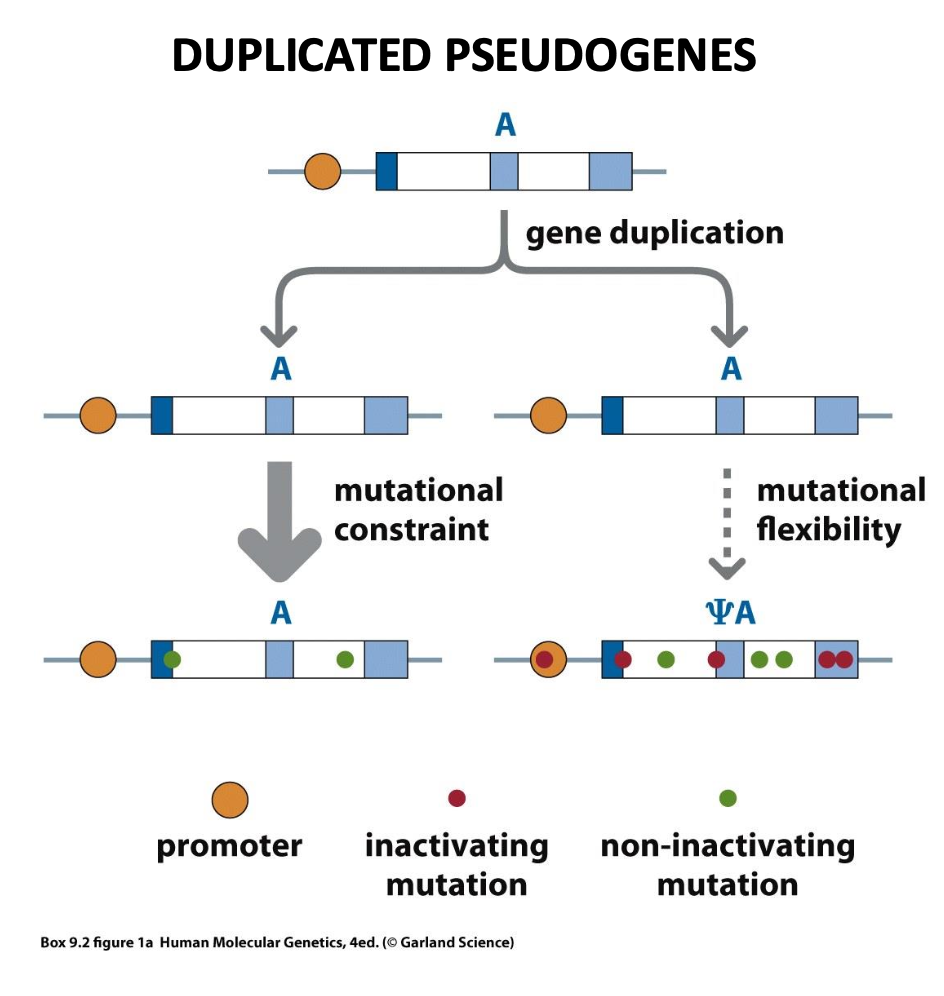
# 15 000 pseudogènes correspondant en majorité à
des retropseudogènes
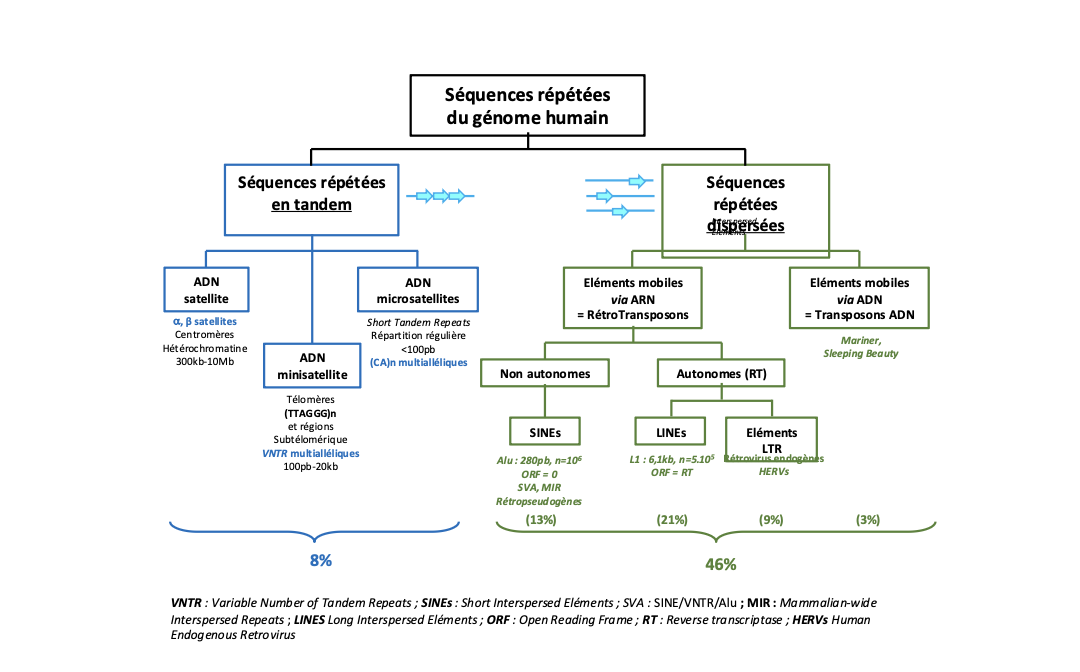
8) Présentation des séquences répétées :
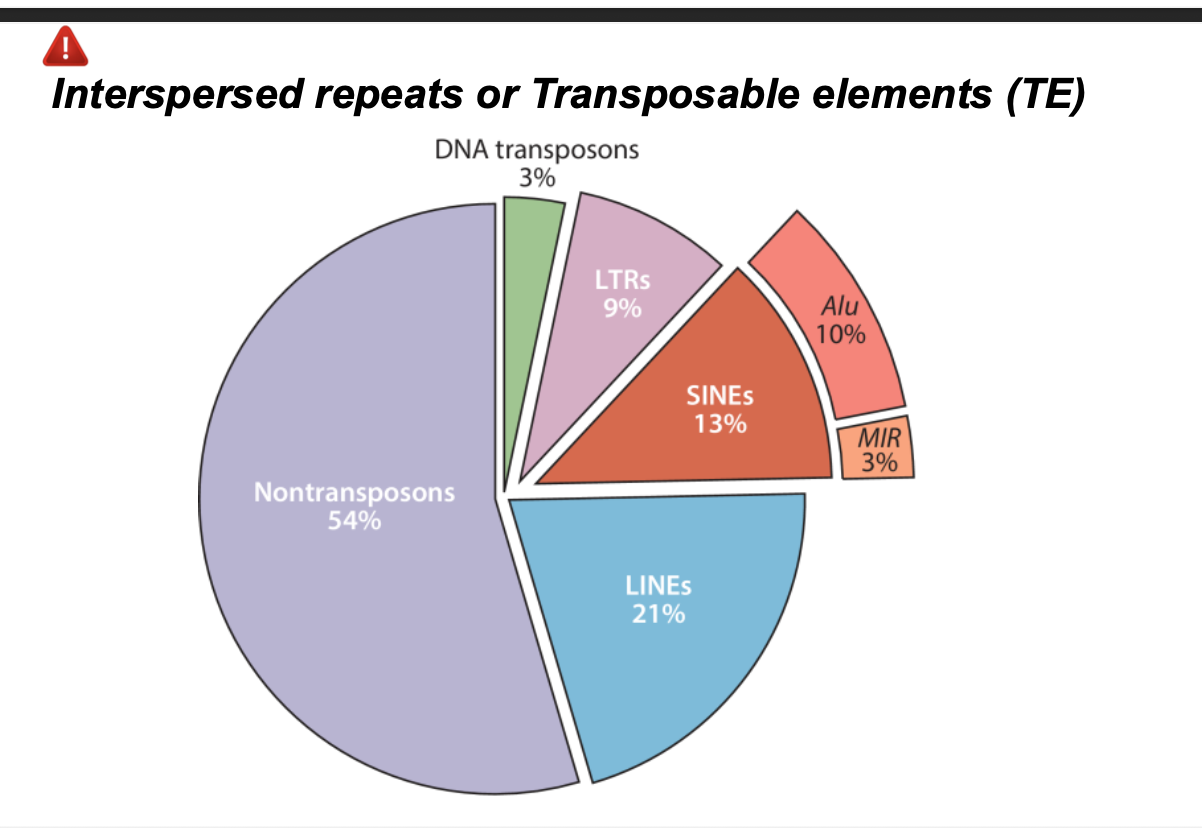
- Séquences répétées dispersées
Interspersed repeats or
Transposable elements (TE) are sequences that are repeated many times
and scattered throughout the genome.
Type 1 - DNA TRANSPOSONS
Type 2 - RETROTRANSPOSONS
Transposable elements: 46% du génome
1 405 625 kb
l’élement L1 est un LINEs connu.
Barbara McClintock?Prix Nobel de physiologie ou médecine en 1983 pour la découverte des « éléments génétiques mobiles »
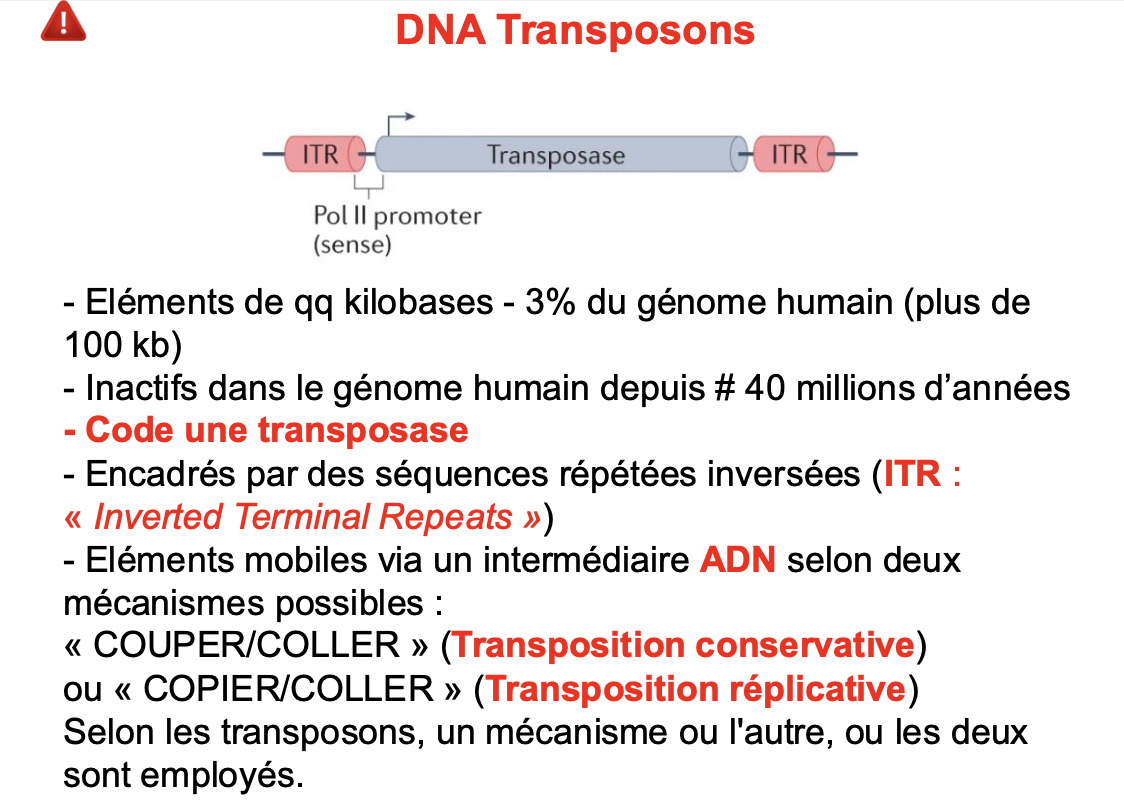
qu’est ce quune transposition conservative?
Type 2 - RETROTRANSPOSONS
- NON-LTR RETROTRANSPOSONS
• SINE : ALU, MIR, …
• SVA
• LINE : L1, L2, ….
- LTR RETROTRANSPOSONS : HERV
LTR: Une séquence terminale longue répétée (en anglais long terminal repeat ou LTR) est
une séquence nucléotidique caractéristique des extrémités des rétrovirus. Elle est
produite par transcription inverse de l'ARN du virus et se retrouve dupliquée aux deux
extrémités de l'ADN ainsi synthétisé. Elle participe à l'intégration de ces éléments viraux
dans le génome de l'hôte. Après intégration dans l'ADN, le génome proviral se retrouve
ainsi encadré par deux LTR, en 5' et en 3'. Le LTR 5' contient le promoteur qui assure la
transcription de l'ARN rétrovirus.
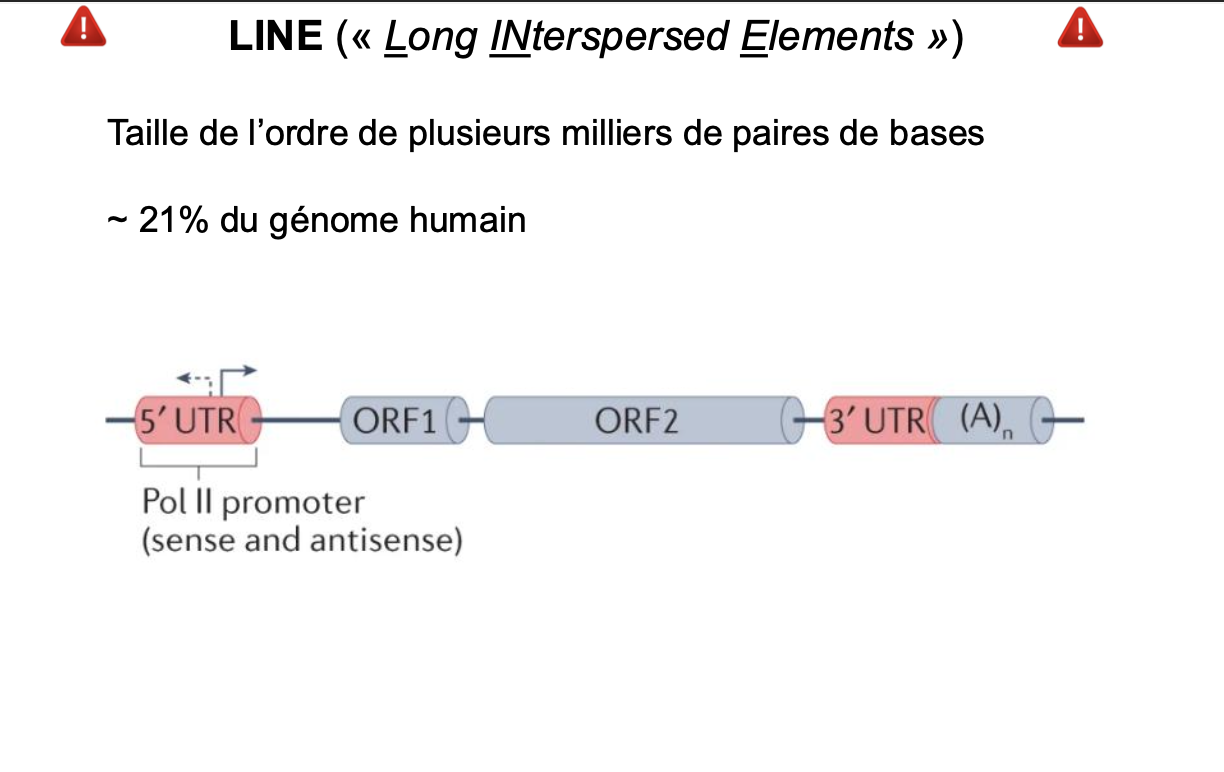
• ~ 500 000 copies, la plupart incomplètes et tronquées en 5’
• ~ 17% du génome humain – Régions AT riche – bandes G
• 40 à 50 éléments actifs (6 hot L1s)
• Spécifiques des mammifères – actifs depuis ~150 millions
d’années
- ORF1 et 2 communes aux mammifères et l’ORF2 code une
reverse transcriptase
- ORF0 antisens spécifique des primates

joue un role dan sla diversité genetique des neurones
Les éléments transposables :
- Contribuent au polymorphisme du génome humain (présence
ou absence) et font partie des variants structuraux (MEI :
Mobile Element Insertion).
- Constituent un élément majeur de l’évolution des génomes.
- Participent à la régulation des l’expression des gènes.
8) Présentation des séquences répétées :
- Séquences répétées en tandem
Tandem repeats are
sequences that are repeated many times adjacent to each other
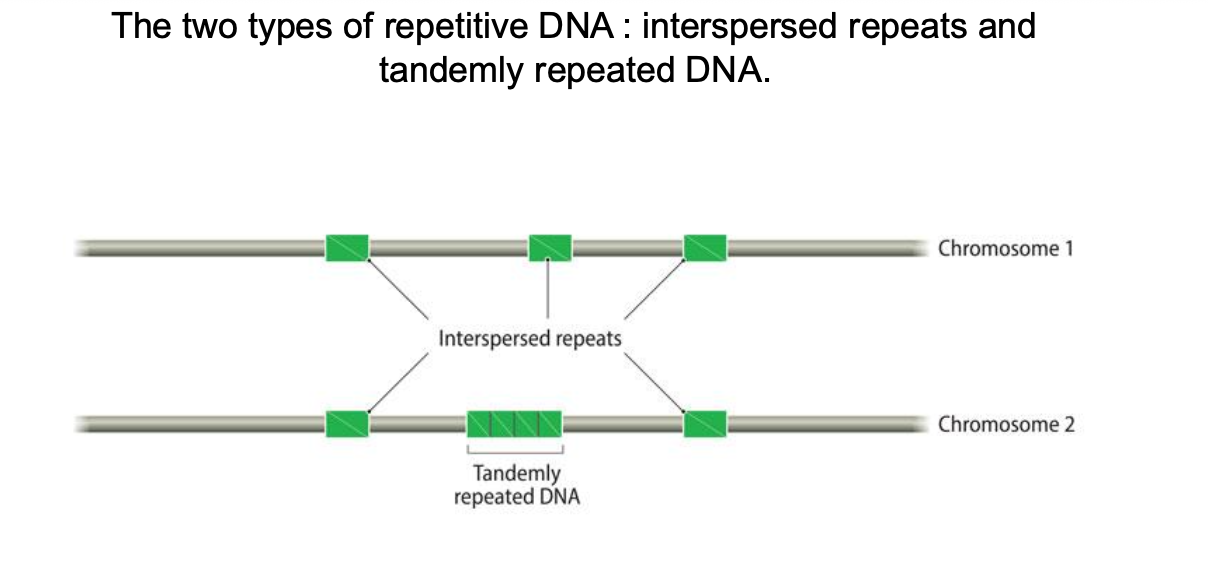
Séquences répétées le plus souvent concentrées dans des
régions chromosomiques (télomères, centromères et régions
sub-télomériques).
Selon la taille de l’unité de répétition et la taille de la région
concernée on distingue :
• ADN satellite (# 150 Mb)
• ADN minisatellites (VNTRs = variable number tandem repeats)
• ADN microsatellites
- STRs (short tandem repeats)
- Télomères : (TTAGGG)n.
~ 54% du génome est constitué de séquences
répétées représentées en majorité par des éléments
transposables (SINEs, LINEs, HERV).
bilan a completer
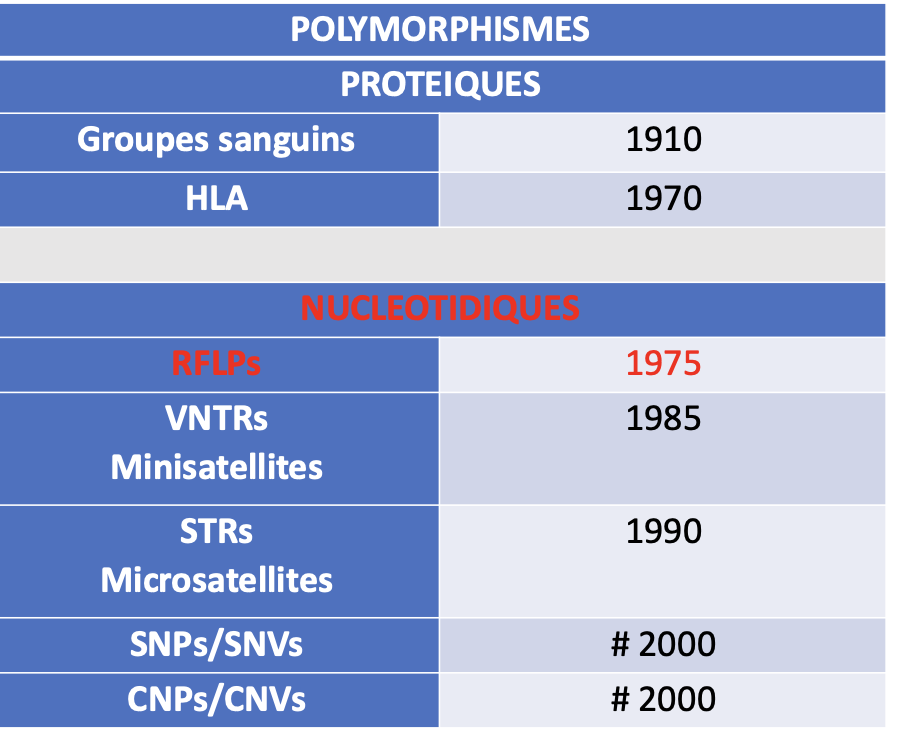
9) Présentation des polymorphismes
def polymorphisme: Le concept de polymorphisme génétique (du grec « poly » plusieurs et « morphê » forme) désigne la coexistence de plusieurs allèles pour un gène ou un locus donnés, dans une population donnée.
Ces polymorphismes peuvent être explorés par analyse :
- des caractères (polymorphismes phénotypiques)
- du produit protéique du gène (polymorphismes protéiques)
- des chromosomes (polymorphismes chromosomiques)
- de l’ADN (polymorphismes nucléotidiques)
Les polymorphismes chromosomiques et nucléotidiques n’ont
pas besoin de siéger dans des séquences codantes pour être
détectés.
RFLPs : Restriction Fragment Length Polymorphisms
iable Number of Tandem Repeats)
• Séquences répétées en tandem d’une unité de taille de l’ordre
d’une dizaine ou quelques dizaines de nucléotides et
s’étendant sur une distance comprise entre 100 bp à 20 kb
• Principalement localisées dans les régions télomériques et
sub-télomériques
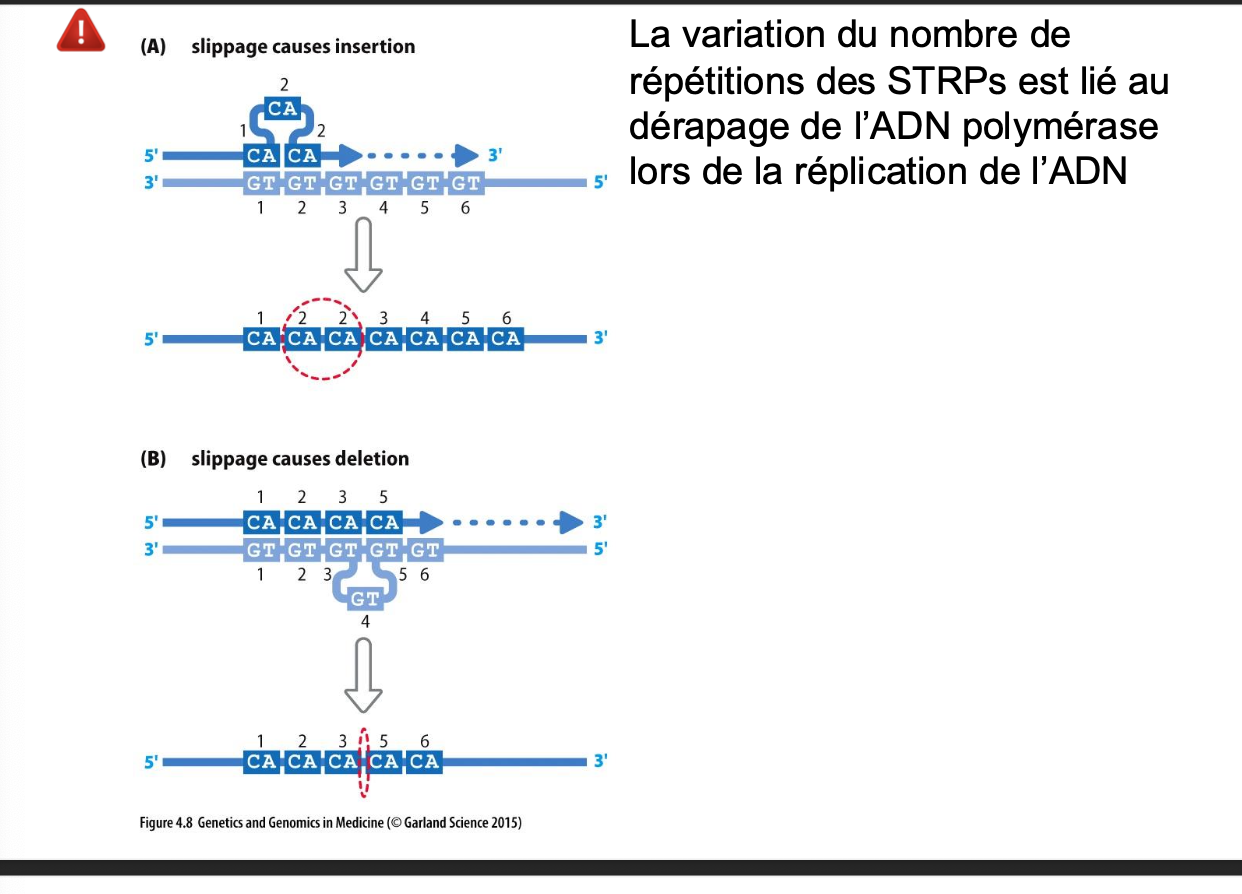
• Lorsque le nombre de répétions est suffisant, le STR est le
siège d’un polymorphisme multi-allélique (STRPs)
• Les STRPs dont l’unité de répétition n’est pas un multiple de 3
nucléotides sont absents des exons codants des gènes
codant les protéines
Exemples de microsatellites :
- Répétitions mononucléotidiques :
(A)n, (T)n, (G)n ou (C)n
- Répétitions dinucléotidiques :
(CA)n : 27.7
- Répétitions trinucléotidiques :
(AAT)n
Comment ca c former?
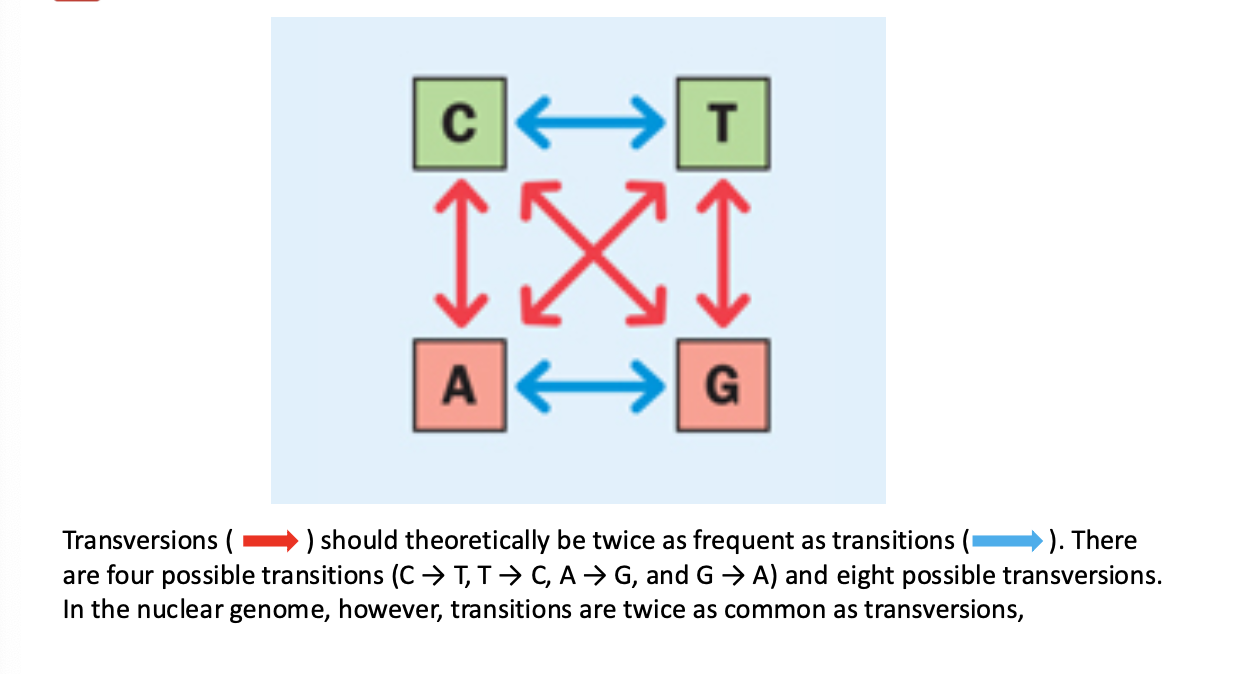
SNPs (Single Nucleotide Polymorphisms)
• Substitutions nucléotidiques : transitions sont plus
fréquentes que les transversions
• Polymorphismes le plus souvent bi-alléliques présents dans
au moins 1% de la population
Fréquence allélique de l’allèle le plus rare (MAF pour Minor
Allele Frequency) ≥ 1%
MAF?
• Analysables individuellement (PCR) ou plus globalement
(puces à ADN ou NGS)
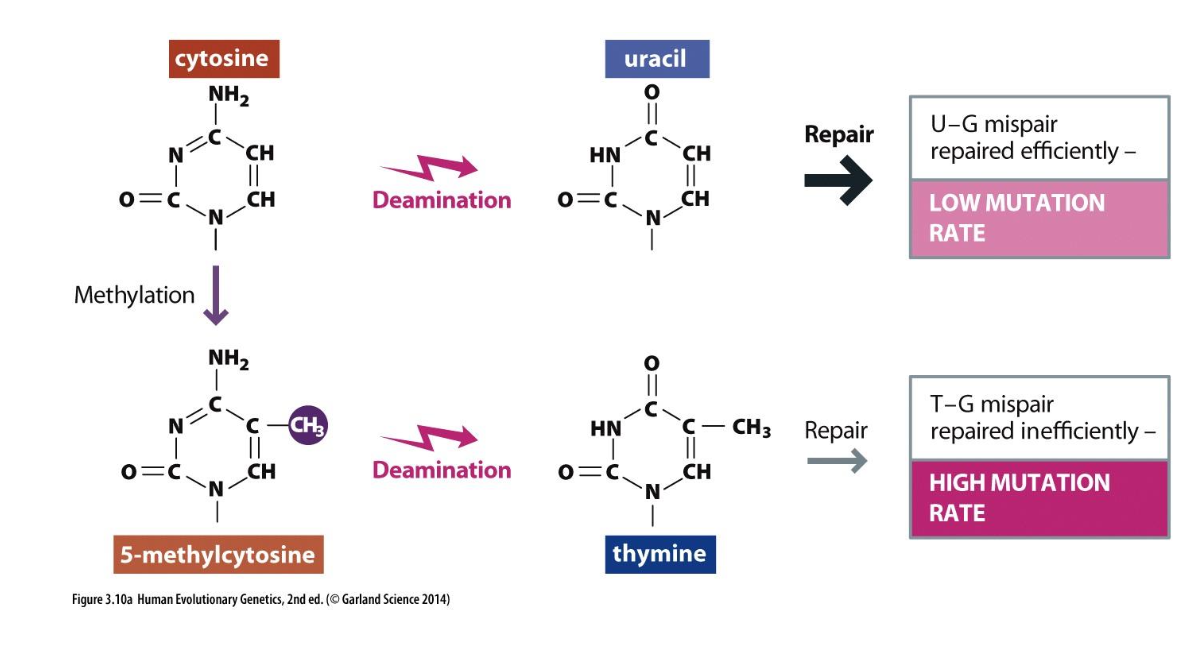
HAPLOTYPES
Définition : succession des allèles d’un gène ou d’un locus
sur une région chromosomique de petite taille.
Du fait de l’absence de recombinaison, les associations d’allèles
de plusieurs polymorphismes très proches sont transmis
généralement en bloc, comme un seul marqueur.
DIFFERENCE GENOTYPE ET HAPLOTYPE

Structural Variants (SVs)
En fonction de la taille de la variation structurale on distingue :
• Les INDELS qui correspondent à des insertions/délétions de
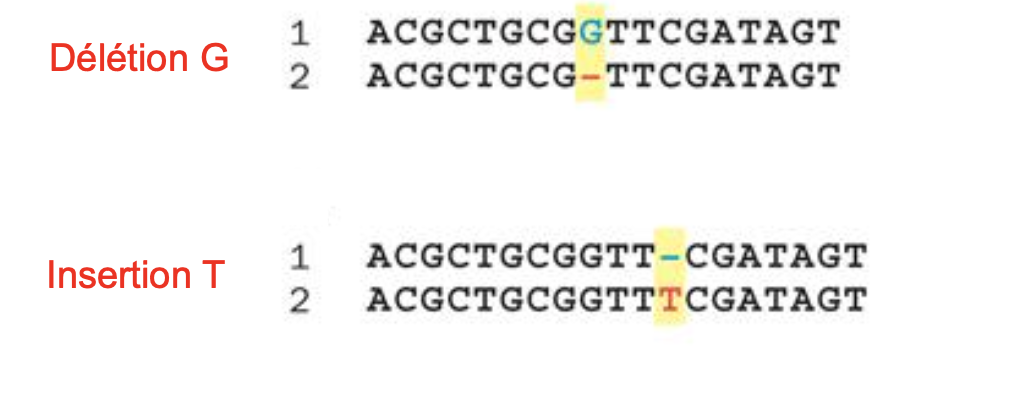
moins de 50 nucléotides (1 à 49)
• Les autres SV correspondent à des variations de plus de 50
nucléotides
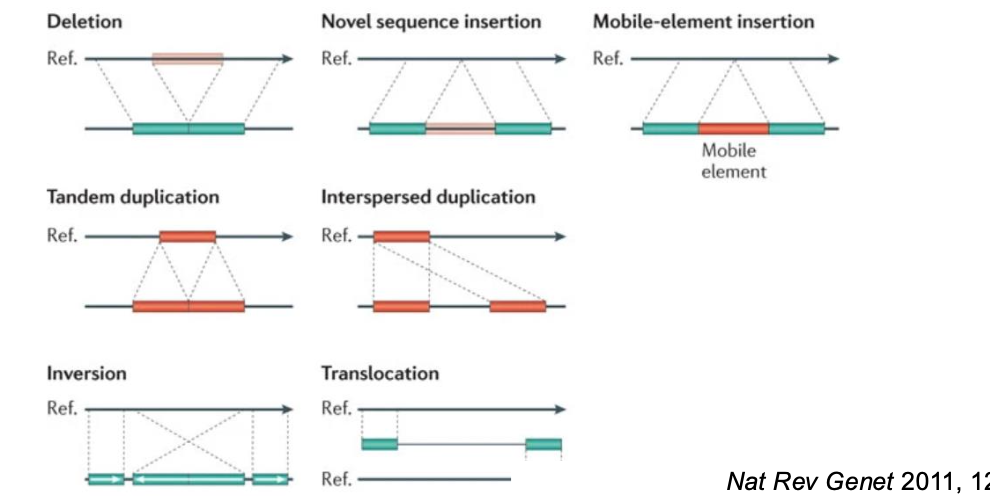
Les classes mutationnelles de SV peuvent être subdivisées en deux groupes :
• Les SV déséquilibrés (Unbalanced SVs) sont des réarrangements qui entraînent des gains ou
des pertes d'ADN génomique (également appelés variantes du nombre de copies, ou CNV).
•Les SV équilibrés (Balanced SVs) sont des réarrangements qui n'impliquent pas de modification
du nombre de copies
De plus, chaque SV peut être décrit comme canonique ou complexe :
Les SV canoniques sont des réarrangements qui impliquent une seule signature de point de
cassure et/ou des modifications du nombre de copie.
Les SV complexes sont des réarrangements qui impliquent au moins deux signatures de points
de cassures et/ou des modifications du numéro de copie.