Chapitre 3 : Introduction à l'épigénétique
1/146
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
147 Terms
1. Concept de l’épigénétique
Qu’étudie l’épigénétique ?
Les mécanismes moléculaires modulant l’expression du patrimoine génétique selon le contexte.
Quels niveaux peuvent être modifiés en épigénétique ?
Protéines interagissant avec l’ADN (histones).
Molécule d'ADN elle-même.
Où se trouve l’hétérochromatine et comment est-elle ?
En périphérie du noyau/nucléole.
Chromatine condensée.
Où se trouve l’euchromatine et comment est-elle ?
Localisation intranucléaire (centre).
ADN décompacté, lâche.
Qu’étudie la génétique ?
Les gènes (séquence ADN, code génétique, régulation).
Les séquences ADN sont-elles stables ?
Oui, relativement stables et transmises entre générations.
Qu’étudie l’épigénétique par rapport à la génétique ?
Modifications réversibles régulant l’expression des gènes sans changer la séquence.
Quelles influences peuvent modifier l’épigénome ?
Environnement, alimentation, stress, vieillissement.
Quelle est la principale marque épigénétique ?
La méthylation des cytosines qui correspond à l’ajout d’un groupement méthyl (CH3) sur certaines cytosines→ 5-méthylcytosine.
Quelle est la « cinquième lettre » symbolique du génome ?
La 5-méthylcytosine.
Que permet la 5-méthylcytosine ?
Régulation de l’expression génétique (activation ou inhibition selon contexte).
Relation génétique/épigénétique en une phrase ?
La génétique écrit le texte, l’épigénétique contrôle la lecture.
Pourquoi les jumeaux monozygotes peuvent différer phénotypiquement ?
Différences épigénétiques même s’ils partagent le même genre génotype
Que montrent les études de méthylation et acétylation chez les jumeaux ?
Jeunes jumeaux : épigénome similaire.
Jumeaux âgés : fortes différences de quantité et répartitions des 5-mC et d’histones acétylées.
Que provoquent ces différences avec l’âge ?
Modifications d’expression génique → différences phénotypiques.
2. Remodelage de la chromatine
Pourquoi l’empaquetage de l’ADN est-il essentiel ?
Organisation et stabilité cellulaire
Compacter des longues molécules d’ADN dans un espace restreint mais aussi d’éviter qu’elles ne s’emmêlent ou ne forment des noeuds.
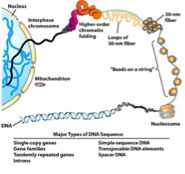
Quelles protéines organisent l’ADN en chromatine ?
Les histones.
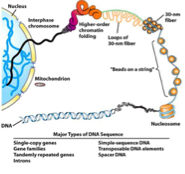
Quelle est l’unité fondamentale de la chromatine ?
Le nucléosome.
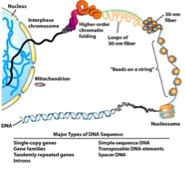
De quoi est composé un nucléosome ?
≈146 pb d’ADN, enroulé autour d’un centre protéique composés d’histone
→ Ce coeur est un octamère constitué de deux copies de chaque histone H2A, H2B, H3 et H4
→ Les histones sont des protéines basiques hautement conservées au cours de l’évolution
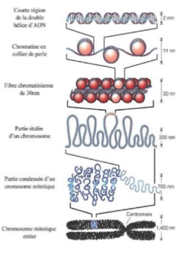
Comment s’appelle structure en “collier de perles” ?
Filament nucléosomique (10 nm).
→ Les nucléosomes s’enchaînent le long de l’ADN
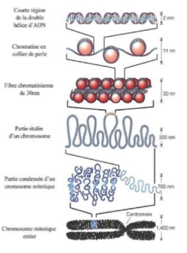
Quel est le rôle de l’histone H1 ?
Compaction supplémentaire → fibre de 30 nm =unité de base de la chromatine
→ Finalement, la chromatine va continuer de se condenser à l’aide de plusieurs protéines de structures pour former un chromosome.
Que provoque la compaction de la chromatine ?
Réduction de l’accessibilité à l’ADN pour les facteurs de transcription.
Que contient un nucléosome ?
Un noyau protéique de 8 histones.
Comment obtient-on un nucléosome isolé en laboratoire ?
Digestion de l’ADN linker par une endonucléase.
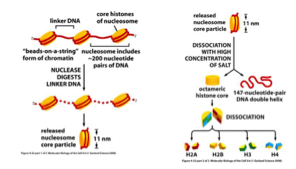
Combien de fois l’ADN s’enroule autour du noyau protéique du nucléosome ?
1,7 tour.
Quelle est la longueur d’ADN enroulée autour du nucléosome ?
147 paires de bases.
De quoi dépend l’expression d’un gène ?
De la structure de la chromatine au niveau du promoteur et des régions voisines.
Comment les nucléosomes influencent-ils la transcription ?
Ils bloquent l’accès des facteurs de transcription et de l’ARN polymérase.
Un gène enroulé autour de nucléosomes peut-il être activé par simple ajout de facteurs de transcription ?
Non, l’accessibilité physique est nécessaire.
Qu’est-ce qui est requis pour transcription et réplication ?
Un remaniement dynamique de la chromatine.
Comment rend-on un gène accessible ?
En écartant les octamères d’histones de l’ADN.
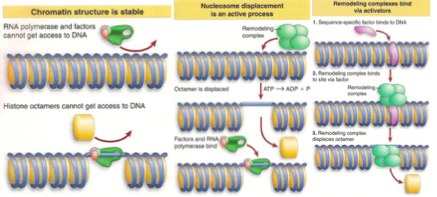
Un complexe de remodelage a-t-il une spécificité de site ?
Non, il n’a pas de spécificité propre. Il doit cependant être recruté par un élément de l’appareil de transcription

Que devient ce facteur après recrutement ?
Il peut être libéré une fois que le complexe s’est fixé.
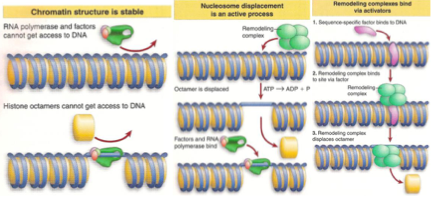
Quelles sont les différentes modifications ?
Glissement de nucléosomes qui permet de changer la position des séquences d’ADN
Un déplacement au niveau des nucléosomes pour avoir une région accessible plus grande (augmentation de l’espace internucléosomique)
Création d’un trou permettant l’accès direct à l’ADN grâce à l’exclusion temporaire d’un nucléosome
Méthylation et hydroxyméthylation de l’ADN
Qu’est-ce que la méthylation de l’ADN ?
Ajout d’un groupement méthyle sur la cytosine en position 5 dans un dinucléotide CpG.
Quelle enzyme réalise la méthylation ?
Les ADN méthyltransférases (DNMTs).
Que permet la 5-méthylcytosine en termes de reconnaissance ?
Recrutement des MBPs (methyl-CpG binding proteins).
→ Compaction de la chromatine
Impact de la méthylation d’un promoteur ?
Empêche la liaison des facteurs de transcription → inhibition de l’expression.
Où se trouve principalement la 5-méthylcytosine chez les mammifères ?
Sur les dinucléotides CG.
→ Pas dans les contextes CC, CA ou CT car seule la configuration CpG est symétrique sur les brins antiparallèles de l’ADN et permet un maintien fidèle.
→ les deux brins de la double hélice ont une polarité opposée.
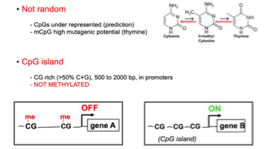
Pourquoi la symétrie du CpG est-elle essentielle ?
Elle facilite la copie exacte du motif méthylé lors de la réplication.
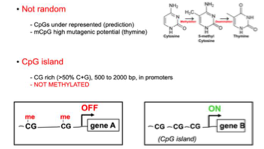
Quel est le rôle de la réplication dans le maintien de la méthylation ?
Elle préserve la symétrie CpG en rétablissant les motifs méthylés sur le brin néo-synthétisé.
Quel est le rôle des DNMTs ?
Aussi appelées Writers
Ajouter un groupement méthyle sur la cytosine → formation de 5mC.
Établir ou entretenir les marques de méthylation.
Quelles DNMT réalisent la méthylation de novo ?
DNMT3A et DNMT3B.
→ C’est à dire la mise en place de nouvelles marques de méthylation durant le développement embryonnaire
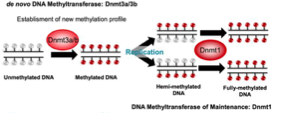
Quelle enzyme maintient les marques après la réplication ?
DNMT1.
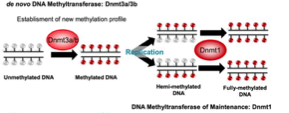
Comment agit DNMT1 ?
Copie la méthylation du brin parent sur le brin fils.
Qu’est-ce qu’un “reader” de méthylation ?
Protéine reconnaissant et se liant spécifiquement les CpG méthylés.
→ protéines MBD (methylated-bindings domains)
Exemple de reader ?
MeCP2.
Rôle des MBD ?
Servir de plateforme de recrutement pour :
Protéines compactant la chromatine
Complexes réprimant la transcription.
Quel est le rôle des TET (readers) ?
Déméthylation de l’ADN.
Remarque : TET = ten eleven translocation
Pourquoi la déméthylation est-elle importante ?
Permet de réactiver des gènes silencieux.
4. Le code histone
Taille typique d’une histone ?
102–135 acides aminés conservés au cours de l’évolution.
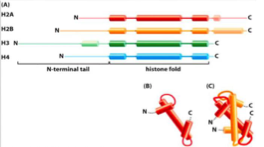
Structure commune ?
3 hélices alpha reliées par 2 boucles.
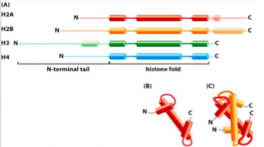
Quelle partie des histones est modifiable ?
Les queues N-terminales, très longues.
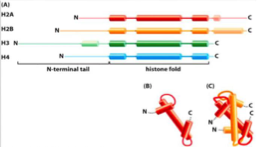
Quelles extensions dépassent du nucléosome ?
N-terminales : 19–39 aa pour toutes les histones du cœur
C-terminales : présentes sur H2A et H2B.
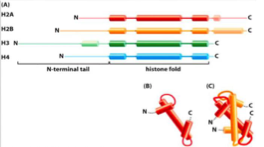
Que forme les histones ?
Les histones H2A avec H2B et H3 avec H4 forment des dimères par une interaction appelée « poignée de main ».
Comment les histones se lient-elles à l’ADN ?
Via interactions électrostatiques : le squelette des acides aminées des histones (+) ↔ le squelette phosphodiester de l’ADN (-).
Quels acides aminés sont cruciaux pour ces interactions ?
Arginine (R) et Lysine (K) (acides aminés basiques).
Ils représentent ~20 % des histones.
Pourquoi sont-ils importants pour l’épigénétique ?
Ils constituent les sites de modifications post-traductionnelles (acétylation, méthylation…).
Quels dimères d’histones se forment par « serre-main » ?
H2A–H2B
H3–H4
Existe-t-il des variants d’histones ?
Oui, leur séquence diffère légèrement des histones classiques.
Ils apparaissent lors de modifications de l’ADN (ex. dommages, irradiation)
H2AX remplace quelle histone classique ?
H2A, incorporé en faible quantité dans la chromatine.
Quand H2AX est-elle phosphorylée ?
Lors de cassures double brin de l’ADN.
Rôle de H2AX phosphorylée ?
Participation aux processus de réparation de l’ADN probablement par recrutement des enzymes de réparation.
Indicateur de l’état de réparation de l’ADN.
Que voit-on après réparation ?
H2AX → revient à H2A.
Quel variant d’histone remplace H3 au centromère ?
CENP-A.
Rôle du centromère avec CENP-A ?
Liaison des microtubules lors de la mitose.
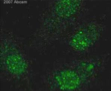
Que montrent les points fluorescents dans une immunofluorescence anti-H2AX phosphorylée ?
Les sites de cassure double brin de l’ADN.
L’accumulation de H2AX phosphorylée aux points de dommage.
Modifications post-traductionnelles des histones
Où se produisent les modifications post-traductionnelles des histones ?
Sur les queues N-terminales des histones.
Pourquoi ces modifications sont-elles importantes ?
Elles régulent la condensation de la chromatine et donc l’accès des facteurs de transcription à l’ADN.
→ En fonction de leur organisation → l’ADN devient plus ou moins accesible
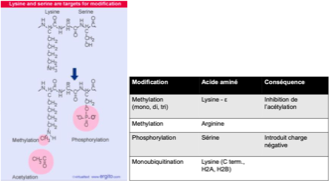
Effet de la méthylation sur la chromatine ?
Elle compacte l’ADN.
Nombre de niveaux de méthylation possibles ?
Mono-, di- ou triméthylation, chacune avec un effet distinct sur la structure de la chromatine et l’expression des gènes.
Quels résidus subissent un acétylation ?
Lysine ou arginine.
Conséquence de l’acétylation ?
Suppression de charges positives
Relâchement de l’ADN (moins compact)
Quels résidus peuvent être phosphorylés ?
Sérine (S), thréonine (T), tyrosine (Y — plus rare).
Conséquence d’une phosphorylation ?
Apparition d’une charge négative
Peut activer ou désactiver l’activité transcriptionnelle selon le contexte
Quel acide aminé est le plus fréquemment modifié sur les histones ?
La lysine (K).
Quel type d’histone est particulièrement modifié et pourquoi ?
Histone H3.
Sa queue N-terminale est :
longue,
flexible,
extrêmement accessible → cible privilégiée des modifications.
Une lysine peut-elle être méthylée et acétylée en même temps ?
Non, ces modifications sont mutuellement exclusives.
Les modifications C-terminales ont-elles un rôle important ?
Leur impact est moins important que celles du N-terminal.
Combien de sites de méthylation sont connus sur la queue N-terminale de H3 ?
8 sites de méthylation connus.
→ Ils jouent un rôle clé dans la régulation de la chromatine et de l’expression génique
Acétylation des histones
Quels acides aminés sont acétylés sur les histones ?
Les lysines (K) des queues N-terminales des histones.
Quelle est la conséquence chimique de l’acétylation d’une lysine ?
L’acétylation neutralise la charge positive du groupement ε-amino de la lysine.
Quel est l’impact de cette neutralisation sur la chromatine ?
Cela influence la compaction de la chromatine, en favorisant son relâchement.
Quel résidu d’histone est crucial pour la formation de la fibre de 30 nm ?
La lysine 16 de l’histone H4 (H4K16).
Que provoque l’acétylation de H4K16 ?
Elle relâche la chromatine et lui donne une conformation de « collier de perles », compatible avec transcription et réplication.
Que montre le traitement de noyaux avec de la DNase I concernant l’acétylation ?
La chromatine acétylée est plus sensible aux endonucléases.
Comment s’appellent les enzymes qui acétylent les histones ?
Les histone acétyltransférases (HATs).
Quel est leur rôle principal ?
Elles sont essentielles pour l’activation de la transcription en relâchant la chromatine.
Citer un coactivateur possédant une activité HAT.
CBP/p300 ou PCAF
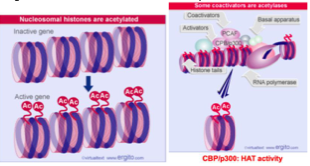
Dans quels contextes l’acétylation des histones se produit-elle ?
Pendant la réplication de l’ADN — acétylation transitoire aidant à la réorganisation de la chromatine sur les nouveaux brins.
Lors de l’activation de l’expression génique — elle facilite le relâchement de la chromatine, rendant les gènes accessibles aux facteurs de transcription.
Quelles histones sont acétylées dans la chromatine active ?
Les queues H3 et H4.
Quelle modification caractérise la chromatine inactive (hétérochromatine) ?
La méthylation de H3K9.
La méthylation des cytosine des doublets CpG
Qu’est-ce qu’un doublet CpG ?
Une cytosine liée via liaison phosphodiester à une guanine sur le même brin d’ADN.
70 à 80% des CpG sont méthylés, ce qui inactive le gène
La méthylation compacte-t-elle l’ADN ?
Oui, généralement, mais ce n’est pas une règle absolue.