Moleculaire biologie H5
1/17
Earn XP
Description and Tags
Organisatie van het humane DNA
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
18 Terms
Beschrijf de samenstelling van het humaan genoom. Wat is het aandeel coderend DNA en wat is het aandeel repetitief DNA?
🧬 1. Algemene opbouw van het humaan genoom
Het humane genoom bestaat uit ongeveer 3,2 miljard basenparen, verdeeld over:
22 autosomen + 2 geslachtschromosomen (XX of XY)
Een klein circulair genoom in de mitochondriën (mtDNA)
Het DNA zit verpakt in chromatine, onderverdeeld in euchromatine (actief) en heterochromatine (inactief).
🧠 2. Aandeel coderend vs. niet-coderend DNA
Slechts ~1 à 2% van het humane genoom codeert voor eiwitten (= exonen).
De overige ~98–99% is niet-coderend DNA, dat o.a. bestaat uit:
Regulatoire sequenties (promotors, enhancers, silencers)
Introns binnen genen
Repetitief DNA
Pseudogenen
Transposon-gerelateerde elementen
Niet-coderende RNA-genen (bv. tRNA, miRNA, lncRNA)
🔁 3. Repetitief DNA – grote fractie van het genoom
Ongeveer 50% van het genoom bestaat uit repetitieve sequenties, waaronder:
LINEs (Long Interspersed Nuclear Elements)
SINEs (Short Interspersed Nuclear Elements, zoals Alu-elementen)
Satelliet-DNA (herhalingen in centromeren en telomeren)
Sommige repetitieve sequenties kunnen regulatoir zijn of bijdragen aan genoomplasticiteit, maar veel hebben onbekende of beperkte functie.
📌 Belangrijke cijfers samengevat
Categorie | % van genoom | Voorbeeld |
|---|---|---|
Coderend DNA (exonen) | 1–2% | CFTR, BRCA1 |
Introns + regulatoire DNA | 25–30% | Promotors, enhancers |
Repetitief DNA | ~50% | LINEs, SINEs, satelliet-DNA |
Overig niet-coderend DNA | 20%+ | Pseudogenen, ncRNA-genen, duplicaties |
Wat is het verschil tussen intragenisch en intergenisch DNA? Geef van elk voorbeeld en leg hun functie uit.
🧬 1. Intragenisch DNA
🔹 Definitie:
Intragenisch DNA is het DNA dat zich binnen de grenzen van een gen bevindt.
🔹 Bestanddelen:
Exonen: coderende regio’s die uiteindelijk vertaald worden in eiwitten.
Introns: niet-coderende stukken tussen de exonen, die uitgespliced worden tijdens RNA-processing.
Regulatoire sequenties binnen het gen zoals interne enhancers of silencers.
🔹 Functies:
Introns kunnen alternatieve splicing mogelijk maken ➝ eiwitdiversiteit.
Sommige introns bevatten regulatoire elementen die de transcriptie beïnvloeden.
Exonen bevatten de feitelijke genetische code (codons) voor eiwitsynthese.
🔹 Voorbeeld:
Het CFTR-gen bevat 24 exonen en lange introns die alternatieve splicing toelaten.
🌌 2. Intergenisch DNA
🔹 Definitie:
Intergenisch DNA is het DNA tussen twee genen in. Het behoort niet tot een gen.
🔹 Bestanddelen:
Regulatoire elementen (enhancers, silencers) op afstand.
Repetitief DNA zoals LINEs, SINEs, satelliet-DNA.
Pseudogenen, transposons of long non-coding RNA-genen.
Insulators en structurele elementen zoals MARs (matrix attachment regions).
🔹 Functies:
Regulatie van expressie van naburige genen.
Structureren van chromatine (bv. vorming van TADs).
Evolutionaire oorsprong van nieuwe genen (door duplicatie, transpositie, mutatie).
Mogelijke rol in genoomplasticiteit.
🔹 Voorbeeld:
Een enhancer van het β-globine gen ligt ver buiten het gen zelf, in een intergenische regio.
📊 Vergelijkingstabel
Kenmerk | Intragenisch DNA | Intergenisch DNA |
|---|---|---|
Locatie | Binnen een gen | Tussen genen in |
Voorbeelden | Exons, introns | Enhancers, pseudogenen, transposons |
Coderend? | Gedeeltelijk (exonen wel, introns niet) | Meestal niet-coderend |
Functie | Eiwitcodering, splicing, genregulatie | Regulatie op afstand, structuur, variatie |
Wat wordt bedoeld met single-copy DNA? Waarom is dit belangrijk voor de organisatie van genen?
🧬 1. Wat is single-copy DNA?
Single-copy DNA (ook wel uniek DNA genoemd) verwijst naar DNA-sequenties die slechts één keer voorkomen in het genoom (of hooguit zeer beperkt gedupliceerd zijn).
Het tegenovergestelde is repetitief DNA, dat in veel kopieën voorkomt (bv. LINEs, SINEs).
📌 2. Kenmerken van single-copy DNA
Bevat de meeste coderende genen (eiwitcoderende exonen).
Komt niet herhaaldelijk voor in het genoom.
Is meestal functioneel uniek en belangrijk voor genetische stabiliteit.
Vaak te vinden in euchromatine (actieve, toegankelijke DNA-regio’s).
🧪 3. Belang voor genorganisatie
✔ Codeert voor functionele genen
→ Zoals transcriptiefactoren, receptoren, enzymes…
✔ Bevat volledige genstructuren
→ Inbegrepen: promotor, exonen, introns, UTR’s (untranslated regions).
✔ Minder ruis bij regulatie
→ Omdat het niet gerepliceerd is, kunnen specifieke transcriptiefactoren er gericht op binden zonder verwarring met andere kopieën.
✔ Genetisch onderzoek & diagnostiek
→ Mutaties in single-copy genen zijn makkelijker te traceren en interpreteren (bv. in het CFTR-gen bij cystische fibrose).
📚 Voorbeeld:
Het insuline-gen, het β-globine-gen en het BRCA1-gen zijn voorbeelden van single-copy genen die slechts op één locus voorkomen in het humane genoom
Wat is het verschil in structuur en functie tussen het nucleair DNA en mitochondriaal DNA in de mens?
🧬 1. Nucleair DNA (nDNA)
🔹 Structuur:
Bevindt zich in de celkern.
Bestaat uit 46 lineaire chromosomen (23 paar: 22 autosomen + 1 paar geslachtschromosomen).
Heeft een totaal van ~3,2 miljard basenparen.
DNA is verpakt in chromatine met histonen.
Bevat exonen, introns, regulatoire sequenties, repetitief DNA, enz.
🔹 Functie:
Bevat bijna alle genetische informatie die nodig is voor de ontwikkeling, functie en regulatie van het hele organisme.
Codeert voor de meeste eiwitten, RNA’s, en regulatoire elementen.
Wordt zowel van vader als moeder geërfd (diploïd genoom).
🔋 2. Mitochondriaal DNA (mtDNA)
🔹 Structuur:
Bevindt zich in de mitochondriën (buiten de kern).
Bestaat uit één circulair DNA-molecuul van ongeveer 16.569 basenparen.
Niet verpakt in chromatine, geen histonen.
Heeft geen introns.
Genetisch compact: genen liggen vaak overlappend of direct achter elkaar.
🔹 Functie:
Codeert voor 13 eiwitten betrokken bij de oxidatieve fosforylatie (ATP-productie),
22 tRNA’s en 2 rRNA’s.
Speelt een centrale rol in de energielevering van de cel.
Wordt uitsluitend maternally (via de moeder) doorgegeven.
Leg uit wat repetitief DNA is. Welke types repetitieve sequenties komen voor in het humane genoom en wat zijn hun mogelijke functies?
🔁 1. Wat is repetitief DNA?
Repetitief DNA bestaat uit sequenties die meerdere keren herhaald voorkomen in het genoom.
Ongeveer 50% van het humane genoom bestaat uit repetitief DNA, waarvan het overgrote deel niet-coderend is.
🧬 2. Twee hoofdgroepen van repetitief DNA
🧱 A. Transposon-afgeleid repetitief DNA (~45%)
Deze sequenties stammen af van mobiele genetische elementen (transposons), waarvan de meeste vandaag inactief zijn, maar nog wel in het genoom aanwezig.
🔹 1. LINEs (Long Interspersed Nuclear Elements)
Lange sequenties (~6 kb).
Autonoom: bevatten eigen omgekeerde transcriptase en endonuclease.
Kunnen zichzelf kopiëren via een copy-paste mechanisme (retrotranspositie).
Voorbeeld: LINE-1 (L1) – actieve elementen in somatische cellen (bv. in tumoren).
➕ Kunnen mutaties of genverstoring veroorzaken bij insertie.
🔹 2. SINEs (Short Interspersed Nuclear Elements)
Korte sequenties (~100–400 bp).
Niet-autonoom: gebruiken enzymen van LINEs voor retrotranspositie.
Voorbeeld: Alu-elementen (~1,5 miljoen kopieën).
Vertegenwoordigen >10% van het genoom.
Kunnen alternatieve splicing beïnvloeden of genen onderbreken.
🔹 3. Retrovirusachtige elementen (HERVs)
Endogene retrovirussen: verouderde virusinserts in het genoom.
Bevatten vaak LTRs (long terminal repeats).
Kunnen dienen als regulatoire sequenties voor genexpressie.
🔹 4. DNA-transposons
Oudere klasse van transposons (niet meer actief in humane cellen).
Verplaatsen zich via een cut-and-paste mechanisme (zonder RNA-tussenstap).
Sporen ervan vormen ~3% van het genoom.
🔂 B. Tandem repetitief DNA (tandem repeats)
Bestaat uit kopieën van korte DNA-sequenties die naast elkaar liggen. Afhankelijk van de lengte en locatie onderscheiden we:
🔹 1. Satelliet-DNA
Grote blokken herhaalde sequenties (100 kb tot megabases).
Vooral in centromeren en telomeren.
➕ Spelen een rol in chromosoomstructuur, segregatie, herkenning door kinetochoren.
🔹 2. Minisatellieten (10–100 bp repeats)
Lengte: 0,1 tot enkele kb.
Hoog polymorf ➝ DNA fingerprinting / VNTRs (Variable Number of Tandem Repeats).
🔹 3. Microsatellieten (1–6 bp repeats)
Zeer kort (bv. (CA)n, (CAG)n).
Hoog polymorf ➝ genetische markers, stamboomonderzoek, forensisch gebruik.
Instabiliteit ➝ betrokken bij ziekten zoals:
Huntington (CAG-repeat expansie),
Fragiele X (CGG-repeat expansie).
⚙ 3. Mogelijke functies van repetitief DNA
✅ Structurele functies
Centromeer- en telomeersequenties → stabiliteit bij celdeling.
MAR/SAR-sequenties → verankering van chromatine aan het nucleair matrix.
Scaffold-eiwitinteractie → betrokken bij chromosoomorganisatie (bv. TAD’s).
🔁 Regulatoire functies
Sommige transposons bevatten enhancer-, silencer- of promotor-activiteit.
Kunnen transcriptie beïnvloeden als ze nabij een gen terechtkomen.
Kunnen functioneren als bindingsplaatsen voor transcriptiefactoren.
🧬 Evolutionaire rol
Zorgen voor genoomplasticiteit:
Gen-duplicaties
Exon-shuffling
Nieuwe regulatoire netwerken
Spelen rol in soortevolutie.
⚠ Ziektegerelateerd
Inserties kunnen leiden tot genverstoring of abnormale expressie.
Repeat expansies → neurologische en genetische aandoeningen.
Activatie van transposons in tumoren → genoominstabiliteit.
📚 Voorbeelden van ziekten gelinkt aan repetitief DNA
Ziekte | Type herhaling | Gen of locatie |
|---|---|---|
Huntington | (CAG)n | HTT-gen |
Fragiele X-syndroom | (CGG)n | FMR1-gen |
Myotone dystrofie type 1 | (CTG)n | DMPK-gen |
Het gen dat codeert voor cystic fibrosis transmembrane conductance regulator (CFTR) is een voorbeeld van een complex gen. Beschrijf de opbouw van dit gen. Wat is er bijzonder aan?
🧬 1. Wat is het CFTR-gen?
Het CFTR-gen (Cystic Fibrosis Transmembrane Conductance Regulator) is een gen dat codeert voor een chloridekanaal-eiwit in epitheelcellen, essentieel voor iontransport en vochtbalans in o.a. longen, pancreas, darmen en zweetklieren.
Mutaties in dit gen veroorzaken mucoviscidose (cystische fibrose), een van de meest voorkomende autosomaal recessieve genetische aandoeningen bij mensen van Europese afkomst.
🧬 2. Opbouw van het CFTR-gen
Gelegen op chromosoom 7q31.2
Lengte van het gen: ongeveer 189 kb (kilobasen) aan DNA
Aantal exonen: 27 exonen (inclusief het initiator-exon)
Groot aantal en lange introns: sommige introns zijn zeer groot, waardoor het totale gen veel groter is dan het mRNA dat eruit voortkomt
Het pre-mRNA ondergaat intensieve splicing
🔁 3. Wat maakt het CFTR-gen bijzonder of complex?
🔹 A. Groot gen met veel introns
Hoewel het volledige gen bijna 190 kb is, is het mature mRNA slechts ± 6,5 kb.
➤ Dit betekent dat slechts ~3% van het CFTR-gen effectief in het eiwit terechtkomt (de exonen).
🔹 B. Intensieve alternatieve splicing
Het CFTR-gen kent alternatieve splicingvarianten afhankelijk van het weefseltype, wat resulteert in verschillende isoformen van het CFTR-eiwit.
🔹 C. Mutatiegevoeligheid
Meer dan 2000 mutaties/varianten beschreven, waaronder:
ΔF508: deletie van fenylalanine op positie 508 (meest voorkomende mutatie)
Splice site-mutaties (bv. in intron 8)
Nonsense, missense, frameshift en insertie/deletiemutaties
Door de grootte van het gen en de veelheid aan introns is het een hotspot voor mutaties.
🔹 D. Regulatoire complexiteit
Bevat meerdere enhancer- en silencer-elementen, waarvan sommige ver stroomopwaarts of stroomafwaarts van het gen liggen (→ vereist DNA-looping).
Expressie wordt gereguleerd door verschillende weefsel-specifieke transcriptiefactoren.
🧪 4. Klinische relevantie
Door zijn complexiteit is CFTR een uitdaging voor genetische diagnostiek.
De combinatie van lange introns, veel splice sites en diverse mutatietypes maakt het nodig om volledige genanalyse (DNA + mRNA) te doen voor correcte diagnose.
Ook doelwit voor gentherapie en mRNA-gebaseerde behandelingen (zoals CFTR-modulatoren).
🧠 Kort samengevat
Het CFTR-gen is een groot, complex gen met 27 exonen, lange introns en uitgebreide splicing. Door zijn structurele complexiteit en mutatiegevoeligheid is het een schoolvoorbeeld van een eukaryoot gen dat zowel biologisch als klinisch belangrijk is.
Wat zijn transposons? Welk type repetitief DNA komt hieruit voort en wat is het belang ervan voor het genoom?
🔁 1. Wat zijn transposons?
Transposons, ook wel "springende genen" genoemd, zijn mobiele genetische elementen die zich binnen het genoom kunnen verplaatsen of kopiëren.
Ze vormen een groot deel van het repetitieve DNA in het menselijke genoom en zijn een belangrijke bron van genetische variatie.
Er zijn twee hoofdtypes van transposons:
🔹 A. Class I – Retrotransposons (copy-paste mechanisme) → LTR en NON-LTR
Verplaatsen zich via een RNA-intermediair.
Worden eerst getranscribeerd naar RNA → dan terug omgezet in DNA door reverse transcriptase → nieuw DNA wordt in het genoom geïntegreerd.
Voorbeeld: LINEs, SINEs (→ non LTR retrotransposons)
Meest voorkomende type in de mens.
🔹 B. Class II – DNA-transposons (cut-paste mechanisme)
Worden direct uit het DNA geknipt en in een andere locatie geplakt door een transposase.
Niet meer actief in het humane genoom, maar nog wel als resten aanwezig.
🧬 2. Transposon-afgeleid repetitief DNA in de mens
Meer dan 45% van het humane genoom is afkomstig van oude of afgeleide transposons. Enkele belangrijke types:
Type | Omschrijving |
|---|---|
LINEs | Long Interspersed Nuclear Elements (~6 kb) — bevatten eigen enzymen zoals RT |
SINEs | Short Interspersed Nuclear Elements (~300 bp) — gebruiken enzymen van LINEs |
LTRs | Long Terminal Repeats — flankerende sequenties in retrovirusachtige elementen |
DNA-transposons | Verouderde, niet-actieve elementen — hebben ooit genoomstructuur beïnvloed |
🧪 3. Wat is het belang van transposons voor het genoom?
Genetische variatie
Inserties kunnen leiden tot mutaties, genverstoringen of nieuw functioneel DNA.
Kunnen exon-shuffling veroorzaken → nieuwe eiwitcombinaties.
Regulatoire elementen
Sommige transposons bevatten promotors, enhancers of silencers.
Kunnen genexpressie beïnvloeden, bv. door inspringen in of nabij een gen.
Evolutie
Belangrijk bij soortevolutie en genoomuitbreiding.
Bepalen mede de genoomstructuur door herhalingen en duplicaties.
⚠ Ziekte en instabiliteit
Insertie in een belangrijk gen kan leiden tot ziekte (bv. kanker, hemofilie).
Actieve LINE-1 inserties zijn teruggevonden in bepaalde tumortypes.
📌 Voorbeeldtoepassing:
Alu-elementen (SINEs) kunnen alternatieve splicing beïnvloeden.
LINE-1 inserties zijn betrokken bij deactivatie van tumorsuppressorgenen in kankercellen.
Wat zijn pseudogenen en hoe ontstaan ze? Wat is het verschil tussen een unitair pseudogen, een processed pseudogen en een retroposed gen?
🧬 1. Wat zijn pseudogenen?
Pseudogenen zijn DNA-sequenties die sterk lijken op echte genen, maar die niet (meer) functioneel zijn:
➤ Ze worden niet tot een werkend eiwit vertaald, door mutaties, deleties, of afwezigheid van regulatoire sequenties.
Ze zijn ontstaan uit duplicatie of retrotranspositie van bestaande genen, of door verlies van functie van een uniek gen.
Hoewel ze geen functioneel eiwit meer coderen, kunnen ze nog regulatoire of evolutionaire functies hebben.
🧬 2. Hoe ontstaan pseudogenen?
Pseudogenen ontstaan op 3 manieren:
🔹 A. Unitair pseudogen
Ontstaat door eenmalige mutatie in een functioneel gen, zonder duplicatie.
Er is geen functionele kopie meer aanwezig.
Het oorspronkelijke gen wordt dus volledig pseudogeen.
➤ Voorbeeld: GULOP-gen bij mensen (voor vitamine C-synthese), dat bij andere zoogdieren nog actief is.
🔹 B. Processed pseudogen
Ontstaat door reverse transcriptie van mRNA → terug geïntegreerd in het genoom.
Komt voort uit een retroposed gen (zie hieronder).
Kenmerken:
Geen introns (want afkomstig van mRNA)
Vaak poly-A-staart zichtbaar
Geen promotor → meestal niet tot expressie gebracht
🔹 C. Retroposed gen (retro-gen)
Zelfde oorsprong als processed pseudogen, maar wél nog functioneel.
Als het toevallig terechtkomt in een regio met promotoractiviteit, kan het nog tot expressie komen → nieuw functioneel gen ontstaat.
➤ Als het niet functioneel wordt → processed pseudogen
➤ Als het wel functioneel wordt → functioneel retrogen
📊 Vergelijkingstabel
Kenmerk | Unitair pseudogen | Processed pseudogen | Retrogen (retroposed gen) |
|---|---|---|---|
Ontstaan uit | Functioneel gen | mRNA + reverse transcriptie | mRNA + reverse transcriptie |
Introns? | Ja | Nee | Nee |
Promotor? | Ja (maar defect) | Nee | Soms wel (dan functioneel) |
Functioneel? | Nee | Nee | Soms wel |
Voorbeeld | GULOP-gen | Alu-achtige sequenties | RPS3 retrogen |
🧠 4. Mogelijke functies van pseudogenen
Bron van genetische evolutie: kunnen veranderen in functionele genen.
Regulatie van expressie:
Kunnen functioneren als miRNA-spons of RNA-competitie.
Marker bij genoomonderzoek: door hoge gelijkenis met actieve genen, kunnen pseudogenen verstoring geven bij sequencing, of bewuste analyse vereisen.
Wat is copy number variation (CNV)? Hoe kan dit bijdragen aan genetische variatie of ziekte?
🧬 1. Wat is Copy Number Variation (CNV)?
Een copy number variation (CNV) is een vorm van structurele variatie in het genoom waarbij grote DNA-fragmenten (50 bp tot miljoenen bp) in een individu:
meer (duplicatie) of
minder (deletie)
voorkomen dan in een referentiegenoom.
CNV’s kunnen dus leiden tot verschillen in het aantal kopieën van een bepaald DNA-segment tussen individuen.
🔍 2. Kenmerken van CNV’s
Komen voor in ~12–15% van het humane genoom.
Bevatten vaak:
volledige genen,
regulatoire regio’s,
of niet-coderend DNA.
Ze kunnen zowel neutraal als pathogeen zijn.
🧪 3. Hoe ontstaan CNV’s?
Onstabiele repetitieve sequenties ➝ fouten tijdens recombinatie.
Non-allelic homologous recombination (NAHR)
Replicatie-fouten
Transposon-activiteit
🧬 4. CNV’s en genetische variatie
CNV’s zijn een belangrijke bron van genetische diversiteit tussen individuen.
Ze beïnvloeden:
Genexpressie (meer of minder transcriptie door kopie-aantal)
Fenotype (zichtbare of meetbare eigenschappen)
Medicatiegevoeligheid (bv. in CYP-genen)
📌 Voorbeeld:
Meer kopieën van het AMY1-gen (amylase) bij populaties met zetmeelrijk dieet.
⚠ 5. CNV’s en ziekte
CNV’s kunnen ook ziekten veroorzaken, zeker als ze:
Dosagegevoelige genen beïnvloeden (waarvan expressie zeer precies gereguleerd moet zijn)
Tumorsuppressoren of oncogenen dupliceren of deleteren
📚 Ziektevoorbeelden:
Ziekte | CNV-effect |
|---|---|
DiGeorge syndroom (22q11.2 deletie) | Deletie van meerdere genen |
Charcot-Marie-Tooth type 1A | Duplicatie van PMP22-gen |
Autismespectrumstoornis | Geassocieerd met verschillende CNV's in synapsgenen |
Neuroblastoom | Amplificatie van MYCN-oncogen |
📊 Detectie van CNV’s
Array CGH (comparative genomic hybridization)
qPCR, MLPA
Next-generation sequencing (NGS) ➝ bioinformatica-analyse
De meeste menselijke genen bevatten introns. Wat is het nut van deze introns, en hoe kunnen alternatieve splicingvarianten bijdragen aan proteïne-diversiteit?
🧬 1. Wat zijn introns?
Introns zijn niet-coderende sequenties binnen een gen die tijdens de transcriptie worden afgeschreven in het pre-mRNA, maar verwijderd worden door splicing tijdens RNA-processing.
Alleen de exonen blijven over in het mature mRNA, dat wordt vertaald naar een eiwit.
Bijna alle menselijke genen (meer dan 90%) bevatten meerdere introns.
🔍 2. Wat is het nut van introns?
A. Regulatie van genexpressie
Sommige introns bevatten regulatoire elementen zoals enhancers, silencers of microRNA-genen.
Introns kunnen de splicing-efficiëntie en mRNA-export beïnvloeden.
B. Alternatieve splicing mogelijk maken
Door de aanwezigheid van introns kan één gen via verschillende splicingpatronen leiden tot meerdere mRNA-varianten (zie punt 3 hieronder).
C. Evolutie en genmodificatie
Introns beschermen coderende sequenties tegen mutaties.
Bevorderen exon-shuffling, wat de evolutie van nieuwe eiwitfuncties mogelijk maakt.
D. Mogelijke opslagplaats voor genetische informatie
Introns kunnen reservekopieën van bepaalde motieven bevatten (bv. voor genregulatie of interactiedomeinen).
🔁 3. Alternatieve splicing en eiwitdiversiteit
Alternatieve splicing = het proces waarbij uit één enkel pre-mRNA verschillende combinaties van exonen worden samengevoegd.
Dit leidt tot:
mRNA-isoformen met verschillende exonen.
➤ Resultaat: verschillende eiwitvarianten met mogelijk verschillende functies, stabiliteit of locatie.
📚 Voorbeelden:
Het Dscam-gen bij fruitvliegen kan via alternatieve splicing tot wel 38.000 verschillende eiwitten produceren.
Bij mensen:
TPM1-gen (tropomyosine): verschillende isoformen voor spier- en niet-spiercellen.
Bcl-x-gen: splicing bepaalt of het eiwit anti-apoptotisch of pro-apoptotisch is.
⚠ 4. Verstoorde splicing en ziekte
Fouten in splicing kunnen leiden tot:
Verlies van functie (bv. exon skipping bij Duchenne spierdystrofie)
Verkeerd eiwit (bv. bij bepaalde vormen van kanker)
Inactief CFTR-eiwit bij cystische fibrose (bv. mutaties in intron 8 of splicesites)
Wat zijn CpG eilanden en wat is hun functie?
🧬 1. Wat zijn CpG-eilanden?
CpG-eilanden zijn DNA-regio’s die rijk zijn aan:
Cytosine (C) direct gevolgd door Guanine (G) op dezelfde DNA-streng, dus:
5'-CG-3' (de 'p' verwijst naar de fosfaatgroep tussen de nucleotiden)
Normaal zijn CpG-combinaties schaars in het genoom omdat cytosines in CpG's vaak gemethyleerd zijn en over de evolutie heen gemuteerd zijn naar T.
In CpG-eilanden is dit níet het geval: de cytosines zijn meestal niet gemethyleerd.
📏 Kenmerken van CpG-eilanden:
Typisch 200 tot 2000 basenparen lang.
Hebben een hoge GC-inhoud (>50%).
Hoge frequentie van CpG-dinucleotiden vergeleken met de rest van het genoom.
Liggen vaak in of nabij promotors van genen (vooral van housekeeping genes).
🧪 2. Wat is de functie van CpG-eilanden?
Regulatie van genexpressie
Ongecodeerde CpG-eilanden in promotors zijn meestal niet gemethyleerd → het gen is actief of kan makkelijk geactiveerd worden.
Wanneer CpG-eilanden wel worden gemethyleerd (bv. op cytosines), wordt het DNA strakker opgevouwen:
→ geen transcriptiefactorenbinding meer
→ transcriptie wordt geblokkeerd
➤ Methylatie van CpG-eilanden = vaak onderdrukking (silencing) van het bijhorende gen
🧬 3. Biologische en medische relevantie
Normale cel: CpG-eilanden zijn niet-gemethyleerd bij actieve genen.
Tijdens differentiatie of X-inactivatie: CpG-eilanden kunnen gemethyleerd worden om genen permanent uit te schakelen.
Kanker: abnormale hypermethylatie van tumorsuppressorgenen → leidt tot onderdrukking van genen die celgroei remmen.
Epigenetische therapieën (bv. DNA-demethylase remmers) richten zich op deze modificaties
Wat zijn SNP’s?
🧬 1. Wat is een SNP?
SNP staat voor Single Nucleotide Polymorphism = enkel-nucleotide-polymorfisme.
Het is een variatie in één enkele nucleotide (A, T, C of G) op een specifieke positie in het DNA, waarbij minstens 1% van de populatie deze variant heeft.
🔹 Voorbeeld:
Op een bepaalde plaats in het DNA heeft:
90% van de mensen de sequentie A
10% heeft daar G
Dan spreken we van een SNP.
📌 2. Waar komen SNP’s voor?
SNP’s komen gemiddeld elke 100 à 300 basenparen voor in het menselijke genoom.
Ze kunnen zich bevinden in:
Coderende regio’s (exonen) ➝ mogelijk effect op het eiwit
Niet-coderende regio’s zoals introns, promotors, UTR’s ➝ invloed op genexpressie of splicing
Intergenisch DNA ➝ vaak neutraal, maar kunnen als marker dienen
🧪 3. Belang en toepassingen van SNP’s
A. Genetische variatie
SNP’s verklaren een groot deel van de interindividuele verschillen:
Uiterlijk (oogkleur, haar)
Metabole snelheid
Reactie op geneesmiddelen
B. Ziekte-associaties
Sommige SNP’s verhogen de kans op een bepaalde ziekte of verstoren genfunctie:
bv. SNP in het APOE-gen → verhoogd risico op Alzheimer
SNP in een splicing-site → abnormaal eiwitproduct
C. Farmacogenetica
Bepalen hoe goed iemand een bepaald geneesmiddel opneemt, afbreekt of verdraagt.
bv. CYP2D6-varianten beïnvloeden metabolisme van antidepressiva
D. DNA-analyse en genetisch onderzoek
SNP’s zijn veelgebruikte genetische markers in:
Genome-Wide Association Studies (GWAS)
Stamboomanalyse
Forensisch onderzoek
🧠 Kort samengevat
Een SNP is een variatie in één base in het DNA tussen individuen, die voorkomt bij minstens 1% van de bevolking. SNP’s dragen bij aan genetische diversiteit, beïnvloeden ziektegevoeligheid, genexpressie en respons op medicijnen, en zijn cruciaal in genetisch onderzoek.
Wat is RFLP?
🧬 1. Wat betekent RFLP?
RFLP staat voor:
🔹 Restriction Fragment Length Polymorphism
= Restrictiefragmentlengte-polymorfisme
Het is een techniek waarmee men genetische variatie tussen individuen kan opsporen op basis van verschillen in lengte van DNA-fragmenten die ontstaan na knippen met een restrictie-enzym.
⚙ 2. Hoe werkt RFLP? 📌 Stappen van de RFLP-analyse:
Isolatie van DNA uit een monster (bv. bloed, speeksel)
Knippen van DNA met een specifiek restrictie-enzym (bv. EcoRI, HindIII)
Dit enzym herkent een bepaalde sequentie (bv. GAATTC) en knipt daar.
Door mutaties of polymorfismen (zoals SNP's) kan deze sequentie bij sommige mensen verdwenen of veranderd zijn:
➤ Restrictieplaats aanwezig? → DNA wordt geknipt
➤ Restrictieplaats afwezig? → geen knip
De verkregen fragmenten worden gescheiden via gel-elektroforese
Detectie gebeurt via:
DNA-kleuring (ethidiumbromide)
of Southern blotting met een DNA-probe
📊 ➤ Dit resulteert in verschillende bandpatronen per individu
🧬 3. Toepassingen van RFLP
A. Genetische identificatie
Forensisch onderzoek (DNA-fingerprint)
Vader- of verwantschapstests
B. Genetische ziekte-analyse
Detectie van mutaties die een restrictieplaats creëren of verwijderen:
bv. sikkelcelanemie: mutatie in het β-globinegen verandert een MstII-plaats
C. Mapping van genen
Gebruikt in linkage-analyse en vroege menselijke genoomstudies
🔬 4. Wat is een RFLP-polymorfisme precies?
Een nucleotidesequentieverschil (meestal een SNP) tussen individuen dat leidt tot:
het aanwezig of afwezig zijn van een herkenningsplaats voor een restrictie-enzym
➤ Dit beïnvloedt de lengte van de DNA-fragmenten
Wat zijn line’s - sine’s - alu sequenties?
❓ Wat zijn LINE’s, SINE’s en Alu-sequenties?
🧬 1. LINE’s (Long Interspersed Nuclear Elements) 🔹 Wat?
Lange repetitieve DNA-sequenties, ~6.000 bp lang
Afgeleid van retrotransposons
Kunnen zichzelf kopiëren en plakken in het genoom via een RNA-intermediair
➤ = autonome retrotranspositie
🔹 Kenmerken:
Bevatten:
Omgekeerde transcriptase (RT)
Endonuclease
Kunnen nog actief zijn in sommige cellen (bv. hersenen, tumoren)
Vertegenwoordigen ~20% van het humane genoom
Belangrijkste type: LINE-1 (L1)
🔹 Belang:
Kunnen mutaties veroorzaken bij insertie
Hebben invloed op genregulatie en chromosoomstructuur
🧬 2. SINE’s (Short Interspersed Nuclear Elements) 🔹 Wat?
Korte repetitieve sequenties, ~100–400 bp
Afgeleid van RNA-genen (bv. tRNA, 7SL RNA)
Niet-autonoom: hebben geen eigen enzymen → gebruiken die van LINEs om zich te verplaatsen
🔹 Kenmerken:
Hebben geen coderende capaciteit
Vertegenwoordigen ~13% van het humane genoom
🔹 Belang:
Kunnen genexpressie beïnvloeden (bv. splicing, transcriptie)
Zijn evolutionair zeer actief geweest
🧬 3. Alu-sequenties: een subtype van SINE’s
🔹 Wat?
De meest voorkomende SINE’s in het humane genoom
Lengte: ongeveer 300 bp
Genoemd naar het AluI restrictie-enzym, dat een herkenningssite bevat in deze sequentie
🔹 Kenmerken:
Meer dan 1,5 miljoen kopieën in het menselijk genoom
➤ Maken ~10% van het genoom uit
Afgeleid van het 7SL RNA (onderdeel van het signaalherkenningsdeeltje)
🔹 Belang:
Kunnen intragenisch terechtkomen → genverstoring
Geassocieerd met ziekten, bv. borstkanker, hemofilie (door inserties)
📊 Samenvattende vergelijking
Kenmerk | LINEs (bv. LINE-1) | SINEs (bv. Alu) |
|---|---|---|
Lengte | ± 6.000 bp | ± 300 bp |
Autonoom? | Ja (heeft eigen enzymen) | Nee (gebruikt LINE-enzymen) |
Herkomst | Retrotransposons | RNA (bv. 7SL, tRNA) |
Aantal kopieën | ± 500.000 | ± 1,5 miljoen (Alu) |
% van het genoom | ~20% | ~13% (waarvan ~10% Alu) |
Effect | Mutaties, genregulatie | Splicing, inserties |
Wat is satelliet-DNA en waar komt het voor in het humane genoom?
Geef twee functies van satelliet-DNA en bespreek kort hoe deze sequenties kunnen bijdragen aan genetische stabiliteit of instabiliteit.
Geef één concreet voorbeeld van waar of hoe satelliet-DNA in de cel functioneel is.
Satelliet-DNA is een vorm van tandem repetitief DNA, bestaande uit grote blokken korte DNA-sequenties die direct naast elkaar liggen in het genoom. De repeats zijn typisch 100 bp tot meerdere megabases lang.
Satelliet-DNA komt vooral voor in:
Centromeren → belangrijk voor de aanhechting van de kinetochoren tijdens de celdeling
Telomeren → beschermen chromosoomuiteinden
➤ Satelliet-DNA speelt een structurele rol in het chromosoom, en is essentieel voor:
Chromosoomstabiliteit
Herkenning en correcte segregatie tijdens mitose en meiose
Soms kan instabiliteit in tandem repeats (vooral micro- en minisatellieten) leiden tot genetische aandoeningen, zoals:
Huntington (CAG-repeat expansie)
Fragiele X (CGG-repeat expansie)
➤ Minisatellieten worden ook gebruikt bij DNA-fingerprinting (VNTRs)
➤ Microsatellieten dienen als genetische markers in forensisch en evolutionair onderzoek
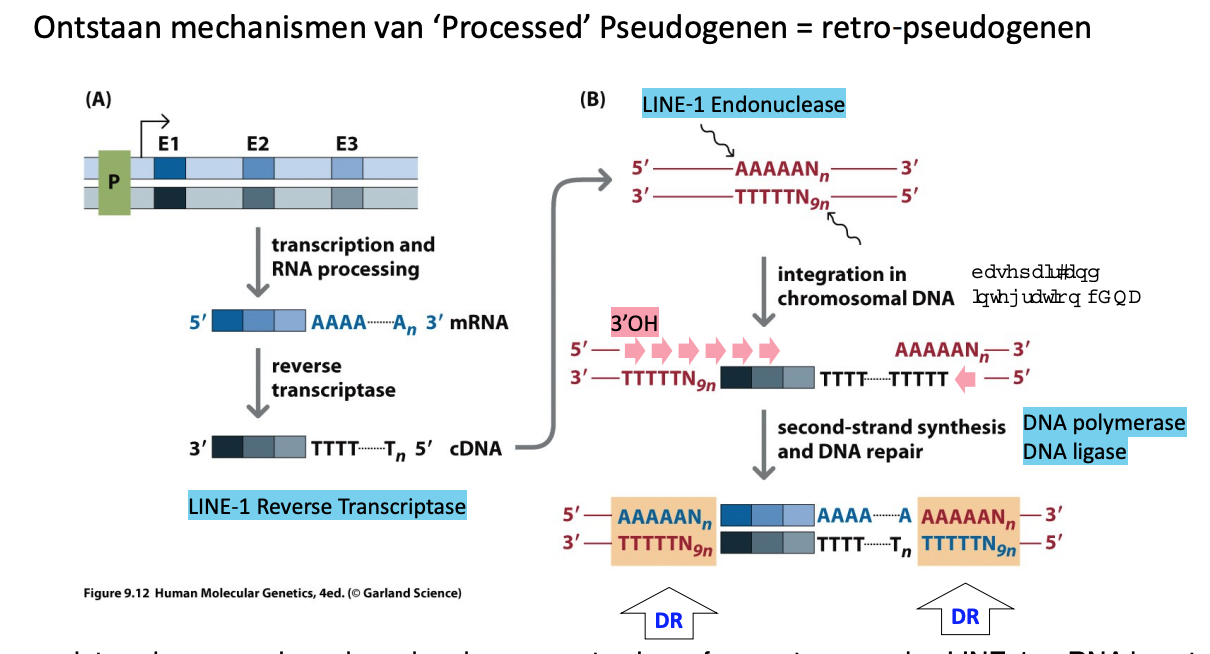
Hoe ontstaat een processed pseudogen (retrogen)?
🧬 Wat zie je in deze afbeelding?
Het toont het ontstaan van een processed pseudogen via retrotranspositie, waarbij een mRNA-kopie van een gen wordt omgezet in DNA en terug in het genoom geïntegreerd wordt, maar zonder introns en zonder regulatoire elementen.
🔄 Stapsgewijze uitleg 📍 (A) Links – Van functioneel gen naar cDNA
Transcription and RNA processing
Een actief gen (met exonen E1, E2, E3) wordt afgeschreven tot mRNA.
Tijdens RNA-processing worden de introns verwijderd → enkel exonen blijven over.
Het mRNA bevat een poly-A-staart aan het 3’-uiteinde.
Reverse transcriptase maakt cDNA
LINE-1 reverse transcriptase (afkomstig van een retrotransposon) zet dit mRNA om in cDNA (complementair DNA).
Het cDNA bevat geen introns en begint met een poly-T-staart (complement van poly-A).
📍 (B) Rechts – Integratie in het genoom
LINE-1 endonuclease knipt chromosomaal DNA
De LINE-1 endonuclease maakt een knip in een A-rijke regio van het DNA → dit is een voorkeursplek voor insertie.
Het cDNA wordt op die plek ingevoegd via strand invasion, dankzij hybridisatie tussen poly-T (cDNA) en A-rijke targetsequentie.
Second-strand synthesis
DNA polymerase en ligase maken de tweede streng van het DNA af → nu is de retrocopy ingevoegd in het genoom.
🔬 Resultaat: een processed pseudogen
Geen introns → want het was afkomstig van mRNA
Poly-A/T-sporen zichtbaar
Geen promotor of regulatie → dus niet actief (of slechts in zeer beperkte mate)
Wordt flankeerd door korte repeats (DR = direct repeats) door foutieve herstelsynthese
Mutaties stapelen op over de tijd → het gen wordt steeds meer inactief
🎓 Belang / waarom dit telt
Dit proces verklaart waarom ons genoom zoveel dode genkopieën bevat.
Deze pseudogenen kunnen soms nog RNA produceren en hebben regulatoire functies (bv. miRNA-spons).
Dit toont hoe retrotransposons zoals LINE-1 bijdragen aan genoomuitbreiding en variatie.
🧠 Kort uitgelegd in 1 zin:
Een processed pseudogen ontstaat wanneer een mRNA via reverse transcriptase wordt omgezet in DNA en zonder introns en promotor wordt teruggeplaatst in het genoom, vaak als inactieve kopie van een gen.

Leg uit
🧬 1. Wat is CpG en waarom is het speciaal?
CpG = een cytosine (C) gevolgd door een guanine (G) op dezelfde DNA-streng, verbonden via een fosfaatgroep (dus: 5'-CpG-3')
Het is een palindroom: de andere streng bevat ook een CpG (namelijk: 3'-GpC-5')
Er zijn 16 mogelijke dinucleotidecombinaties in het DNA (zoals AA, AG, GC, enz.), elk zou gemiddeld ~6,25% mogen uitmaken.
🔴 MAAR: CpG komt veel minder vaak voor dan verwacht (<6%) → dat is de grote “WAAROM?”-vraag uit je dia.
🔬 2. Waarom is CpG zeldzaam in het genoom?
Het antwoord zit in methylatie en spontane mutaties:
➤ A. DNA-methylatie
In het genoom wordt cytosine in CpG’s vaak gemethyleerd tot 5-methylcytosine (5mC).
➤ Dit is een epigenetische markering die genexpressie kan onderdrukken.
➤ B. Spontane deaminatie van cytosine
Niet-gemethyleerd cytosine → deaminatie → uracil
➤ Wordt herkend als fout → gerepareerd door het DNA-herstelmechanisme.
➤ C. Spontane deaminatie van 5-methylcytosine
5mC → deaminatie → thymine (T)
➤ Probleem: T is een normale base → wordt niet als fout herkend
➤ Hierdoor ontstaat blijvend een C→T mutatie op de ene streng en G→A op de andere = transitie-mutatie
🔁 Dit leidt tot:
Langzaam verlies van CpG’s in het genoom over evolutionaire tijd → ondervertegenwoordiging van CpG-dinucleotiden
📌 3. Wat zijn CpG-eilanden dan?
In sommige genregio’s — vooral promotors van genen — zijn er zones waar:
CpG’s wel rijk aanwezig zijn
CpG’s niet gemethyleerd zijn ➝ zodat het gen actief kan blijven
Die zones heten CpG-eilanden
Gemiddeld >200 bp
>50% GC-inhoud
Ongeveer 30.000 CpG-eilanden in het humane genoom
Liggen vaak bij gen-promotors ➝ dienen als genexpressie-regulatieplaatsen of zelfs genmarkers
🧠 Samenvatting in eenvoudige woorden
CpG's zijn zeldzaam in het genoom omdat ze vaak gemethyleerd worden, en gemethyleerde C’s kunnen spontaan muteren naar T. Omdat die fout niet herkend wordt, verdwijnen CpG’s geleidelijk. Alleen in CpG-eilanden, die belangrijk zijn voor genactivatie, blijven ze behouden én ongemethyleerd.
Wat is exon shuffling?
🧬 1. Definitie
Exon shuffling is een proces waarbij exons (coderende stukken van genen) herverdeeld of gecombineerd worden tussen verschillende genen, vaak als gevolg van recombinatie, duplicatie of transpositie.
➡ Hierdoor kunnen nieuwe gencombinaties ontstaan, wat leidt tot nieuwe eiwitstructuren of -functies.
⚙ 2. Hoe gebeurt exon shuffling?
Exon shuffling kan optreden door:
🔁 Homologe recombinatie
Tijdens celdeling (meiose) kunnen ongelijke cross-overs gebeuren tussen intron-sequenties → exons wisselen van plaats tussen genen.
🧬 Transposonactiviteit
Mobiele elementen (zoals LINEs of SINEs) kunnen exons meeslepen en invoegen op nieuwe locaties.
📦 Gen-duplicatie gevolgd door hercombinatie
Een gen wordt gekopieerd, en vervolgens worden exons uitgewisseld tussen kopieën.
🔹 Belangrijk: introns maken shuffling mogelijk zonder coderende sequenties te verstoren (intron–exon-grenzen blijven behouden).
💡 3. Wat is het nut van exon shuffling?
✔ Creëren van nieuwe eiwitcombinaties
Exons coderen vaak voor functionele domeinen van eiwitten (bv. bindingsdomein, katalytisch domein)
Door exons te combineren, ontstaan nieuwe modulaire eiwitten met nieuwe functies
✔ Evolutie versnellen
Genen hoeven niet volledig vanaf nul te evolueren, maar kunnen delen hergebruiken
✔ Voorbeeld:
Immunoglobulinen (antilichamen) → bestaan uit herhaalde domeinen die ontstaan zijn via exon shuffling
Fibronectine: bevat verschillende bindingsdomeinen voor collageen, integrines, fibrine, … die waarschijnlijk via exon shuffling ontstonden
📊 Vergelijking met alternatieve splicing
Exon shuffling | Alternatieve splicing |
|---|---|
Gebeurt op DNA-niveau (evolutief) | Gebeurt op RNA-niveau (regulatoir) |
Exons herschikt tussen genen | Exons geselecteerd binnen één gen |
Leidt tot nieuwe genstructuren | Leidt tot meerdere eiwitvarianten uit 1 gen |
🧠 Kort samengevat
Exon shuffling is het evolutionair proces waarbij exons tussen genen worden herverdeeld of gecombineerd, wat leidt tot nieuwe eiwitten met nieuwe functies. Het draagt bij aan de eiwitdiversiteit en evolutie van complexe organismen