bio MOL chap 2.2 ADN: composition, strucutre et compactage
1/43
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
44 Terms
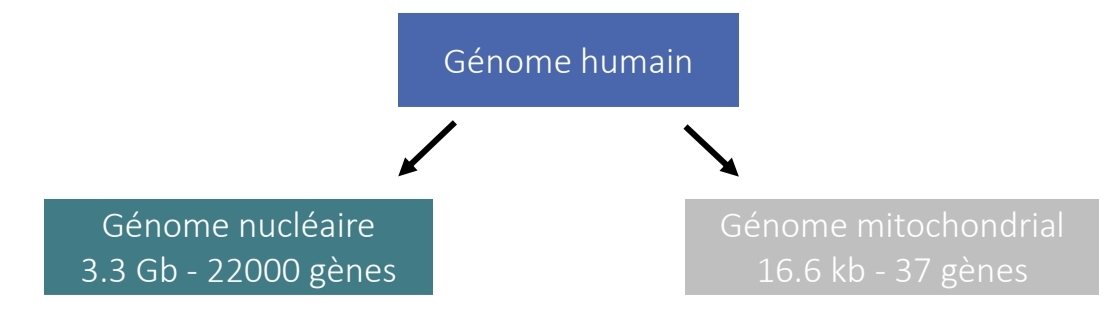
Organisation génome eucaryote
Génome nucléaire → ADN linéaire / chromosomes
Génome nn nucléaire → ADNmito / ADN circulaire / multi-copies
Génome humain
Très hétérogène
Classes séquences ≠
Taille + répétition variable
Fonctions différentes
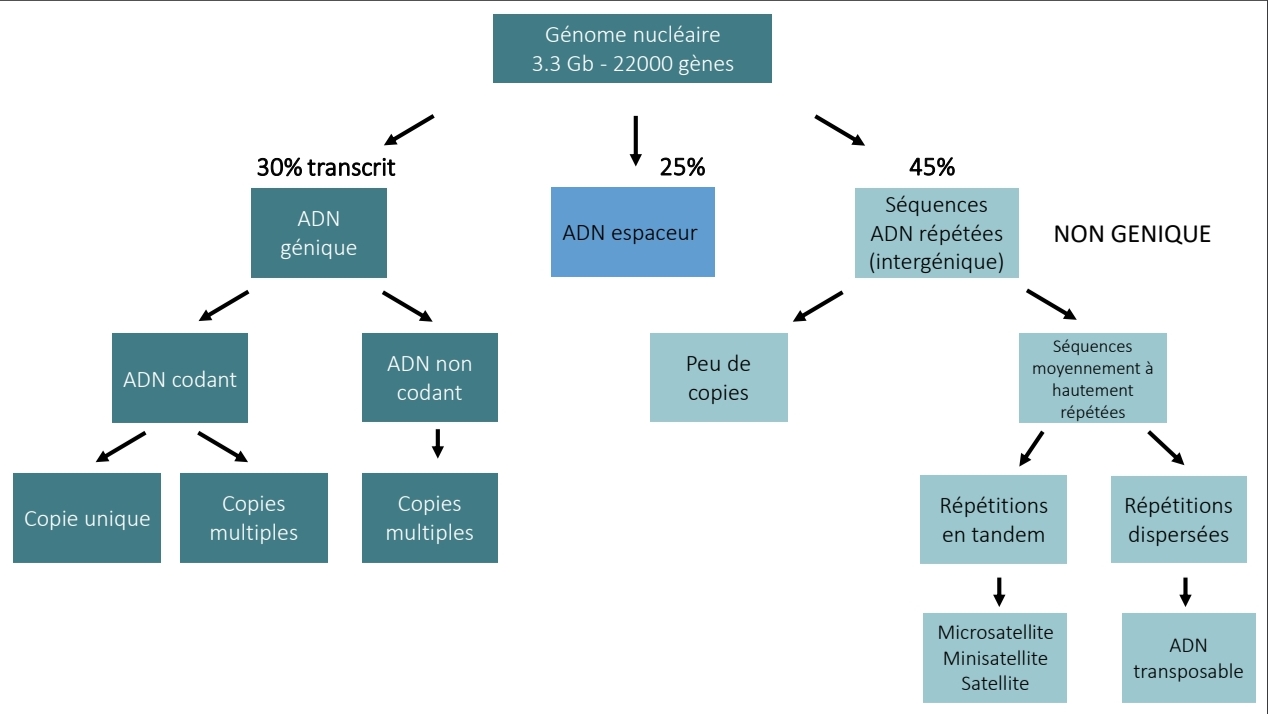
Organisation génome nucléaire
ADN génique
ADN génique 2 type
ADN codant
ADN non codant (transcrit / régulateur)
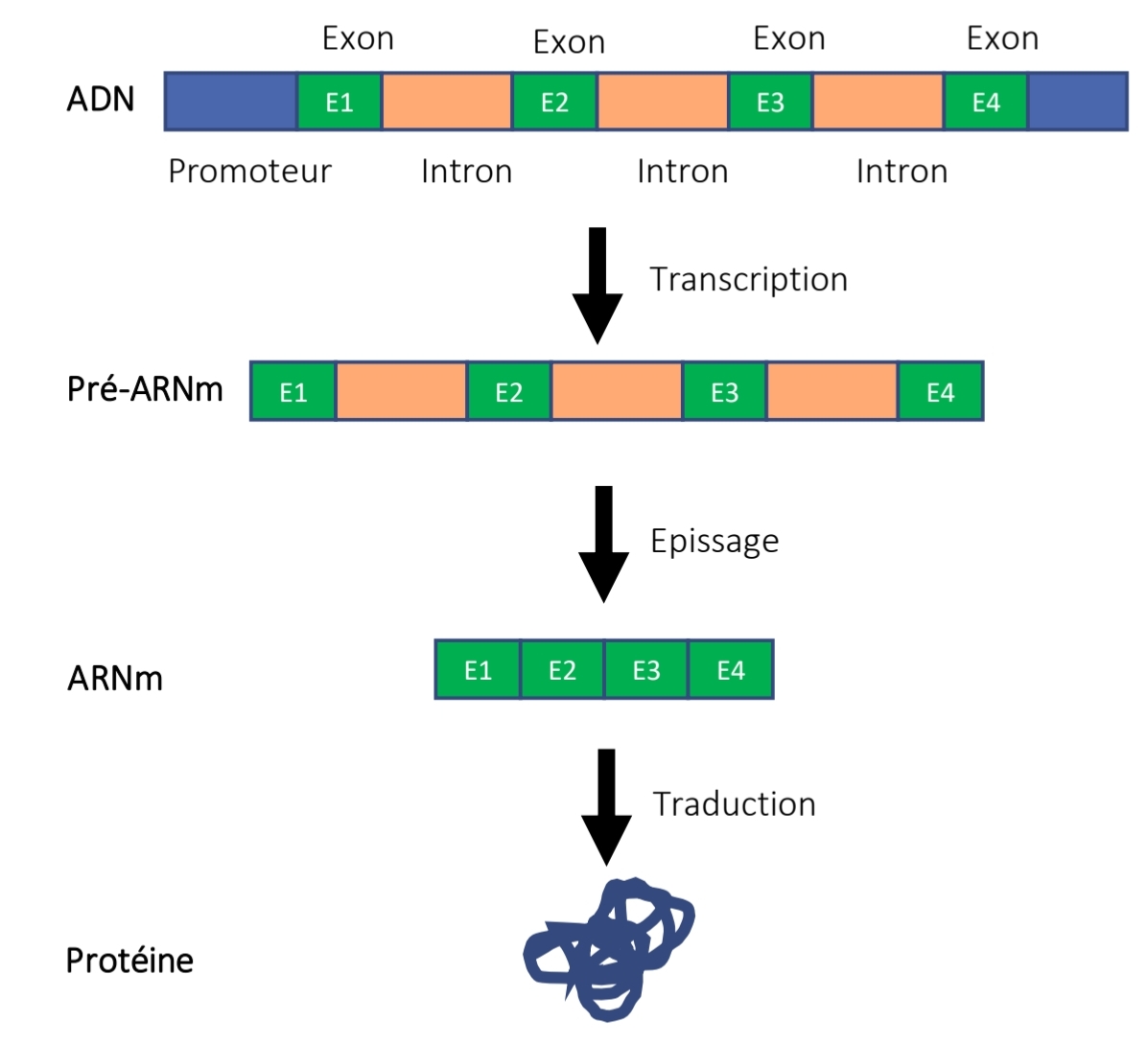
ADN codant : structure gène
Promoteur
Exons + introns
Transcription → pré-ARNm
Épissage → ARNm
Traduction → protéine
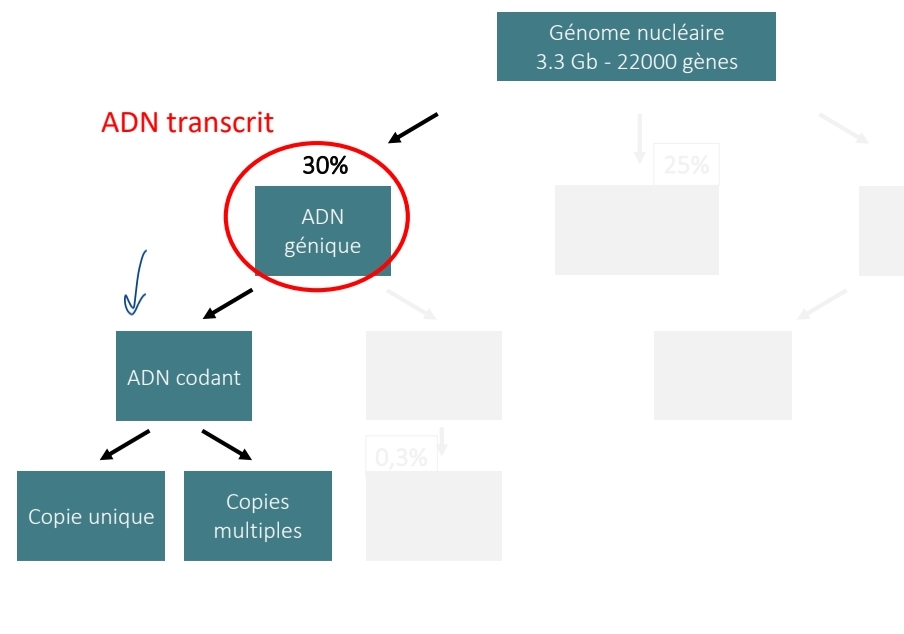
Organisation génome nucléaire
ADN génique
1.1 ADN codant : structure gène
→ Gènes copie unique
1 copie
≈ 15% ADN génomique
Exons ≈ 1,5% + introns
Organisation génome nucléaire
ADN génique
1.1 ADN codant : structure gène
→ Gènes copies multiples =
Gènes dupliqués / divergés → superfamilles
caractéristique proche → seq/struct/ mm type fonction
Séquences proches
Structure proche
Fonction biochimique proche
Rôle physiologique divergent
Organisation génome nucléaire
ADN génique
1.1 ADN codant
→ Gènes copies multiples
Duplication gènes : mécanisme
Alignement séquences répétées (LINE)
Recombinaison homologue
→ crossing-over inégal
Organisation génome nucléaire
ADN génique
1.1 ADN codant
→ Gènes copies multiples
Famille multigénique : formation
Gène ancestral
Duplication +/ transposition
évolue chaccun de leur coté : Mutations
→ gènes divergents
Organisation génome nucléaire
ADN génique
1.1 ADN codant
→ Gènes copies multiples
Superfamille globines
Expression temporelle (embryon → fœtus → adulte)
Perte fonction/expression → pseudogènes
Organisation génome nucléaire
ADN génique
1.1 ADN nn codant
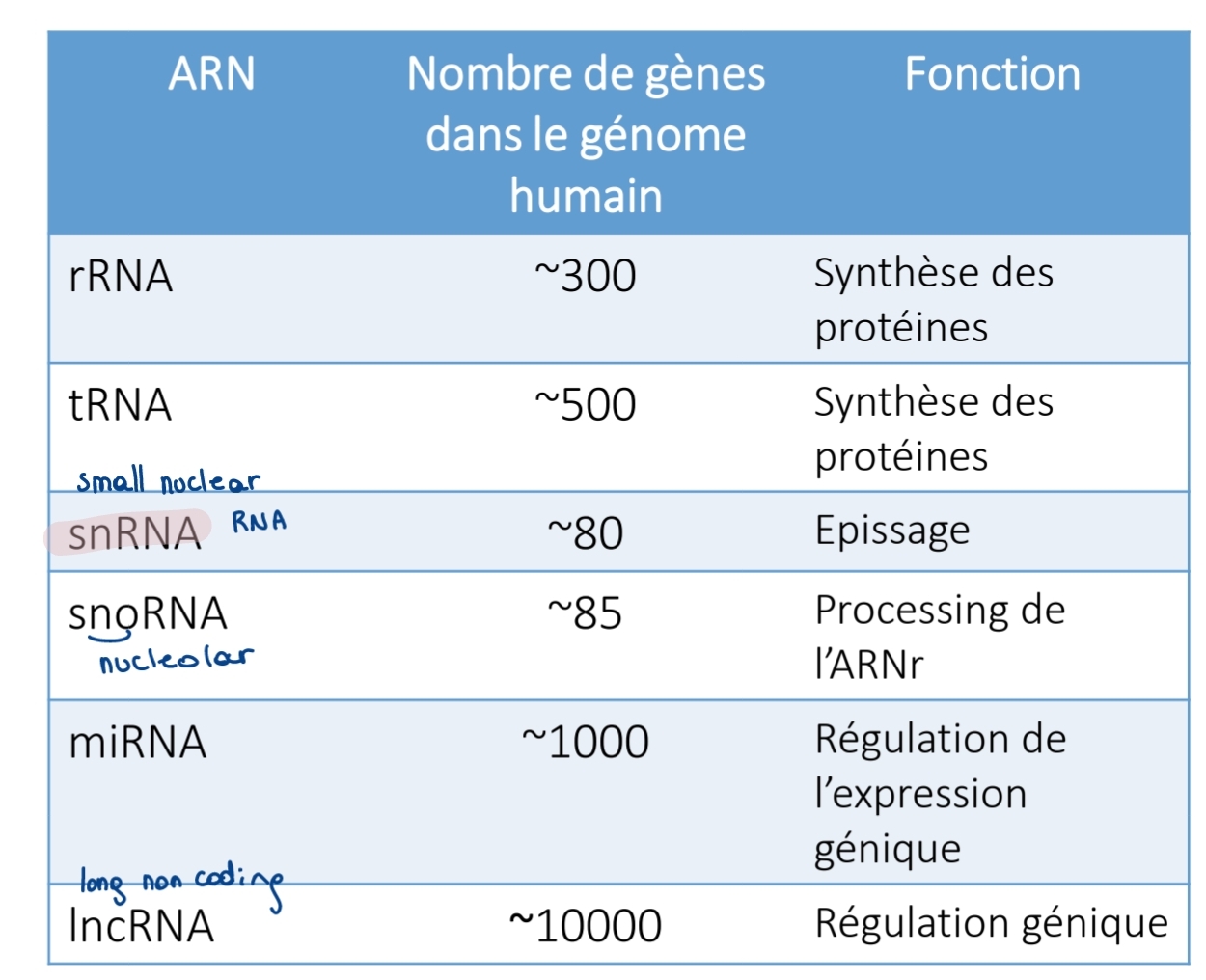
ARN nn codant fonctionnels
Régulation + mécanisme
miRNA: ↓ expression gènes (post-transcription)
lncRNA : Régulation génique
Organisation génome nucléaire
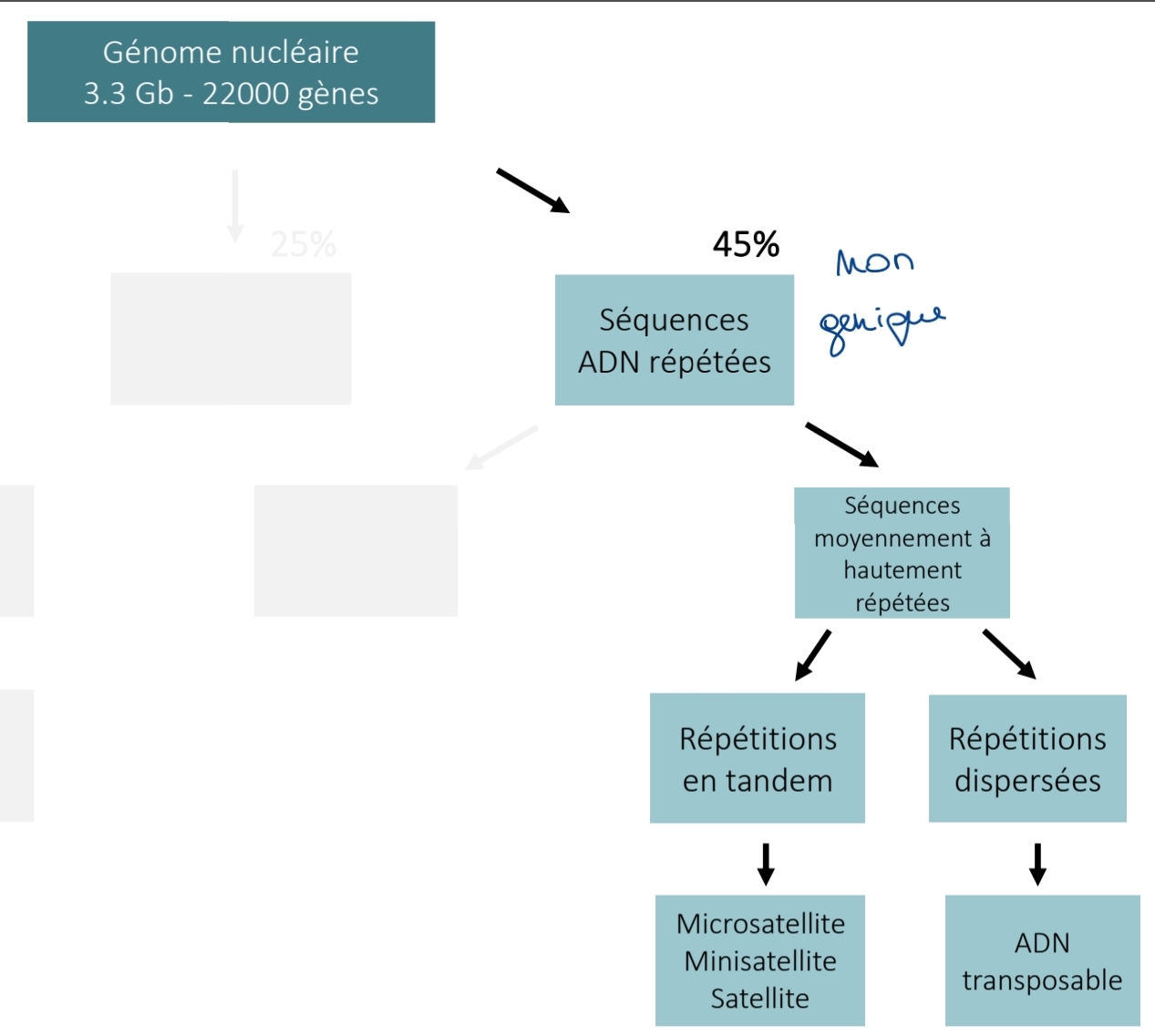
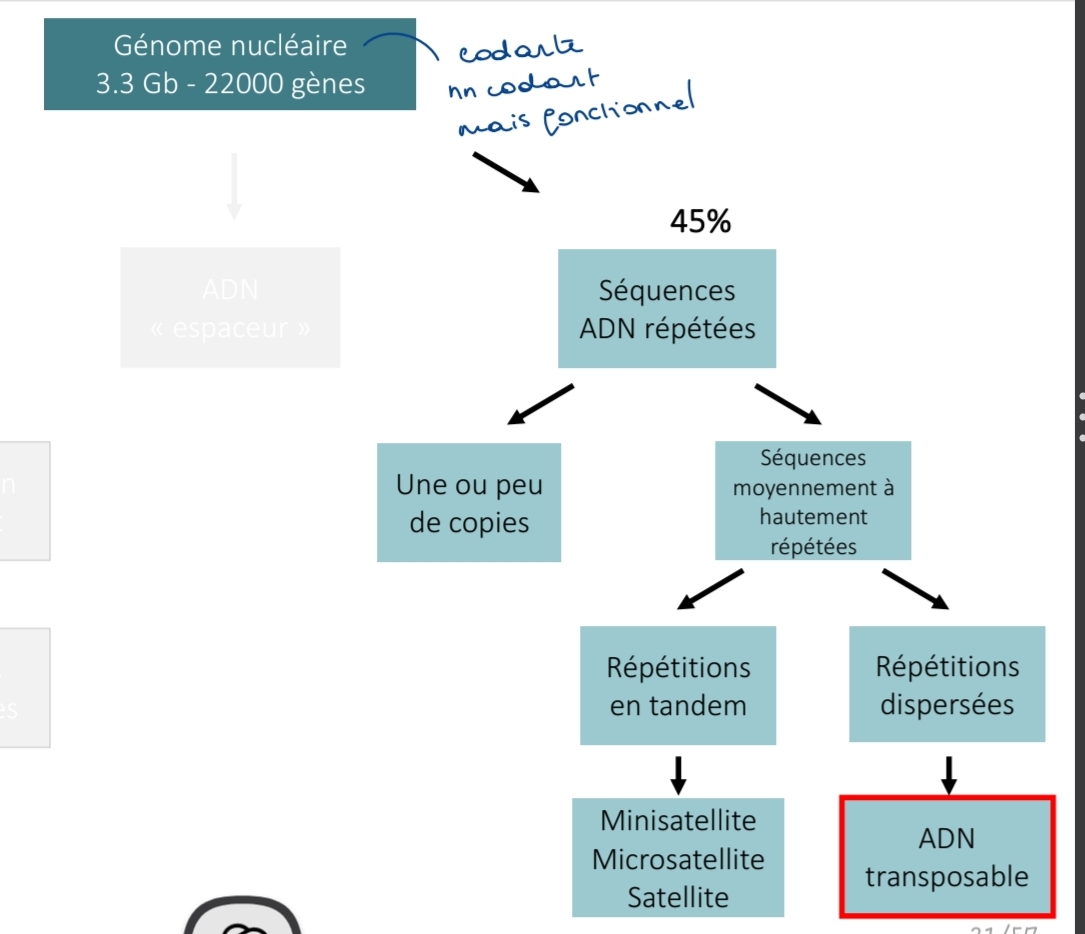
séquence ADN répété
1.2 séquence moyennement à hautment répété
→?
→ ?
Tandem : micro/mini/ … satellite
Dispersées : ADN transposables
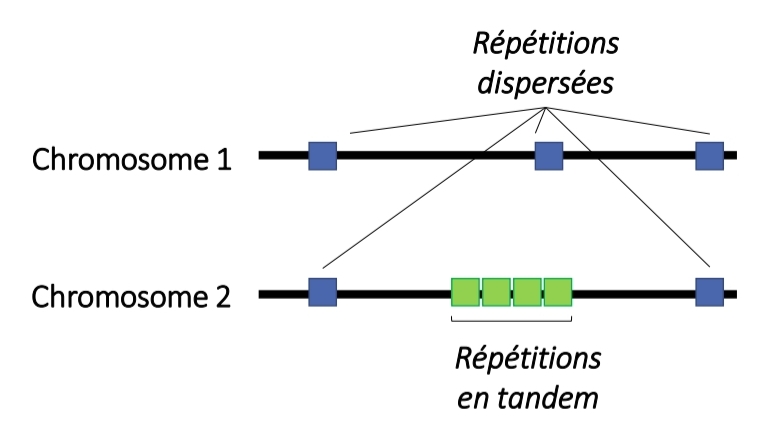
Répétitions dispersées vs répétition en tandem
Copie unique svt / +sieur
mm chromo/≠
vs
gène unique “queu leu leu”
classé : nbr copie/L
→Satellite, Minisatellite, Microsatellite
Organisation chromosome gène répéter
Gènes dispersés
Répétées ↑ près centromères + télomères
ADN répétitif non fonctionnel : général
î génome est d'ADN répétitif entrecoupé par
de l'ADN à copie unique
classé:
microsatellite
minisatellite
satellite
=> toto répété en tandem
élément transposable (LINE/SINE)
=> répété dispersé
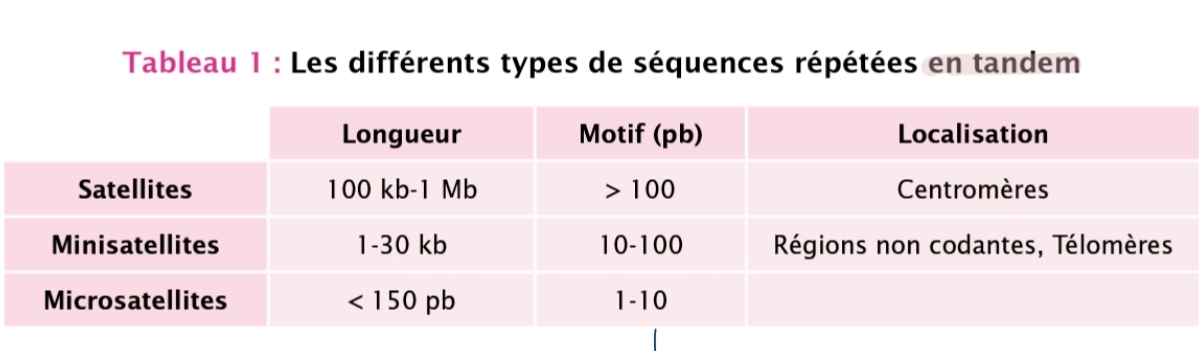
Microsatellites
STRs-SSRs
répétitions tandem
Polymorphisme ↑ (nbr pas cst)
très cours +++
dispersé ds génome (svt télomère)
Dans régions codantes → mutations → protéines anormales
Ex : Huntington (CAG ↑) = dégenerescence neurone
Minisatellites
répétitions tandem
VNTR
courte (-macro)
Minisatellites : détection
PCR
Variants alléliques → tailles ≠
Satellites
Hautement répétitif tandem
plus grand
svt Centromères + télomères
Organisation génome nucléaire
séquence ADN répété
1.2 séquence moyennement à hautment répété
répétitition dispérer
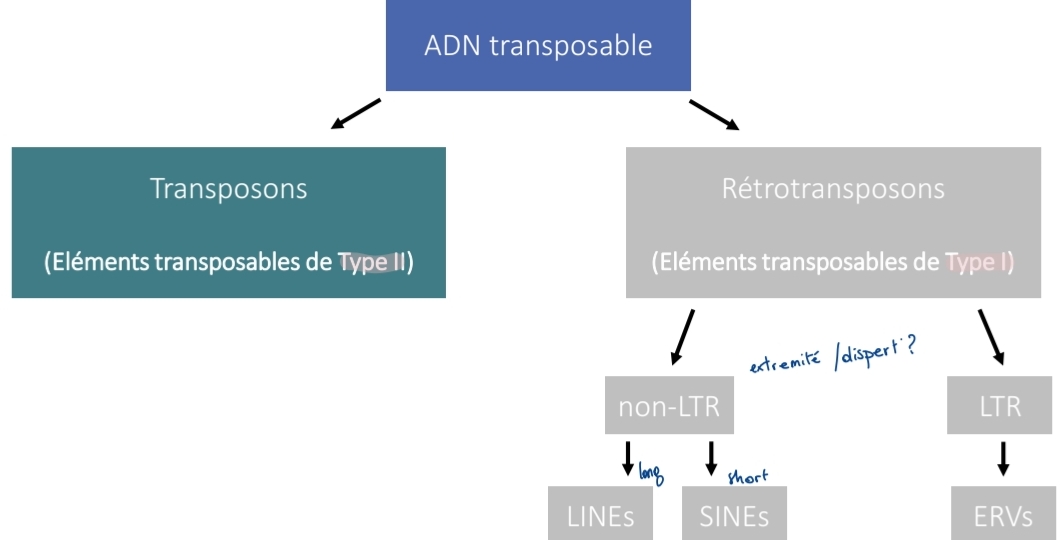
→ADN/Éléments transposables : général
≈ 45% génome humain
Dispersés
Mobiles
~10% actifs
Mutations possibles
Rôle évolution
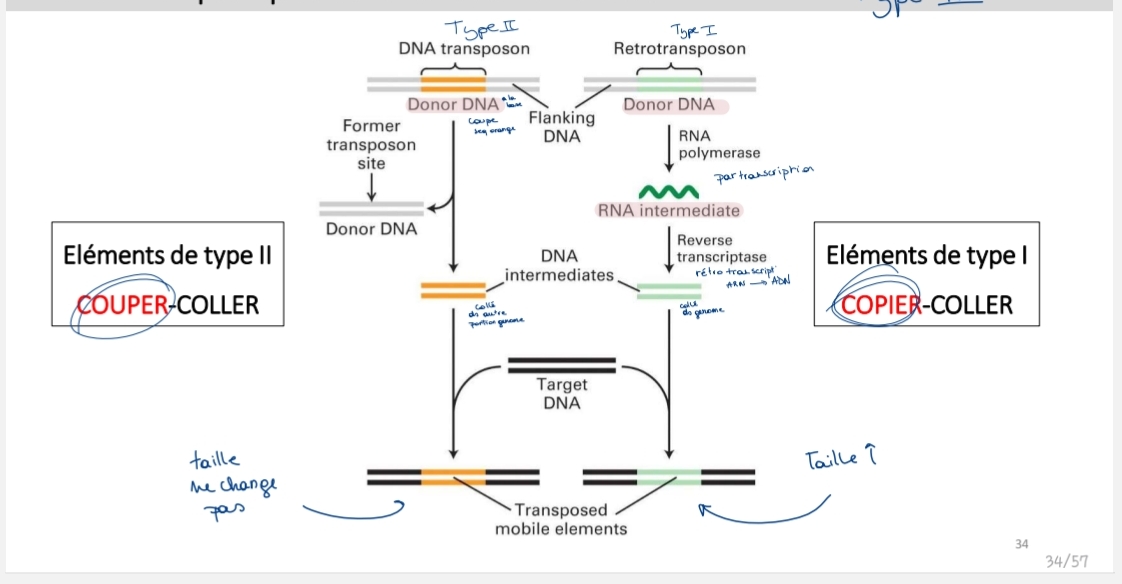
ET : classes
Type I = rétrotransposons
copier-coller
Type II = transposons
couper-coller
Type II
Couper-coller
Transposase
Taille génome = (pas ↑)
≈ 3% génome humain
Type I
Copier-coller
Intermédiaire ARN
Transcription → RT → insertion
Taille génome ↑
Autonome vs non-autonome
Autonome → se déplace seul (LINE-1)
Non-autonome → dépend autre (SINE Alu)
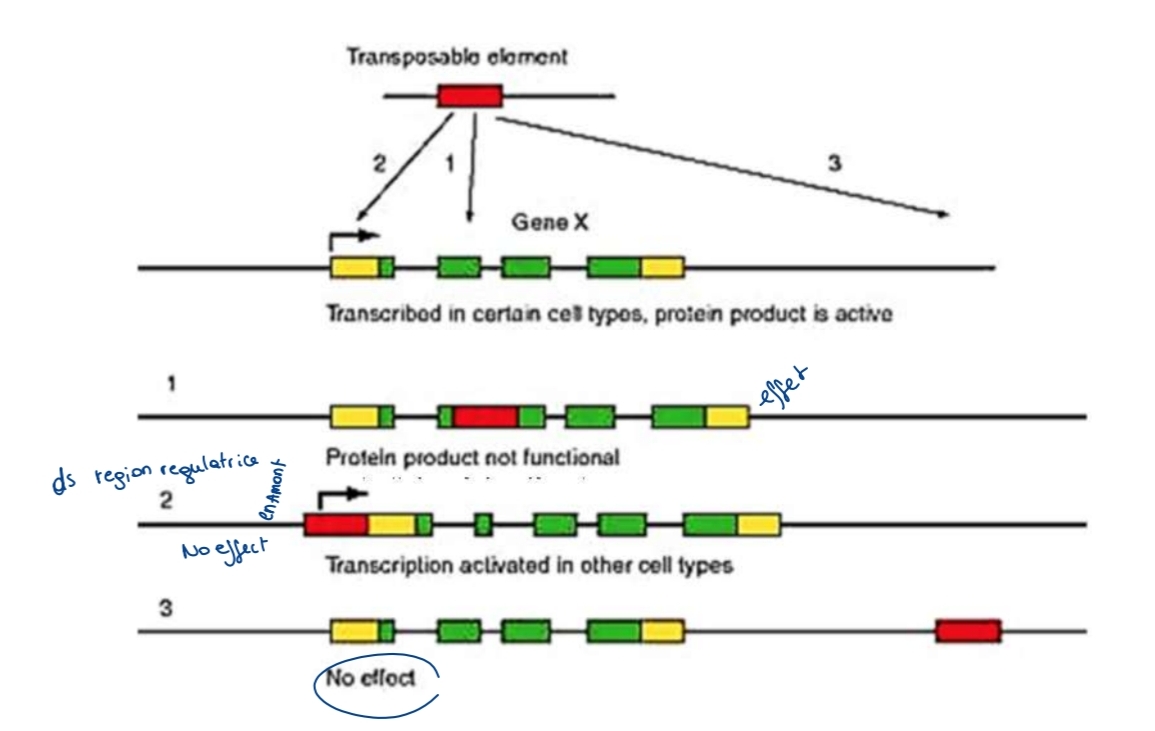
Effets délétères ET
Insertion → gène KO
Activation ectopique transcription
Modification épissage / régulation
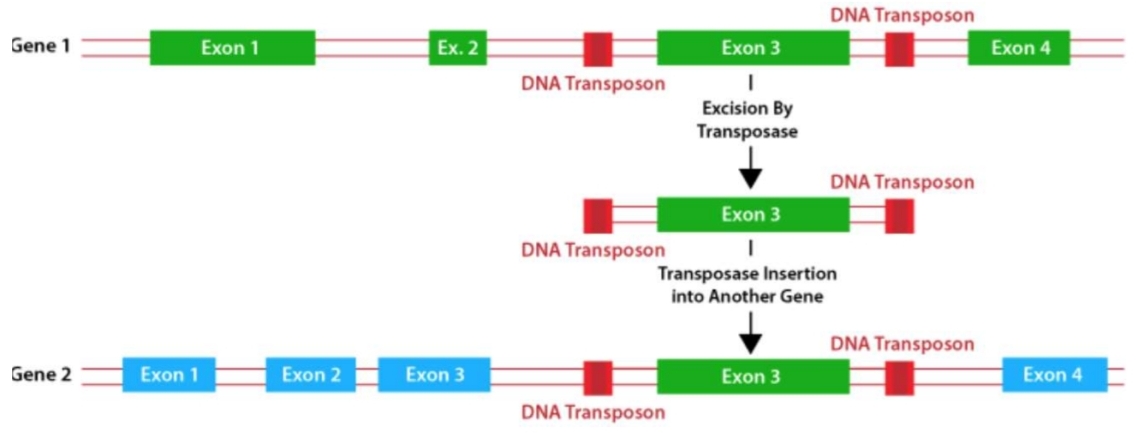
Exon shuffling
Double recombinaison via Alu (SINE)
2 exxon vont echanger ds 2 gène différent vias seq Alu
2 gènes → 2 nouveaux gènes
Nouvelle combinaison exons

Brassage d’exons par transposition
crée nv gène
Nouvelle fonction possible
≈ 19% exons eucaryotes
ET : part mutations
Humain 0,1–0,2% mutations spontanées
Souris ~10%
Drosophile ~50%
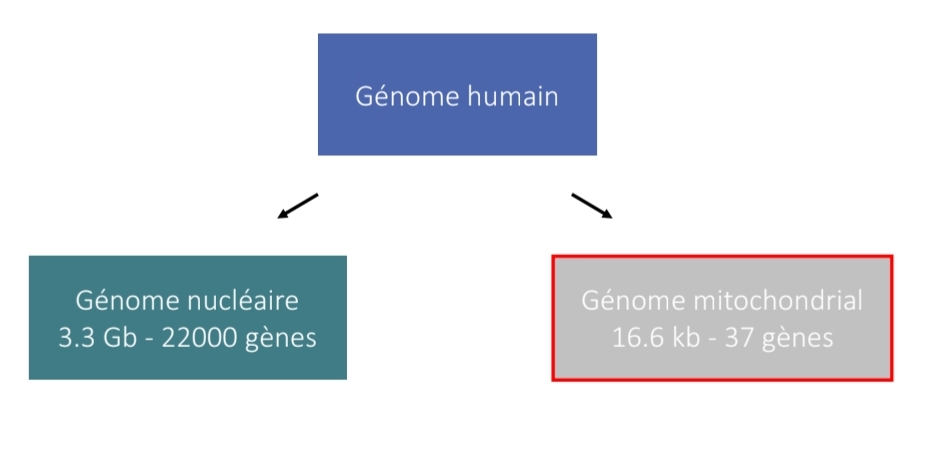
Mitochondrie : ADNmt
Seul compartiment eucaryote avec ADN propre
Respiration + ATP + apoptose
dynamique + svt hétéroplasmique
ADN circulaire bicaténaire
ADNmt : gènes
13 protéines chaîne respiratoire
2 ARNt + 2 ARNr
3 promoteur
Brin H code = brin lourd
12 sous-unités chaîne respiratoire
ARNr
14 ARNt
Brin L code = brin léger
1 sous-unité complexe I
8 ARNt
ADNmt : copies
nbr varie selon localisation cell
4à5 mito hépatique humaine
6500 ds myocarde
mutation :
Cancer de la prostate, Myopathies, Encéphalopathies, Leigh Syndrome
Mutations Ponctuelles: Neuropathie optique héréditaire de Lebe , épilepsie myoclonique avec fibres rouges déchiquetées (MERRF).
PCR =
Amplification ADN in vitro
ADN polymérase + matrice
PCR : composants
ADN matrice
Amorces
dNTP
ADN polymérase
PCR : 3 étapes
Dénaturation
Appariement (hybridation)
Élongation
PCR : amplification
Exponentielle
Après n cycles → 2^n copies
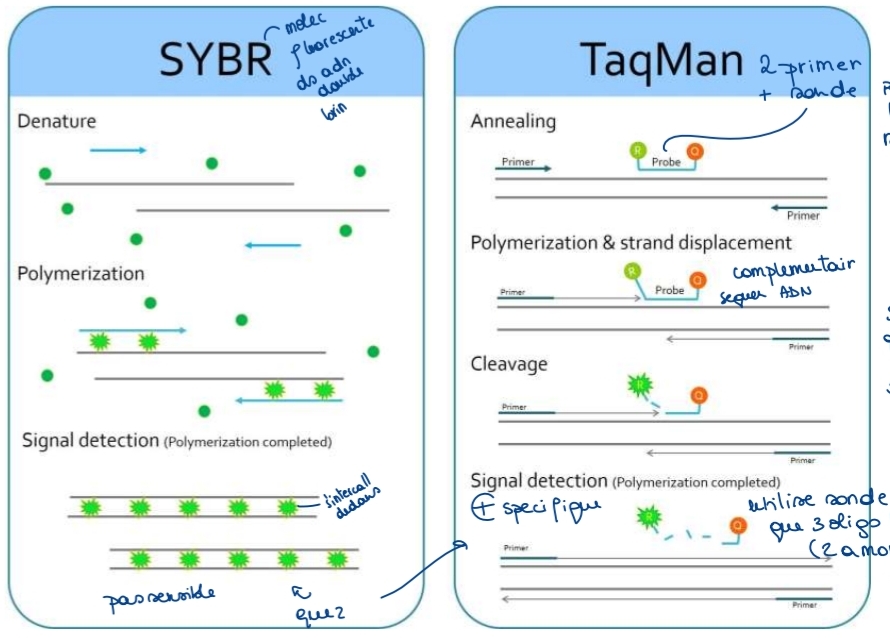
qPCR (temps réel)
Quantification fluorescence / cycle
SYBR Green (intercalant)
TaqMan (sonde spécifique)
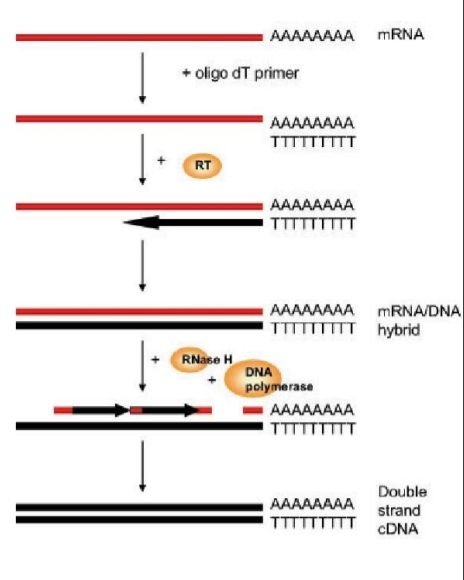
RT-PCR
ARN → ADNc (reverse transcriptase)
Amorce (oligo-dT / hexamères)
Puis PCR
RT-PCR : applications
Expression génique
Charge virale virus ARN
PCR digitale
Partitionnement milliers réactions
Sensibilité ↑
Variants rares
Microsatellites + PCR : usages
Médecine légale
Suivi animaux
Typage cancers
Instabilité microsatellites (MSI)
Défaut MMR (réparation mismatch)
Répétitions anormales ADN tumoral
MSI : détection
PCR 5 marqueurs microsatellites
Comparaison tumoral vs sain
MSI : intérêt clinique
Identifier prédisposition familiale
Adapter traitement
Tumeur hypermutée → TILs ↑ → immunothérapie ↑