TEMA 7: RIBOSOMAS
5.0(2)
Card Sorting
1/20
Study Analytics
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
21 Terms
1
New cards
1\. Caractéristicas de los ribosomas
Son ARN+PROTEINAS, se encuentran libres o agrupados en el citosol, unidos al retículo endoplasmático (RER) y en las mitocondrias.
2
New cards
Función de los ribosomas
Síntesis de proteínas, es el lugar de interacción del ARNm y ARNt para la producción de las cadenas polipeptídicas.
Sintesis: Las dos subunidades se sintetizan por separado en el nucleolo pero sin embargo ejercen su función de síntesis en el citosol.
Sintesis: Las dos subunidades se sintetizan por separado en el nucleolo pero sin embargo ejercen su función de síntesis en el citosol.
3
New cards
2\. Estructura y composición
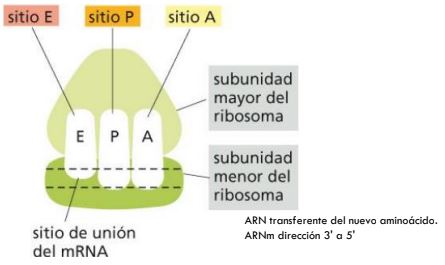
Los ribosomas contienen un sitio de unión para el ARNm y 3 sitios de unión para los ARNt: el **sitio P** que une el ARNt que lleva la cadena polipeptídica en formación; el **sitio A** une el ARNt que lleva el aminoácido que va a unirse y el **sitio E** de salida de los ARNt vacíos
4
New cards
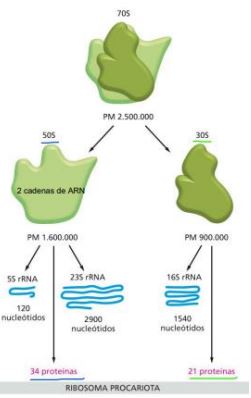
3\. Ribosomas en procariotas
• E. coli. (ribosomas más estudiados)
• 70 S.
• Pm: 2500 kDa.
• Están compuestos por más ARNr que proteínas
• ARNr: 65 %. (forma una doble helicoide y rodea las proteínas
• Proteínas tanto ácidas como básicas: 35 %.
• Predominio de bases A y G metiladas.
• 70 S.
• Pm: 2500 kDa.
• Están compuestos por más ARNr que proteínas
• ARNr: 65 %. (forma una doble helicoide y rodea las proteínas
• Proteínas tanto ácidas como básicas: 35 %.
• Predominio de bases A y G metiladas.
5
New cards
Ribosomas en procariotas SUBUNIDADES
• Subunidad mayor (50 S) 3 salientes a modo de picos :
* ARNr 23 S.
* ARNr 5S.
* 34 proteínas.
• Subunidad menor (30 S) muestra dos lóbulos :
* ARNr 16 S.
* 21 proteínas.
De las 34 y 21 proteínas de ambas subunidades, sólo una es común en ambas.
* ARNr 23 S.
* ARNr 5S.
* 34 proteínas.
• Subunidad menor (30 S) muestra dos lóbulos :
* ARNr 16 S.
* 21 proteínas.
De las 34 y 21 proteínas de ambas subunidades, sólo una es común en ambas.
6
New cards
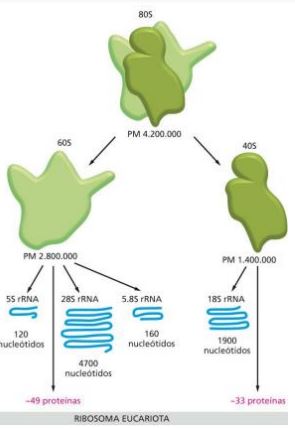
Ribosomas en eucariotas
• 80 S.
• Pm: 4200 kDa.
• Existen pequeñas diferencias entre especies
• Están compuestos por más proteínas que ARNr
• ARNr: 40 %.
• Proteínas: 60 %.
• Ribosas metiladas.
• Pm: 4200 kDa.
• Existen pequeñas diferencias entre especies
• Están compuestos por más proteínas que ARNr
• ARNr: 40 %.
• Proteínas: 60 %.
• Ribosas metiladas.
7
New cards
Ribosomas en eucariotas SUBUNIDADES
• Subunidad mayor (60 S):
* ARNr 28 S.
* ARNr 5,8 S.
* ARNr 5 S.
* 49 proteínas.
• Subunidad menor (40 S):
* ARNr 18 S.
* 33 proteínas.
No tienen proteínas en común. Las mitocondrias y los plastos también contienen ribosomas que se asemejan a los de procariotas.
* ARNr 28 S.
* ARNr 5,8 S.
* ARNr 5 S.
* 49 proteínas.
• Subunidad menor (40 S):
* ARNr 18 S.
* 33 proteínas.
No tienen proteínas en común. Las mitocondrias y los plastos también contienen ribosomas que se asemejan a los de procariotas.
8
New cards
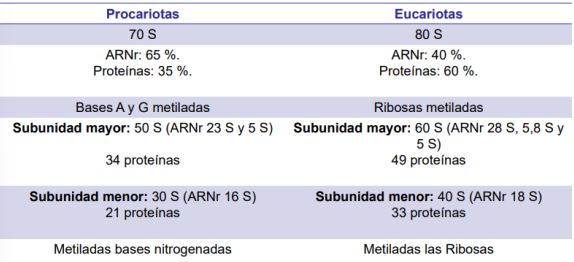
Diferencias entre procariotas y eucariotas
Tabla resumen
9
New cards
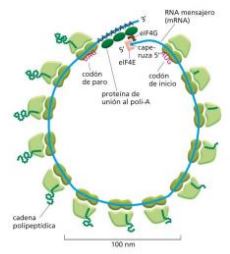
4\. Polisomas o polirribosomas
• Asociación de ribosomas a una hebra de ARNm.
• Espiral.
• Función: síntesis proteínas en citosol y REr.
• No de ribosomas en polisoma según Pm del péptido a sintetizar:
* Ej.:150 aa (aminoácidos) son 5 ribosomas
• Síntesis 5 ́ → 3 ́.
Siempre dirección 5' a 3' , empieza subunidad pequeña y después la subunidad grande.
Por cada ribosoma que termina la síntesis se une otro.
Para la síntesis de proteínas intervienen las dos subunidades(no siempre las mismas).
• Espiral.
• Función: síntesis proteínas en citosol y REr.
• No de ribosomas en polisoma según Pm del péptido a sintetizar:
* Ej.:150 aa (aminoácidos) son 5 ribosomas
• Síntesis 5 ́ → 3 ́.
Siempre dirección 5' a 3' , empieza subunidad pequeña y después la subunidad grande.
Por cada ribosoma que termina la síntesis se une otro.
Para la síntesis de proteínas intervienen las dos subunidades(no siempre las mismas).
10
New cards
5\. Destino de las proteínas sintetizadas
• Proteínas solubles en el citosol.
• Proteínas periféricas de la membrana plasmática (espectrina, actina, enzimas).
• Proteínas constituyentes del citoesqueleto (actina, tubulina, filamentos intermedios ).
• Proteínas con destino a mitocondrias.
• Enzimas de peroxisomas.
• Proteínas nucleares (Histonas, láminas).
• Proteínas periféricas de la membrana plasmática (espectrina, actina, enzimas).
• Proteínas constituyentes del citoesqueleto (actina, tubulina, filamentos intermedios ).
• Proteínas con destino a mitocondrias.
• Enzimas de peroxisomas.
• Proteínas nucleares (Histonas, láminas).

11
New cards
Síntesis en REr:
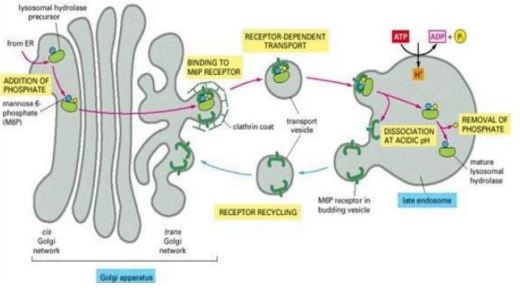
• Modificaciones postraduccionales -> Glicosilación.
• 1 o varias secuencias señal -> clasificación y determinación de destino -> receptores específicos en orgánulo diana.
• Destinos: Golgi, lisosomas, mitocondrias, peroxisomas cloroplastos, y núcleo.
• Ausencia de péptido : libre en el citosol •Vesículas de secreción.
• 1 péptido señal -> amino terminal y se une al orgánulo correspondiente.
• Proteínas transmembrana de paso múltiple: varios péptidos señal -> lugares de inserción a membrana.
• 1 o varias secuencias señal -> clasificación y determinación de destino -> receptores específicos en orgánulo diana.
• Destinos: Golgi, lisosomas, mitocondrias, peroxisomas cloroplastos, y núcleo.
• Ausencia de péptido : libre en el citosol •Vesículas de secreción.
• 1 péptido señal -> amino terminal y se une al orgánulo correspondiente.
• Proteínas transmembrana de paso múltiple: varios péptidos señal -> lugares de inserción a membrana.
12
New cards
6\. Chaperonas
plegamiento y reparación.
13
New cards
Chaperonas moleculares
• Conjunto de proteínas que ayudan al plegamiento de las proteínas recién sintetizadas.
• Las chaperonas no forman parte de la estructura primaria de las proteínas funcional que modifican, solo
se unen a ellas para ayudar en el plegamiento, ensamblado y transporte de las proteínas sintetizadas.
• Los cambios conformacionales pueden estar afectados por varias chaperonas que trabajan de forma
coordinada
• Las chaperonas no forman parte de la estructura primaria de las proteínas funcional que modifican, solo
se unen a ellas para ayudar en el plegamiento, ensamblado y transporte de las proteínas sintetizadas.
• Los cambios conformacionales pueden estar afectados por varias chaperonas que trabajan de forma
coordinada
14
New cards
Chaperonas moleculares FUNCIONES
– Favorecen el plegamientos de proteínas sintetizadas de novo.
– Evitan el plegamiento incorrecto o la formación de agregados y permiten que la cadena polipeptídica
adquiera una conformación activa.
– Reparación del plegamiento de proteínas desnaturalizadas.
– Colabora en el plegamiento de proteínas que han sido traslocadas hacia algún compartimento celular.
– Evitan el plegamiento incorrecto o la formación de agregados y permiten que la cadena polipeptídica
adquiera una conformación activa.
– Reparación del plegamiento de proteínas desnaturalizadas.
– Colabora en el plegamiento de proteínas que han sido traslocadas hacia algún compartimento celular.
15
New cards
Tipos de Chaperonas
• Chaperonas se unen a polipéptidos no plegados -> transporte a orgánulos -> las chaperonas del interior
del orgánulo -> finalizan el plegamiento.
del orgánulo -> finalizan el plegamiento.
16
New cards
PROTEÍNAS DE SHOCK TÉRMICO o del ESTRÉS (heat shock proteins)
Se sintetizan en cantidad elevada cuando las células se exponen a situaciones de estrés como altas temperaturas, estrés celular, calor, isquemia, acidosis, metales pesados, etc. Así a altas temperaturas (42 oC para células que viven normalmente a 37 oC) se ha visto un incremento de chaperonas en respuesta a un aumento de proteínas mal plegadas como consecuencia del aumento de temperatura- con el fin de recuperar el plegamiento correcto de estas proteínas. • Hsp (Heat shock proteins): Hsp 60 y Hsp70 -> solubilizan y repliegan proteínas desnaturalizadas o mal plegadas mediante la hidrólisis de ATP. • No proporcionan una información adicional para el plegamiento -> secuencia aminoácidos.
* Hsp 70: actúan en el primer momento de la vida de la proteína, antes de que ésta deje el ribosoma.
* Hsp 60: actúa una vez que la proteína está completamente sintetizada y forman una estructura en barril que aísla la proteína procediendo a su reconfiguración.
* Hsp 70: actúan en el primer momento de la vida de la proteína, antes de que ésta deje el ribosoma.
* Hsp 60: actúa una vez que la proteína está completamente sintetizada y forman una estructura en barril que aísla la proteína procediendo a su reconfiguración.
17
New cards
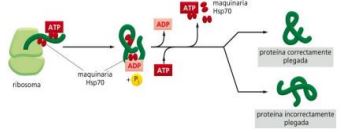
* Hsp 70:
Actúan en el primer momento de la vida de la proteína, antes de que ésta deje el ribosoma.
18
New cards
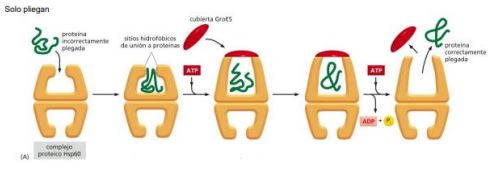
* Hsp 60:
Actúa una vez que la proteína está completamente sintetizada y forman una estructura en barril que aísla la proteína procediendo a su reconfiguración.
19
New cards
7. Degradación de proteínas
Tasa de degradación depende del tipo de proteína:
Proteínas de vida corta tienen una alta tasa de degradación. Se degradan por la vía de
la Ubiquitina-proteasoma (destruye proteínas de vía corta).
* Proteínas reguladoras (factores de transcripción).
* Proteínas dañadas o defectuosas. Proteínas de vida larga: Proteínas de membrana.
Se degradan por vía lisosómica (destruye proteínas de vía larga).
Proteínas de vida corta tienen una alta tasa de degradación. Se degradan por la vía de
la Ubiquitina-proteasoma (destruye proteínas de vía corta).
* Proteínas reguladoras (factores de transcripción).
* Proteínas dañadas o defectuosas. Proteínas de vida larga: Proteínas de membrana.
Se degradan por vía lisosómica (destruye proteínas de vía larga).
20
New cards
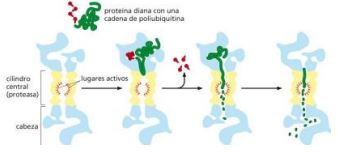
Vía ubiquitina-proteasoma
• Proteínas a degradar -> señal -> determina su destrucción por enzimas proteolíticas.
• Unión de complejo multienzimático -> múltiples copias de **ubiquitina**.
\- Ubiquitina se une por su grupo carboxiterminal al grupo amino de un residuo de lisina de la proteína diana que va ser degradada.
\- Adición de moléculas de ubiquitina adicionales para formar una cadena.
• Proteína ubiquitinada -> **Proteasoma** -> hidrolizan el ATP y capturan las proteínas ubiquitinadas.
• Destrucción de proteínas ubiquitinadas por proteasas.
• Unión de complejo multienzimático -> múltiples copias de **ubiquitina**.
\- Ubiquitina se une por su grupo carboxiterminal al grupo amino de un residuo de lisina de la proteína diana que va ser degradada.
\- Adición de moléculas de ubiquitina adicionales para formar una cadena.
• Proteína ubiquitinada -> **Proteasoma** -> hidrolizan el ATP y capturan las proteínas ubiquitinadas.
• Destrucción de proteínas ubiquitinadas por proteasas.
21
New cards
Lisosoma
• Degrada proteínas de vida larga como pueden ser las proteínas de membrana