Kaarten: moleculaire biologie H17: Replicatie en DNA foutenherstel | Quizlet
1/68
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
69 Terms
17.1 DNA replicatie
Welke cellen delen in ons lichaam?
Alleen stamcellen, een random werkende spiercel gaat dus niet terwijl je 100kg aan het benchen bent effe mitose knallen
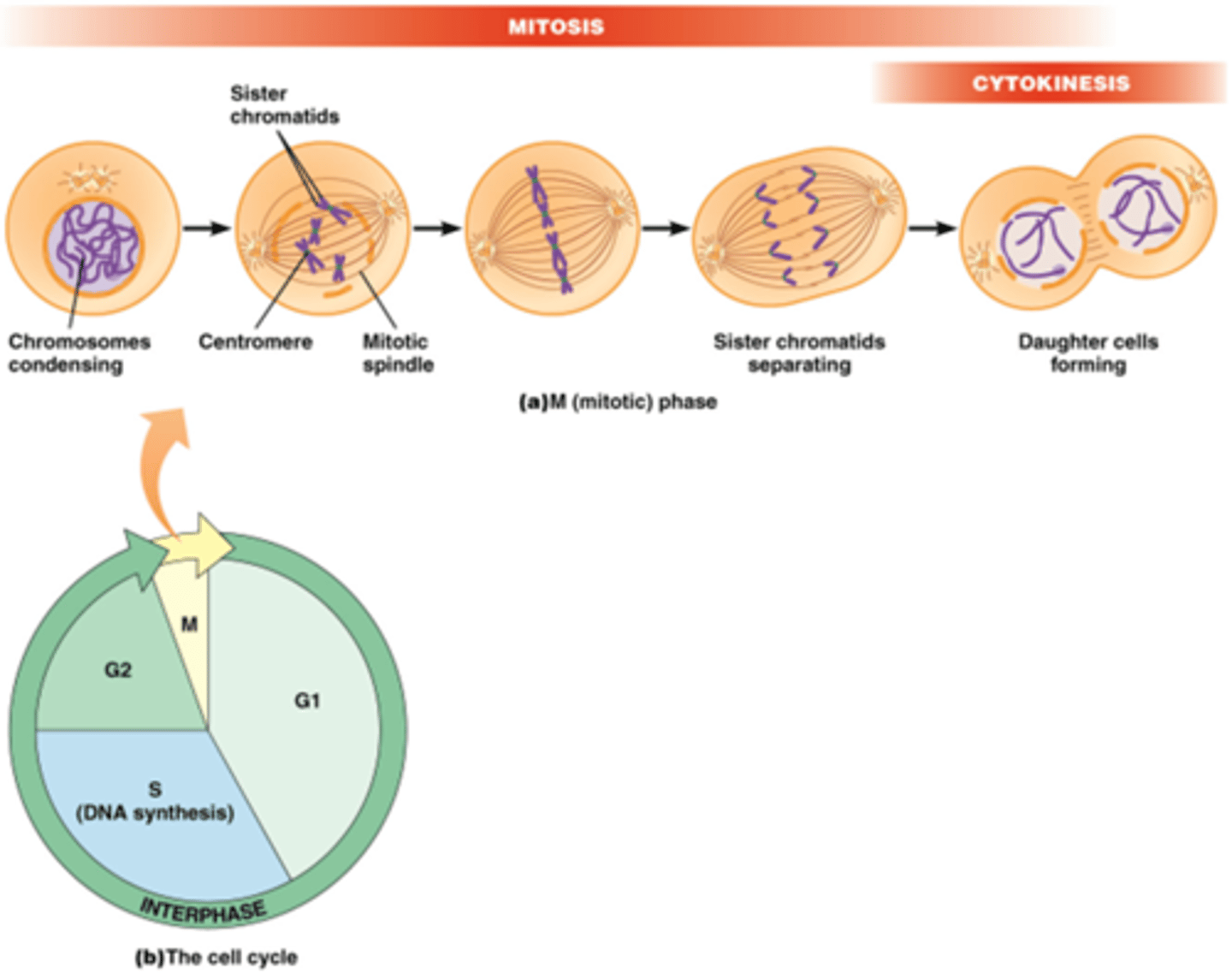
In welke fase van de celcyclus vindt de DNA replicatie plaats?
Celcyclus:
M fase (mytose en cytokinese) en interfase
Interfase
-G1
-S: DNA replicatie! (6-8h)
-G2
G=GAP
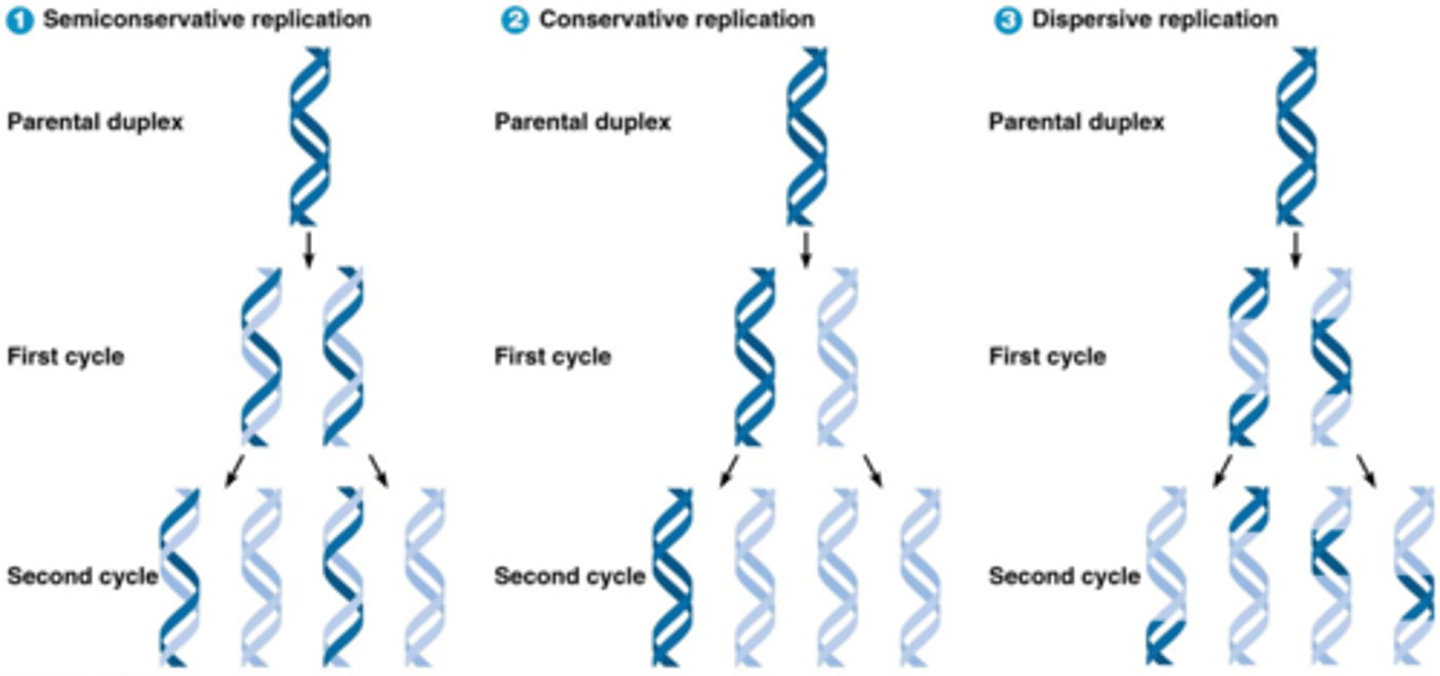
Welke 3 hypotheses waren over DNA replicatie (parentale strengen en nieuwe strengen). Wat was de juiste hypothese?
semi-conservatief is juist bij DNA-replicatie!
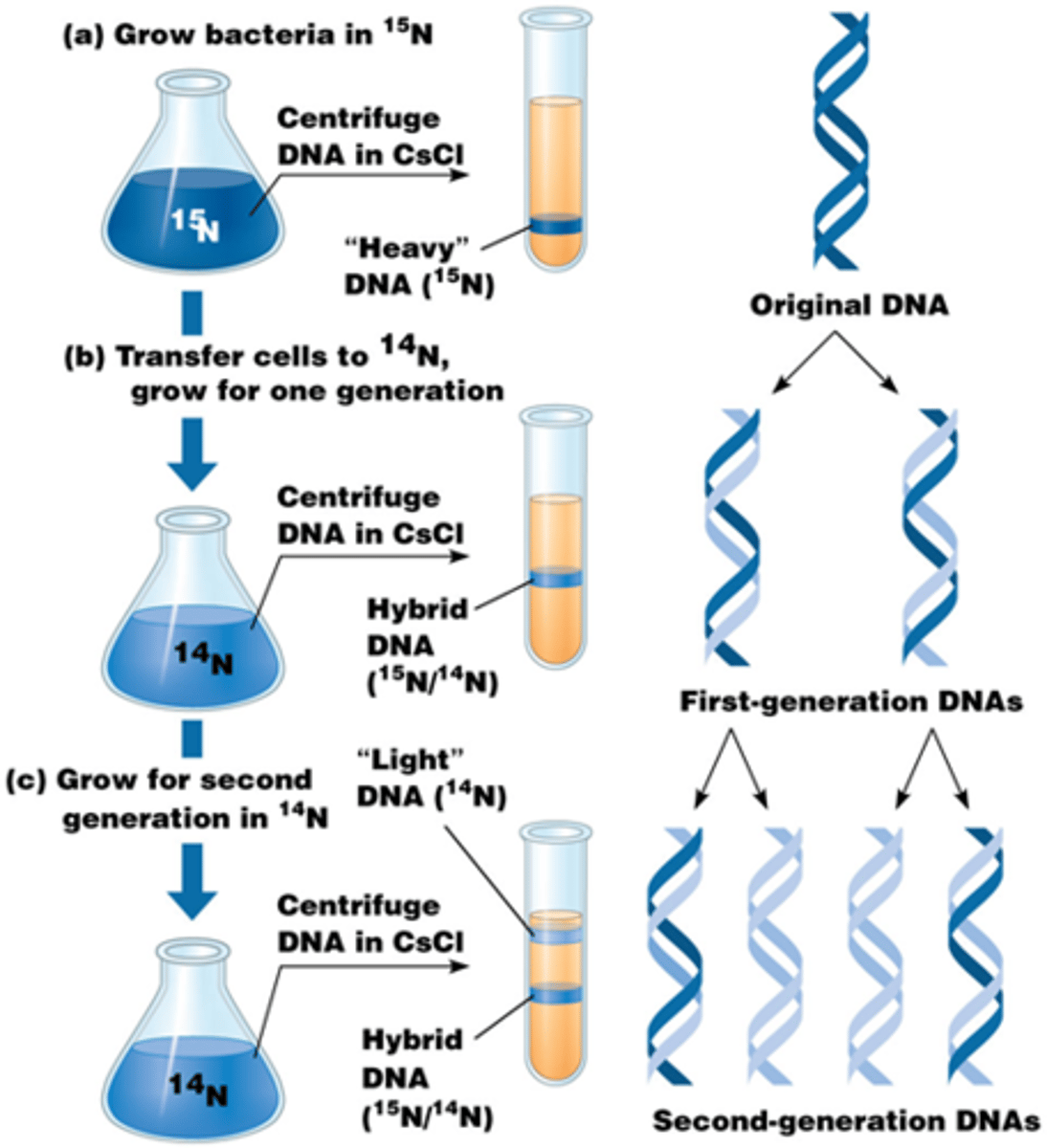
Hoe zijn onderzoekers erachter gekomen dat DNA replicatie semi-conservatief is bij prokaryoten?
Meselson & Stahl experiment:
-E. coli werd gekweekt in een medium met zware stikstof ->15N DNA
-De bacteriën werden overgezet naar 14N, zodat nieuw DNA alleen nog met de lichtere stikstof zou worden gemaakt
-ze pakken DNA van elke sample en ze centrifugeren, dit scheidt het zware en lichte DNA op basis van hun dichtheid
-na 1 replicatie twijfelden ze tussen semiconservatief of dispensive maar na een een tweede keer wisten ze dat het semi-conservatief was!
->dit is bij bacterien! DNA van prokaryoten is heel anders dan eukaryotisch DNA (lineair, chromosomen)
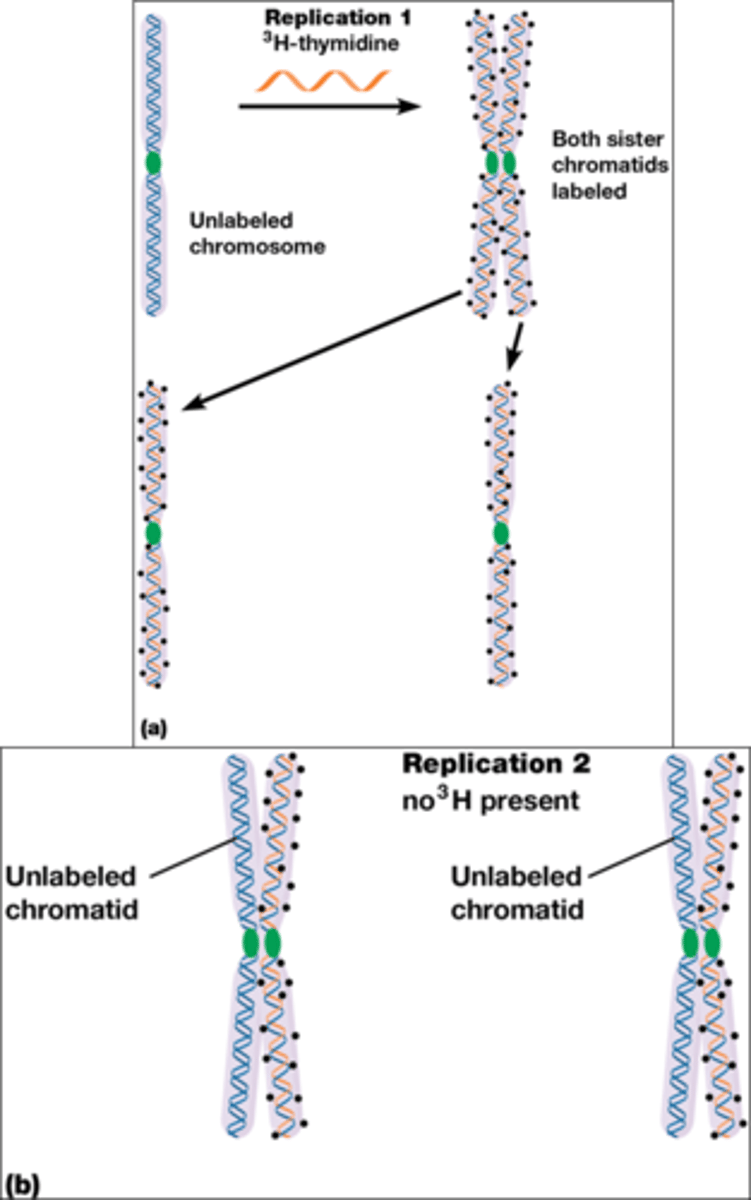
Hoe zijn onderzoekers erachter gekomen dat DNA replicatie semi-conservatief is bij eukaryoten?
-Een chromosoom werd gerepliceerd in aanwezigheid van tritium (3H: 1 proton en 2 neutronen) gelabeled thymidine
->1 streng radioactief + 1 streng niet radioactief
-bij de mitose gaan de zusterchromatiden gescheiden worden
-bij de 2de replicatie gaat het zonder tritium gebeuren
->1 radioactieve zusterchromatide + 1 normale zusterchromatide
Bij de eukaryoten is de DNA replicatie dus ook semiconservatief!
Wat is een ORI?
Origin of replication: de plek waar de DNA replicatie begint, ze heeft altijd een specifieke nucleotiden sequentie: Consensus sequentie
20 000 bij eukaryoten en 1 bij prokaryoten!
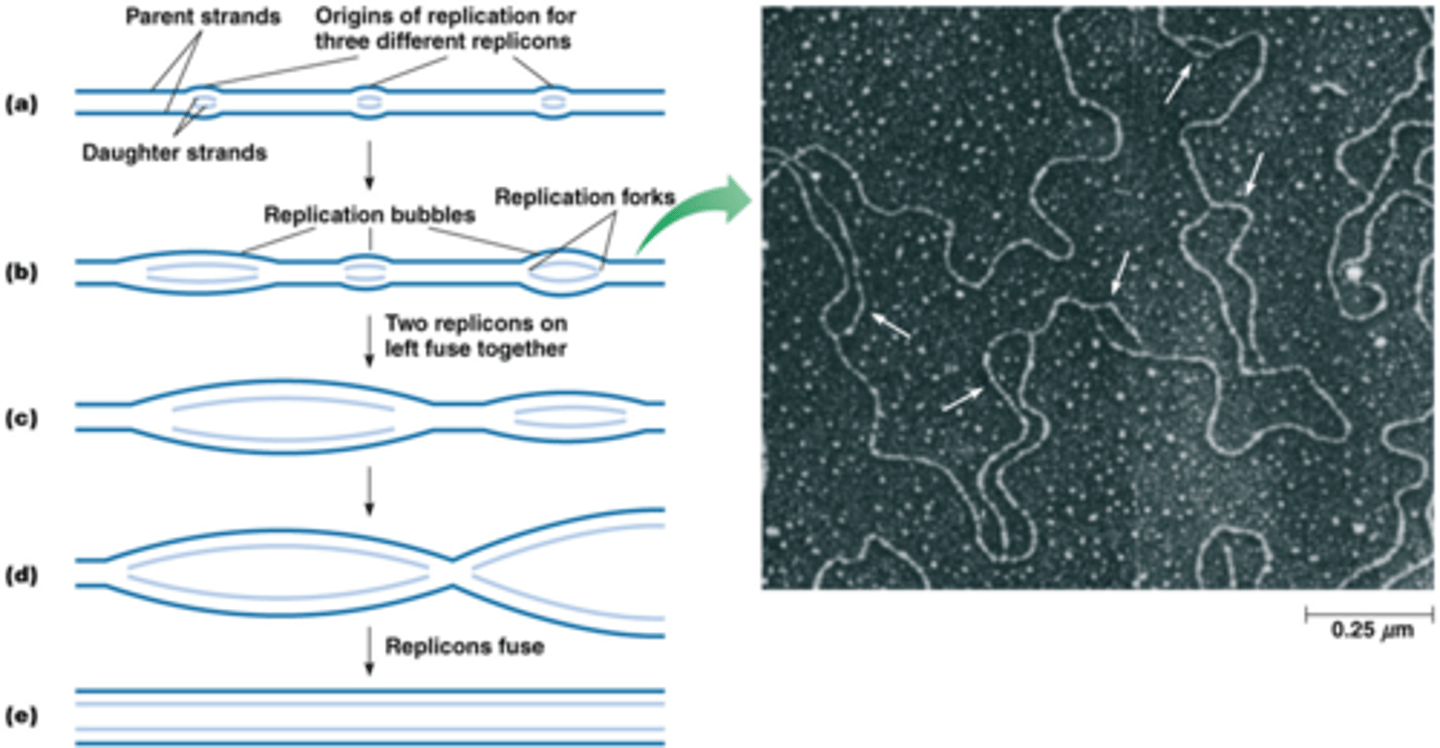
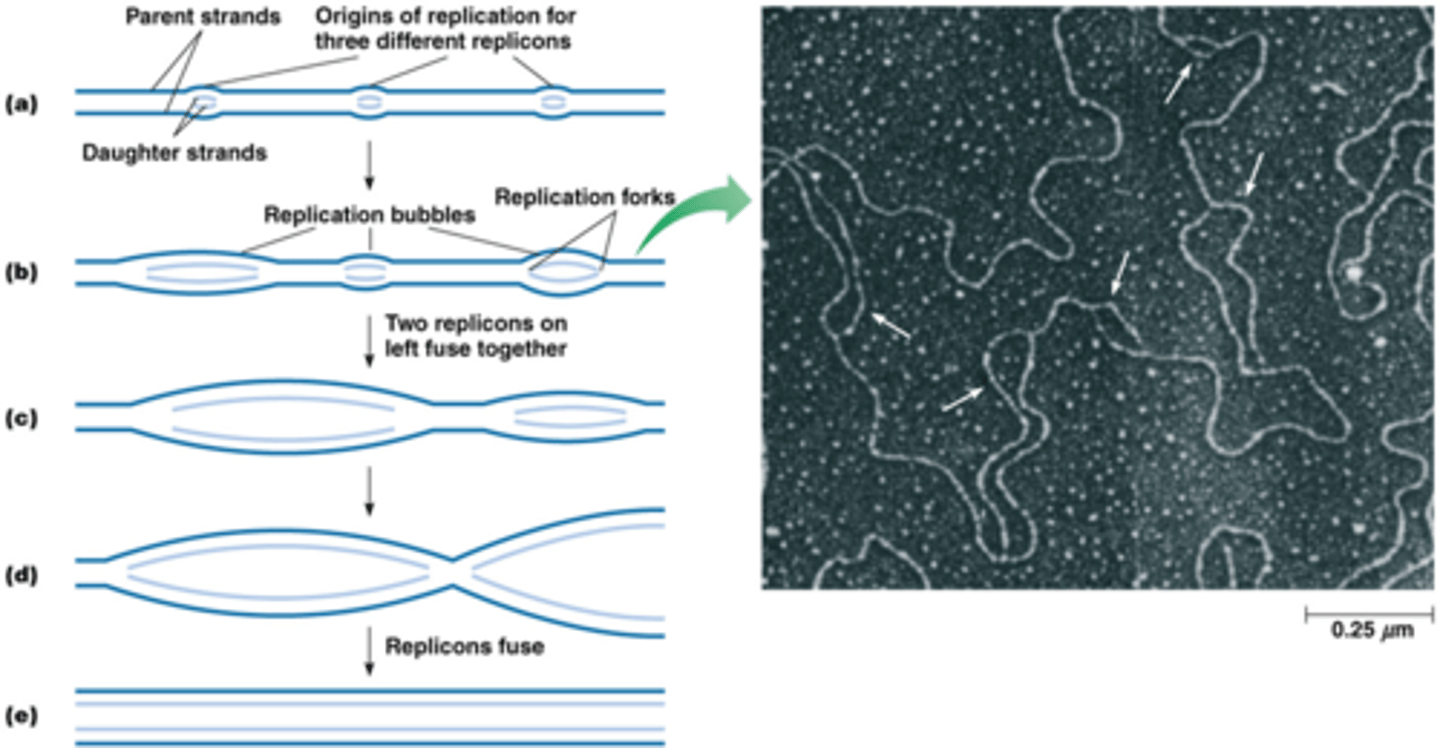
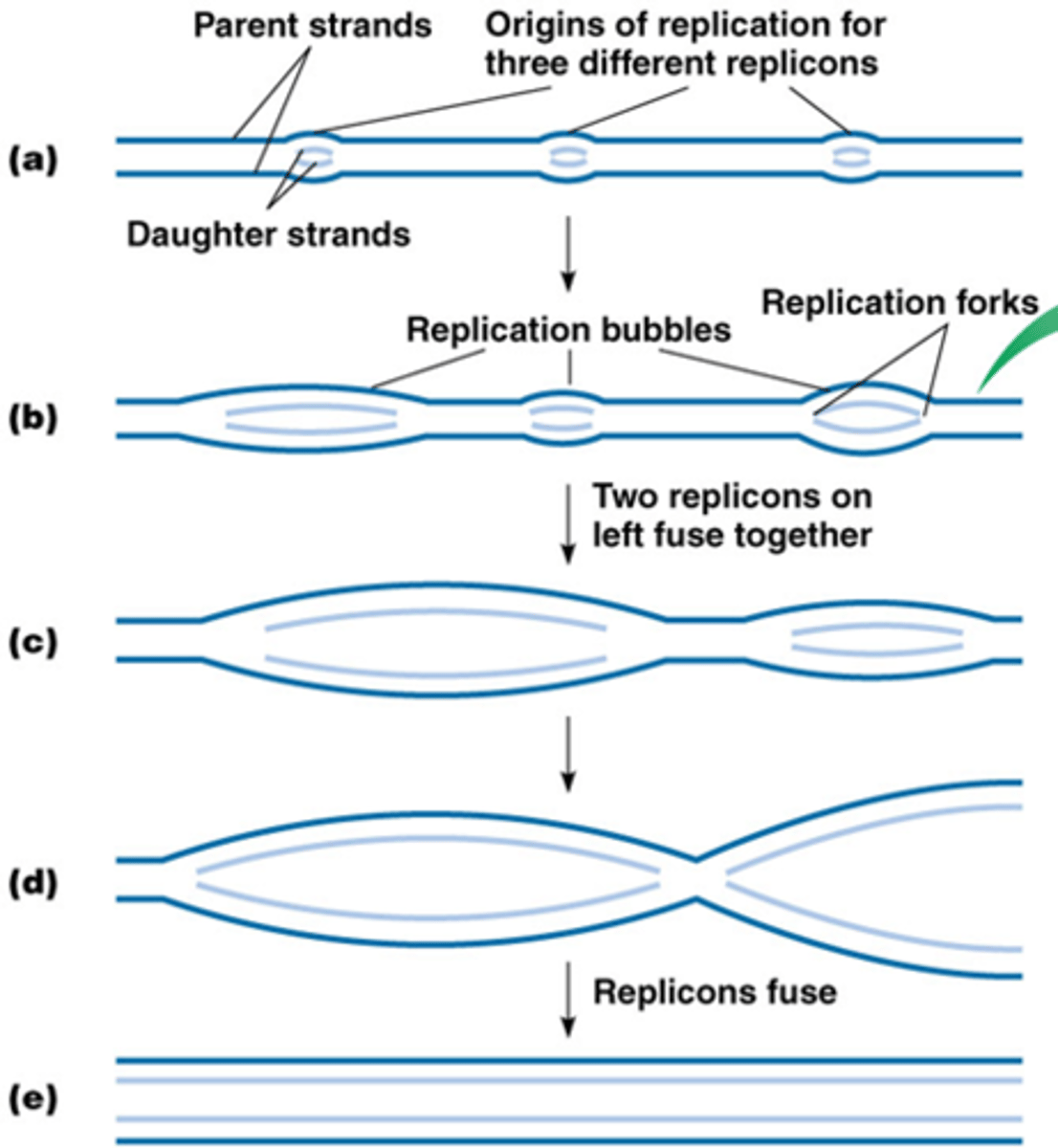
Wat is een replicon?
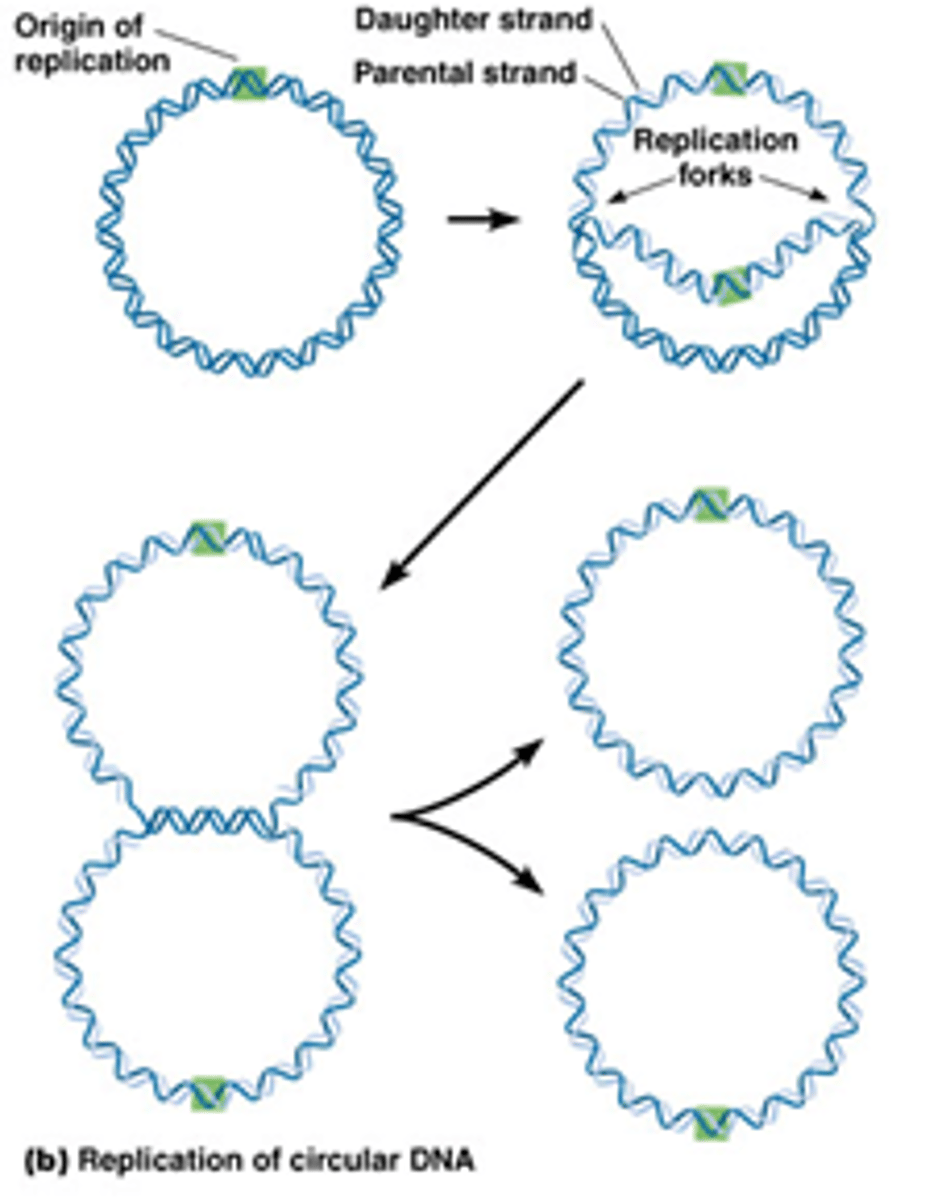
replicon: 1 replicatiebubbel met 2 vorken en wanneer 2 replicon's elkaar tegen komen gaan ze fuseren met elkaar
Een replicon wordt gevormd wanneer een enkele oorsprong van replicatie zich opent om een replicatiebel te vormen-> er is 1 replicon per ORI (quiz)
Hoe verloopt de DNA replicatie bij bacterien?
Thèta-replicatie:
-ORI= AT-rijke regio
-verloopt bidirectioneel* via twee replicatievorken
-vormt intermediaire thèta structuur (links beneden)
-Replicatie gekoppeld aan splitsing (rechts beneden)
*links en rechts
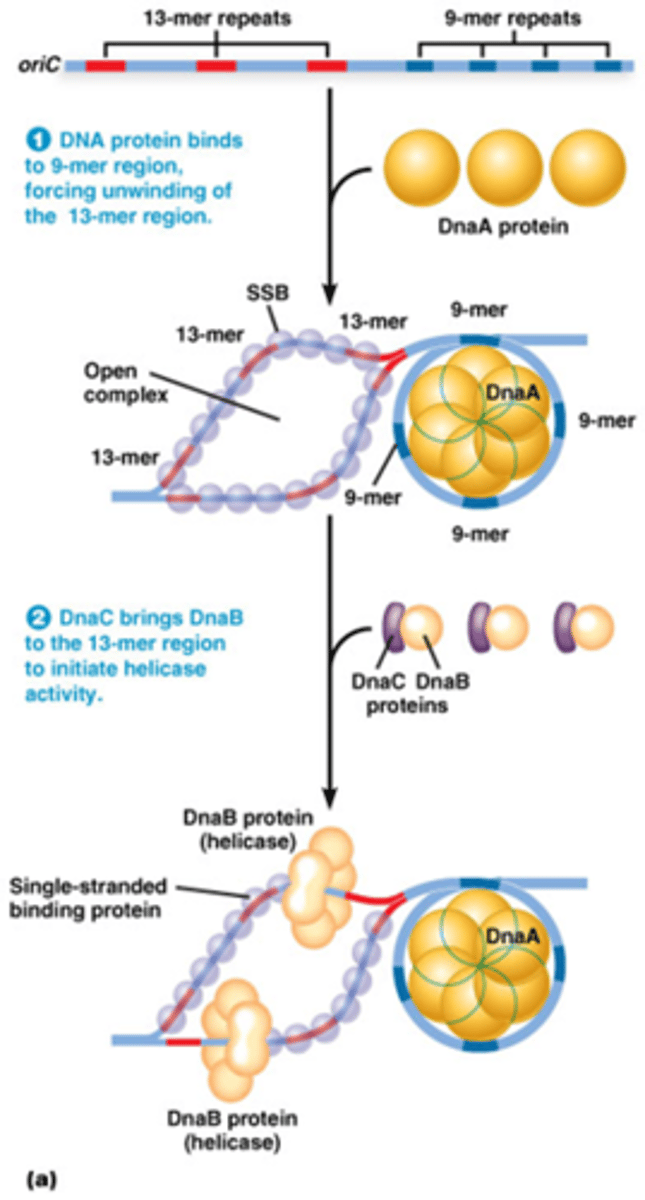
Hoe start de replicatie bij bacteria?
1. Dna-A (een eiwit) bindt aan 9-meer sequenties van de ORI
->ontwinding van 13-meer sequenties.
2. SSB (Single-Strand Binding protein) bindt aan ssDNA en voorkomt re-hybridisatie van 2 ssDNA strengen
3. DnaB (DNA helicase) gerecruteerd via DnaC (een helicase-lader): laat bidirectionele replicatie toe.
tekening legt het weeral perfect uit
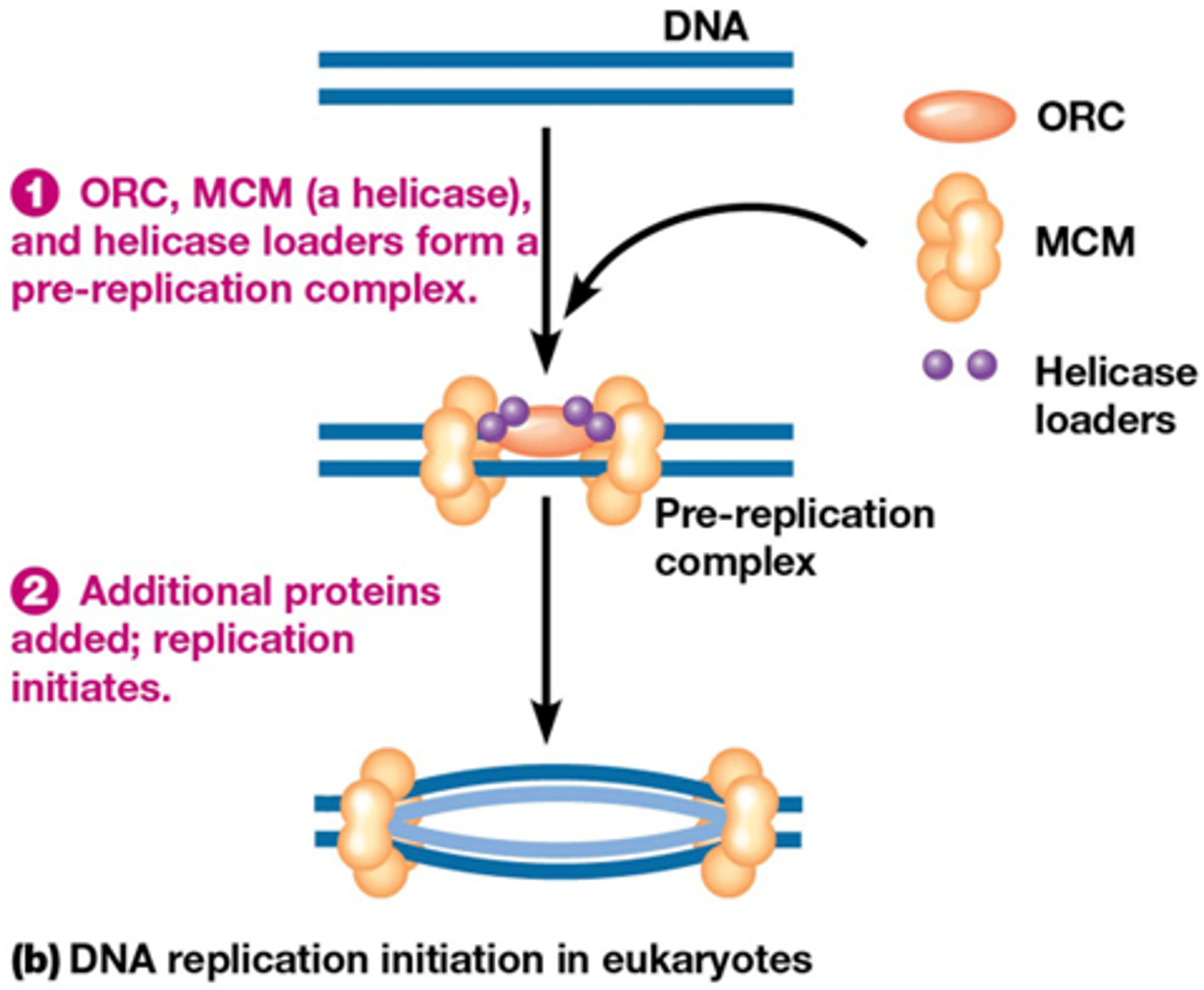
Hoe start de replicatie bij eukaryoten?
Vorming van pre-replicatie complex
(1) ORC (origin of Recognition Complex) bindt aan ORI
(2) helicase-laders binden aan ORC
(3) recrutering van MCM (MiniChromosome Maintenance) complex , het heeft helicase activiteit
vorming pre-replicatie complex op de ORI tijdens de G1-fase van de celcyclus, gekend als replicatie-licentie
In het begin van de S-fase omvorming tot ‘replicatie complex’ door recrutering van bijkomende eiwitten in het begin van de S-fase. (Hfd 24)
Wat is de functie van de pre-replicatie complex, wanneer worden ze geactiveerd?
Ze zorgen ervoor dat alle DNA gerepliceerd wordt en dat het allemaal maar 1 keer gebeurt! -> complexen gemaakt op voorhand zodat de cel weet waar te beginnen in de S fase
In de S fase wordt het pre-replicatie complex geactiveerd
Waardoor wordt de lengte van de S-fase beinvloed?
Lengte van S-fase = bepaald door aantal replicons + snelheid van activatie van pre-replicatie complexen (snel in embryonale cellen, traag in adulte cellen).
De activatie van pre-replicatie complexen gebeurt niet gelijktijdig
->veel later voor heterochromatine (centromeren en telomeren)
Door welk enzyme wordt DNA synthese gekatalyseerd, wat doet het?
DNA polymerase verlengt een DNA streng aan het 3’ OH uiteinde (start bij een RNA primer)
Het heeft dNTP's (deoxynucleoside trifosfaat) als substraat
DNA synthese is gekoppeld aan de hydrolyse/breking van 2 fosfoanhybride bindingen (1 waarbij PPi vrijkomt en 1 van de splitsing van PPi)
katalyseert de vorming van fosfo-diesterbinding*
*Een fosfodiesterbinding verbindt een fosfaatgroep met twee moleculen via zuurstofatomen.
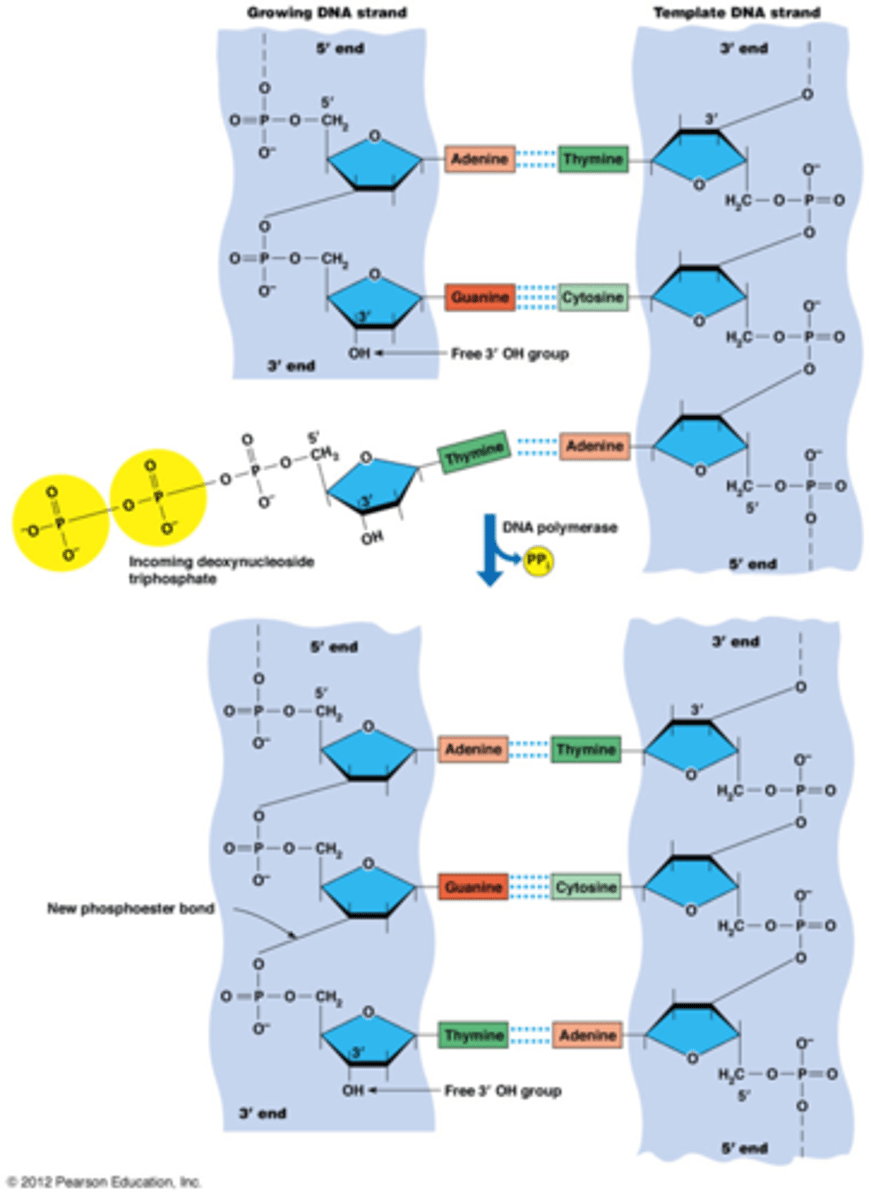
Wat is het grote verschil tussen DNA polymerase en RNA polymerase?
RNA polymerase kan de novo een streng creeren, die maakt zelf de fosfodiester binding en is gestart
DNA polymerase heeft een vrije 3' OH groep nodig voor hij kan starten, primase gaat een RNA primer hiervoor geven
Tot welke klasse van enzymen behoort DNA polymerase
DNA polymerase behoort tot de klasse van de transferasen enzymen (EC 2)
wat is de deltaG van de vorming van de fosfodiesterbinding?
het is een endergonische reactie (deltaG is positief) die kan plaatsvinden door de koppeling met de hydrolyse van PPi
PPi zijn die 2 gele fosfaatgroepen die dus vrijkomen
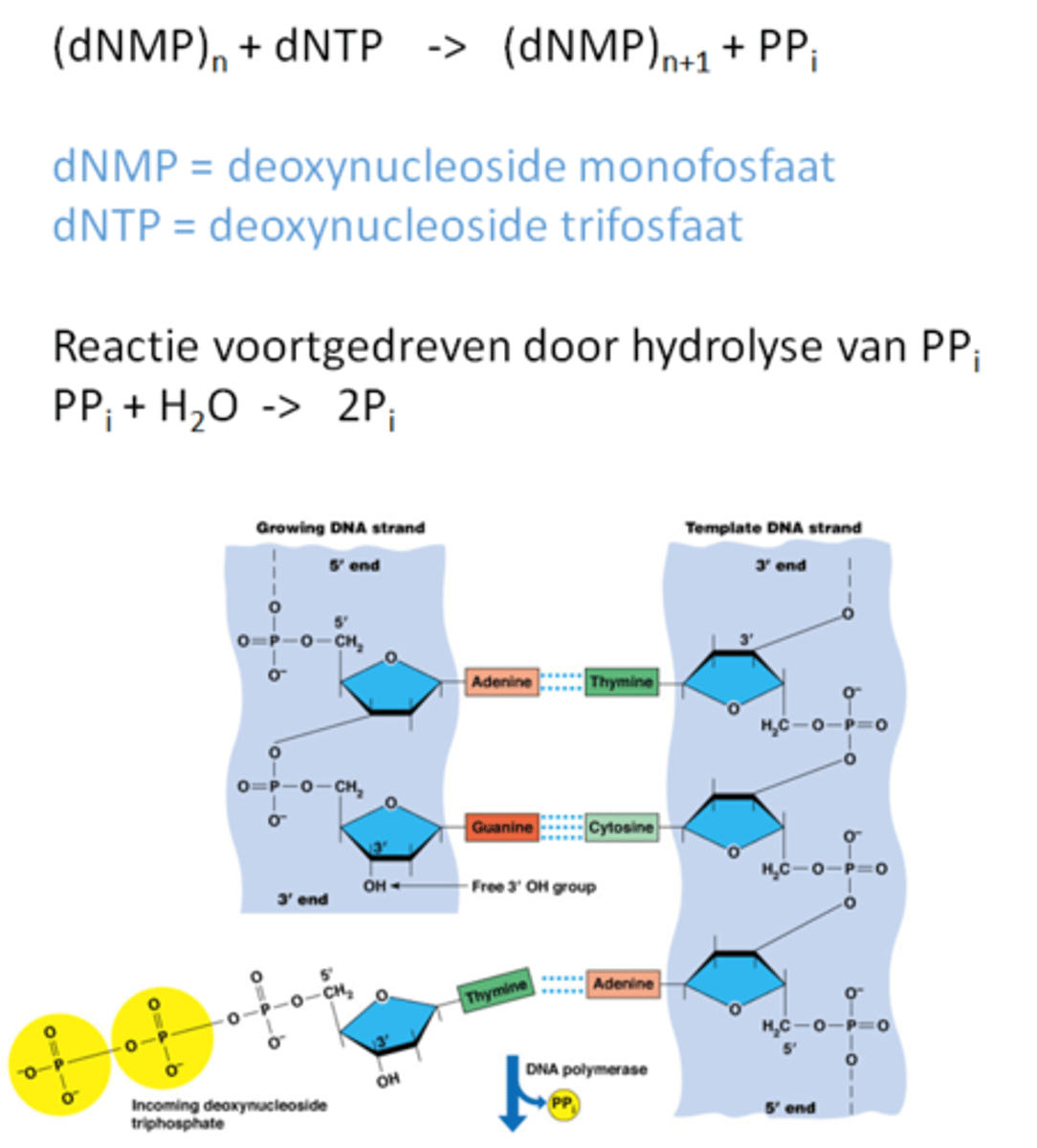
In welke richting verloopt de DNA replicatie?
synthese gebeurt altijd van 5' naar 3'
(op de 3' komt er een nieuw nucleotiden opgeplakt!)
DNA polymerase leest de streng van 3' naar 5'
foto: hier gebeurt de DNA replicatie van rechts naar links
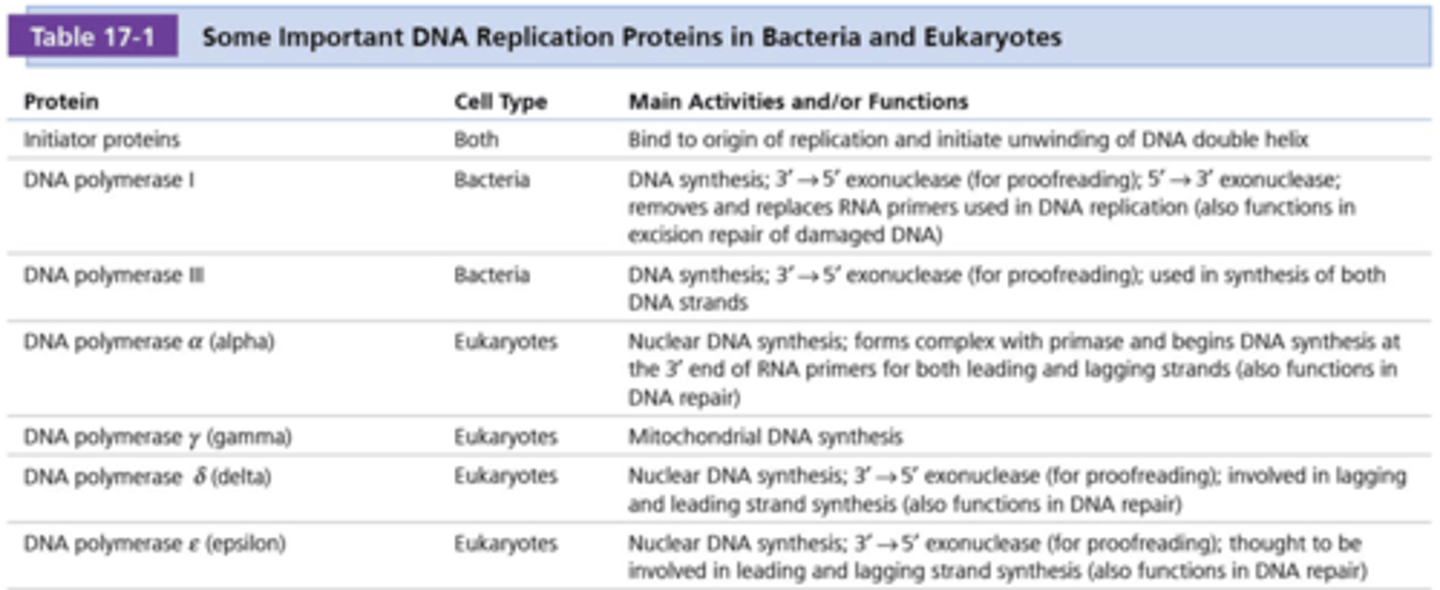
Welke vormen van DNA polymerases bestaan er? kennen! vertaling via chatgpt
let op voor de puntkomma's, als er staat DNA Synthese; 3'-5' exonuclease activiteit; betekent dat dat de exonuclease activiteit 3'-5' is en niet de DNA synthese
Eiwit: Initiatoreiwitten
Celtype: Beide
Belangrijkste Activiteiten en/of Functies: Binden aan oorsprong van replicatie en initiëren het ontwinden van de DNA-dubbelhelix.
Eiwit: DNA-polymerase I
Celtype: Bacteriën
Belangrijkste Activiteiten en/of Functies: DNA-synthese; 3' → 5' exonuclease (voor proofreading); 5' → 3' exonuclease; verwijdert en vervangt RNA-primers die gebruikt worden in DNA-replicatie (functioneert ook in excisieherstel van beschadigd DNA).
Eiwit: DNA-polymerase III
Celtype: Bacteriën
Belangrijkste Activiteiten en/of Functies: DNA-synthese; 3' → 5' exonuclease (voor proofreading); wordt gebruikt bij de synthese van beide DNA-strengen.
extra: DNA polymerase III is essentieel voor de replicatie in prokaryoten. Aangetoond met temperatuur-afhankelijke mutanten: functioneel bij 37°C maar niet bij 42°C
Eiwit: DNA-polymerase α
Celtype: Eukaryoten
Belangrijkste Activiteiten en/of Functies: Nucleaire DNA-synthese; vormt complex met primase en start DNA-synthese aan het 3'-einde van RNA-primers voor zowel de leading als de lagging strand (functioneert ook in DNA-herstel).
Eiwit: DNA-polymerase γ (gamma)
Celtype: Eukaryoten
Belangrijkste Activiteiten en/of Functies: Mitochondriale DNA-synthese.
Eiwit: DNA-polymerase δ (delta)
Celtype: Eukaryoten
Belangrijkste Activiteiten en/of Functies: Nucleaire DNA-synthese; 3' → 5' exonuclease (voor proofreading); betrokken bij de synthese van zowel de leading als de lagging strand (functioneert ook in DNA-herstel).
Eiwit: DNA-polymerase ε (epsilon)
Celtype: Eukaryoten
Belangrijkste Activiteiten en/of Functies: Nucleaire DNA-synthese; 3' → 5' exonuclease (voor proofreading); vermoedelijk betrokken bij de synthese van zowel de leading als de lagging strand (functioneert ook in DNA-herstel).
let op: In bacteriën vertoont alleen DNA-polymerase I 5' -> 3' exonuclease-activ
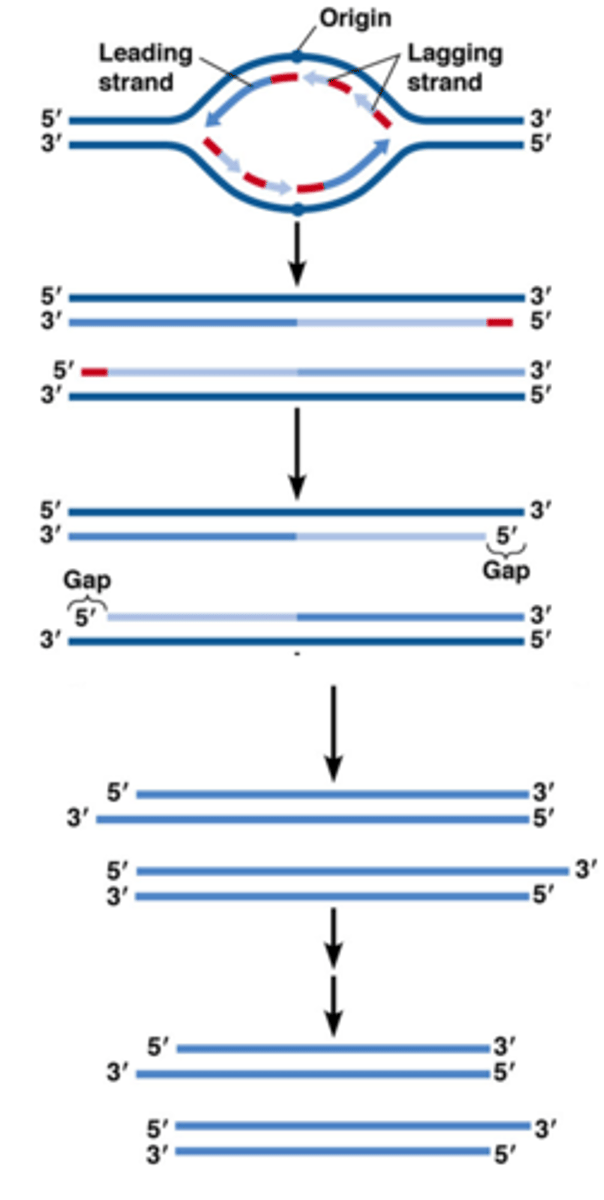
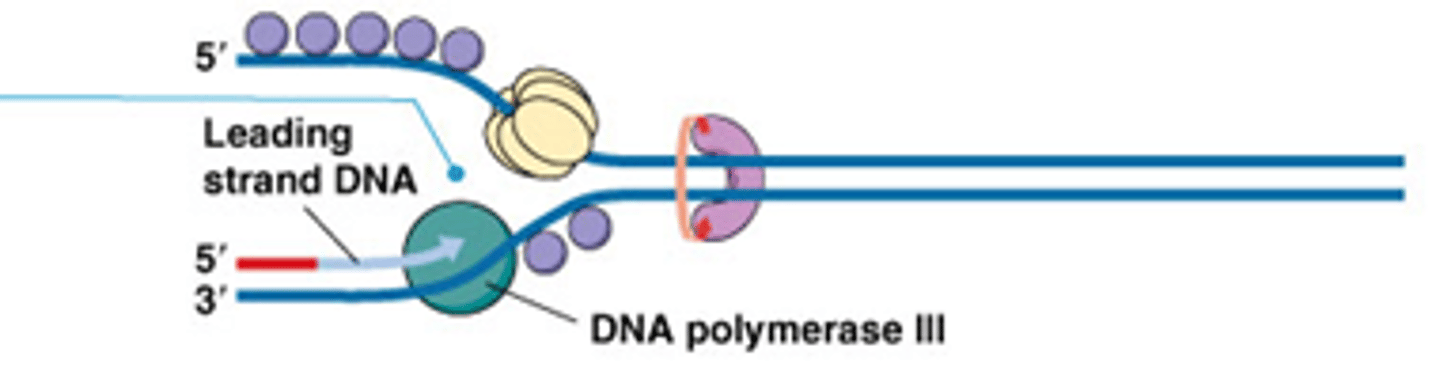
Wat is de snelle en wat is de trage streng, waarom?
continu op de leading strand= 3' -> 5' streng
discontinu op de lagging strand= 5' -> 3' streng
->omdat DNA polymerase altijd van van 5' -> 3' werkt
->beide strengen worden wel degelijk gerepliceerd
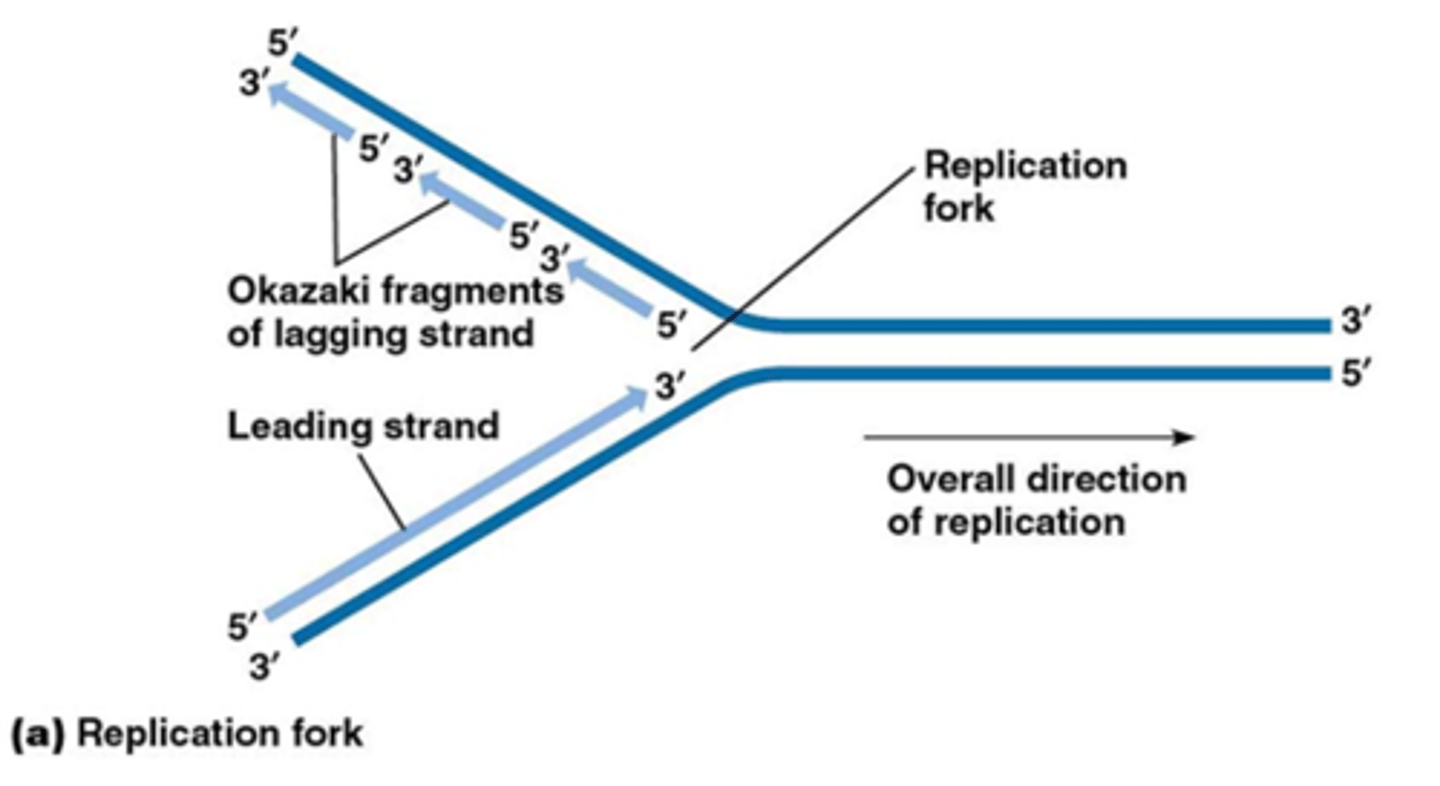
Hoe verloopt de DNA replicatie
DNA splitst nadat het pre-replicatiecomplex geactiveerd wordt door het enzym DNA-helicase tussen A=T (dubbele binding)
Er wordt een primer, een kort stukje RNA, gebonden op de enkelvoudige DNA-streng. Het enzym DNA-polymerase verlengt deze primer bidirectioneel door complementaire nucleotiden toe te voegen aan de openstaande basenparen. DNA-polymerase werkt heel snel in de 5'-3'-richting.
Aan de andere kant moet DNA-polymerase constant een sprongetje maken en moet er telkens een nieuwe primer gebonden worden. Een primer met een stukje DNA-streng noemt men een Okazakifragment.
DNA-ligase zorgt ervoor dat de Okazakifragmenten aaneengehecht worden.
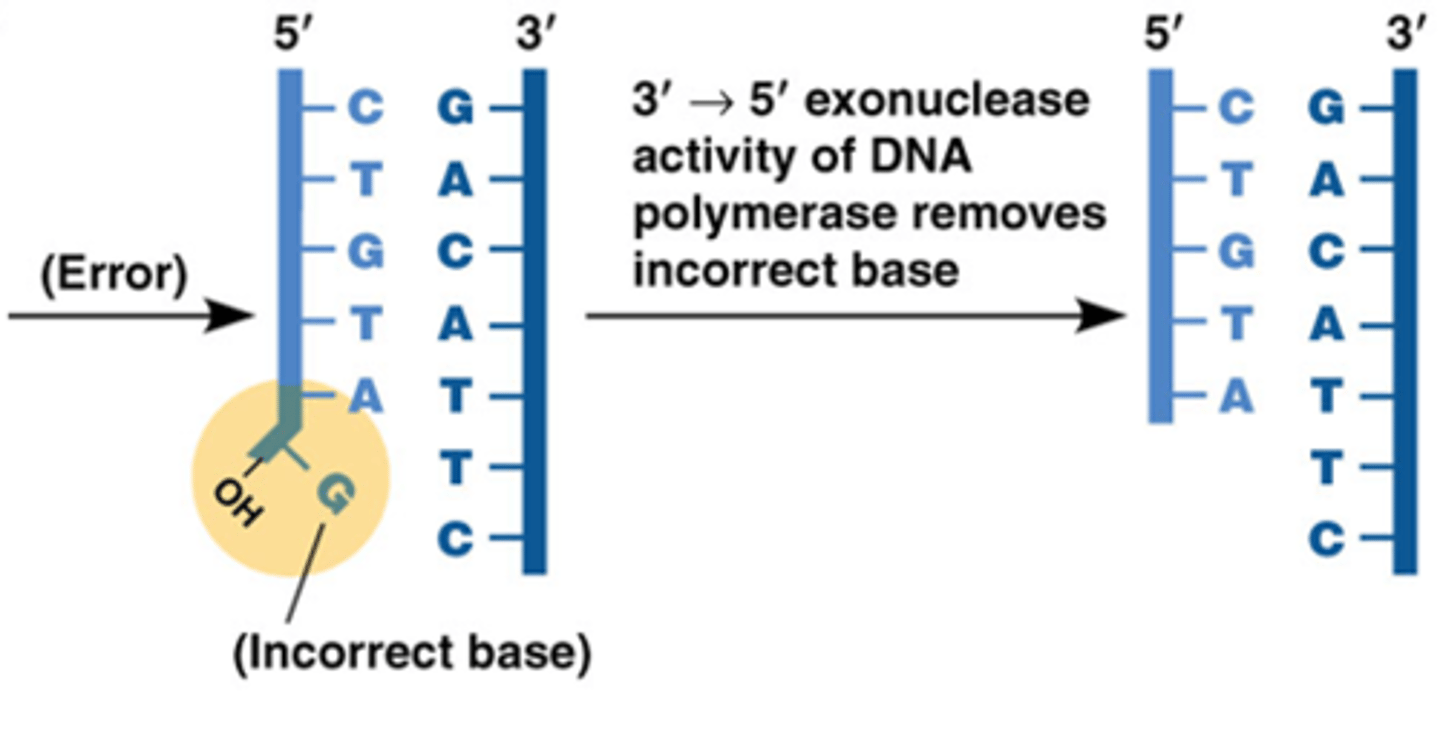
Wat is de foutenmarge van DNA polymerase en hoe vermijdt het lichaam fouten?
1fout /100.000
-wordt verder gereduceerd tot ca. 1 fout /10.000.000 door proeflezing
proeflezing: 3' → 5' exonuclease werking van DNA polymerase: het verwijdert foute basen door de fosfodiester binding te breken
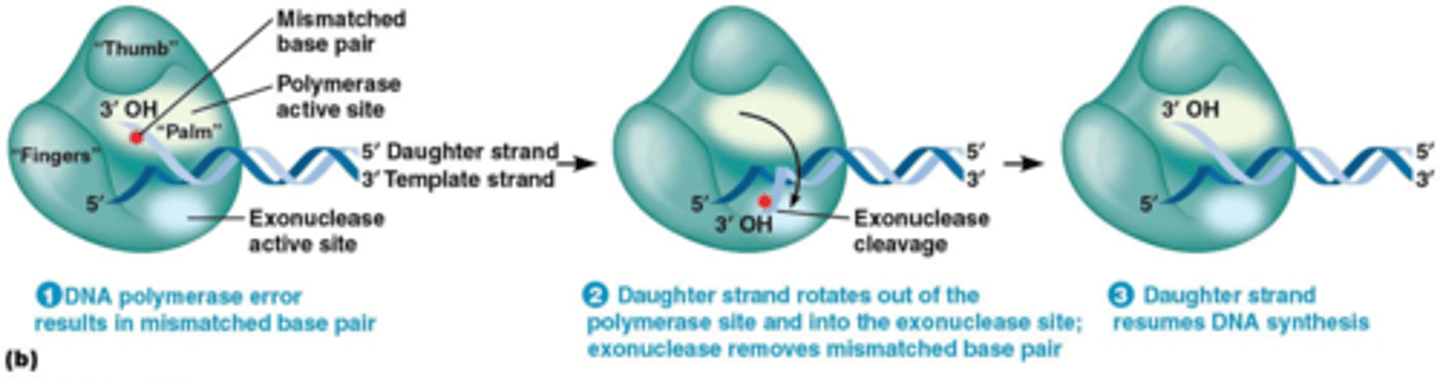
Hoe kan het dat DNA polymerase zowel een synthese activiteit als exonuclease activiteit kan hebben?
DNA polymerase heeft 2 katalytische sites in het complex:
Er is een synthese site en een exonuclease site die op 2 verschillende plekken van het eiwitcomplex liggen
het draait het foutje naar de grijze zone, daar wordt de fosfodiester binding gebroken
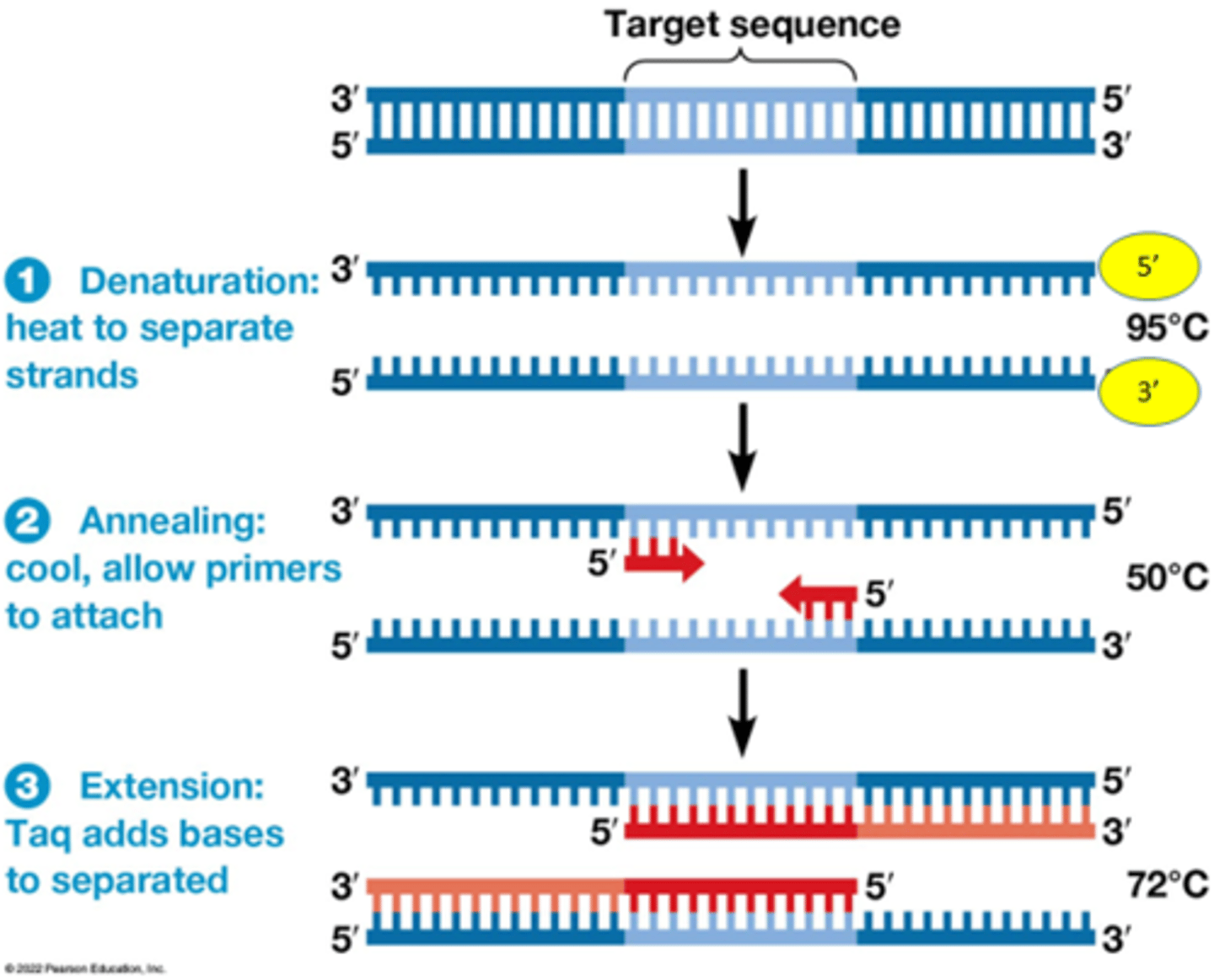
Hoe werkt PCR?
•Laat toe snel veel identieke kopieën van een DNA molecule te maken
•Afwisselend hoge (denatureren DNA) en lagere temperaturen (hybridisatie + DNA synthese).
3 stappen worden telkens herhaald:
-denaturation
-annealing
-extension
•Hittestabiel DNA polymerase: Taq-polymerase
•Synthetische primers op basis van complementariteit
Handboek p646
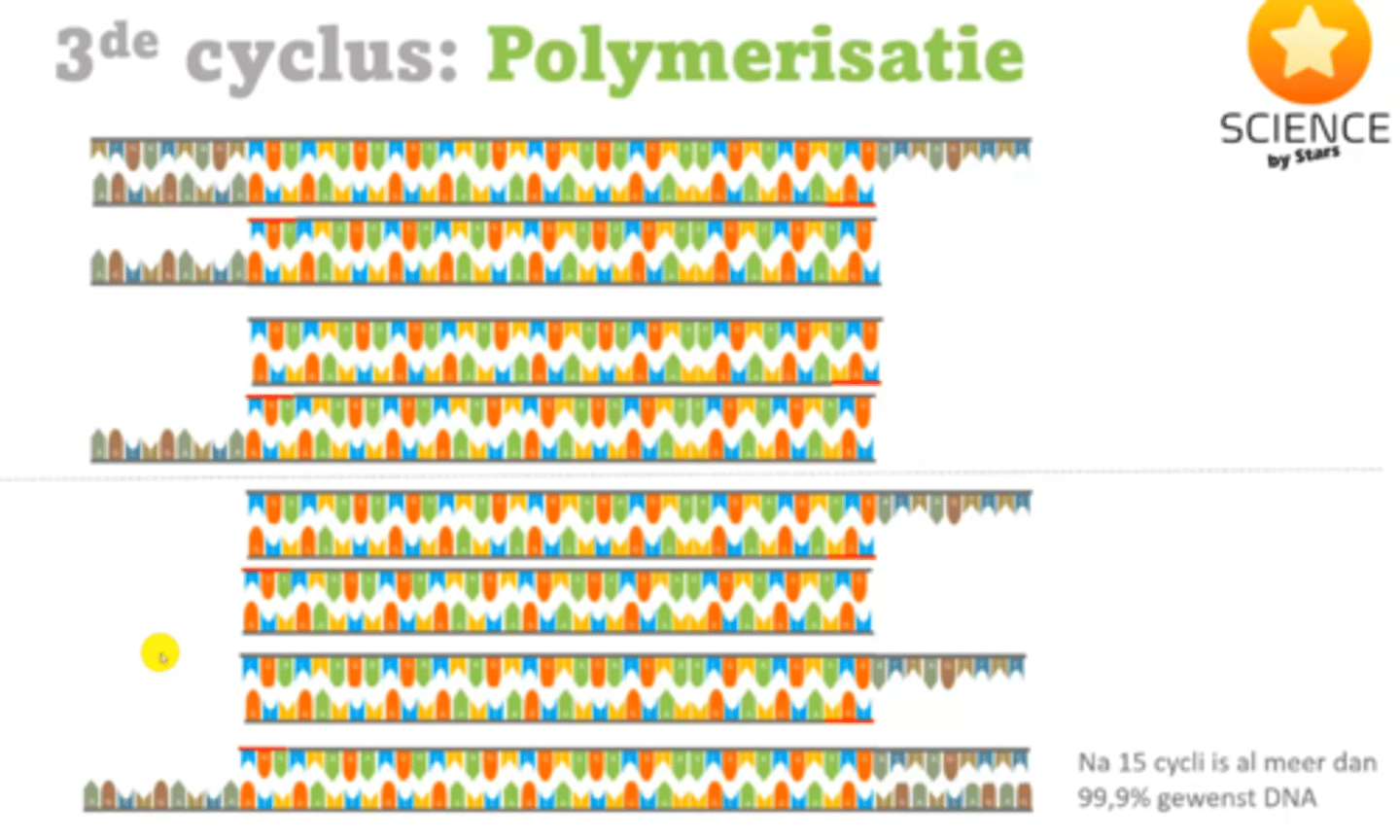
Wat krijg je als je PCR blijft herhalen?
je krijgt exponentieel meer DNA van de gewenste lengte
Wat is de rol van primase in de DNA replicatie
RNA polymerase dat RNA primers synthetiseert (van ca. 10 nucleotide) met DNA als matrijs in 5' naar 3' richting. De RNA synthese is de novo.
Primase + helicase vormen primosoom in bacteria.
NTP's als substraat en geen dNTP's als substraat
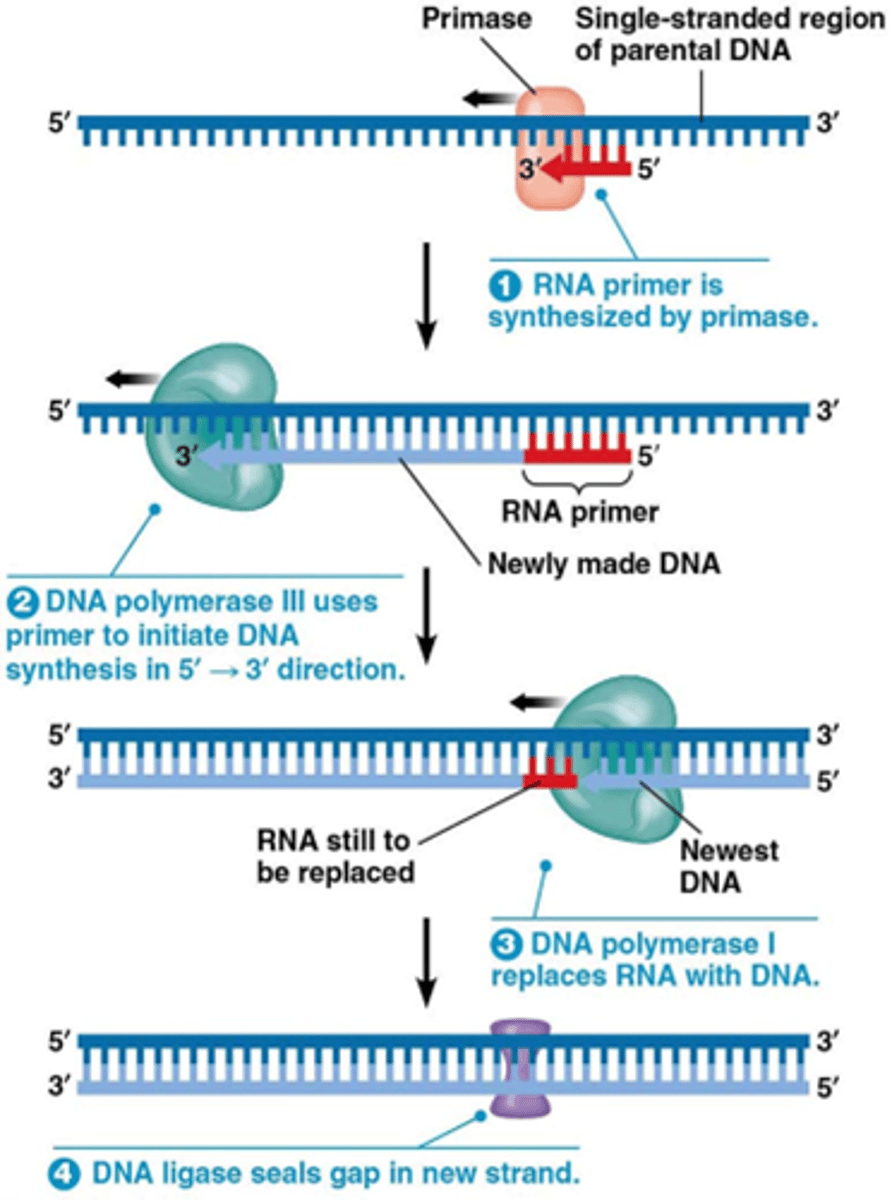
Wat is de rol van DNA polymerase III in de DNA synthese
DNA polymerase III verlengt de RNA primer aan de 3’ uiteinde met DNA (DNA synthese 5’->3’)
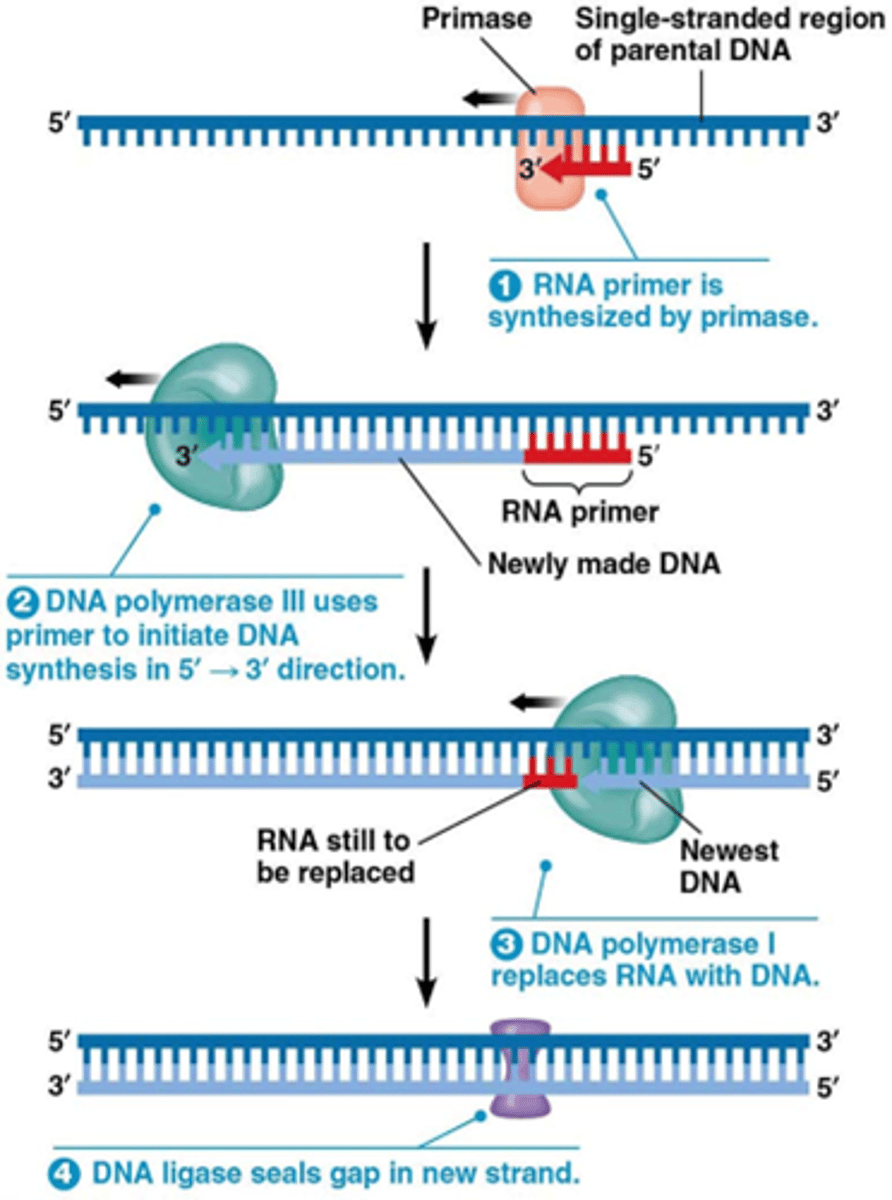
Wat is de rol van DNA polymerase I in de DNA synthese
DNA polymerase I
- verwijdert de RNA primers via 5'->3' exonuclease activiteit
- voert proofreading uit via 3'->5' exonuclease activiteit
- vult de gaten via 5'->3' DNA synthese
dit is anders bij eukaryoten! wij verwijderen RNA primers door endonuclease RNAse H (specifiek voor DNA-RNA duplex) en exonuclease FEN1
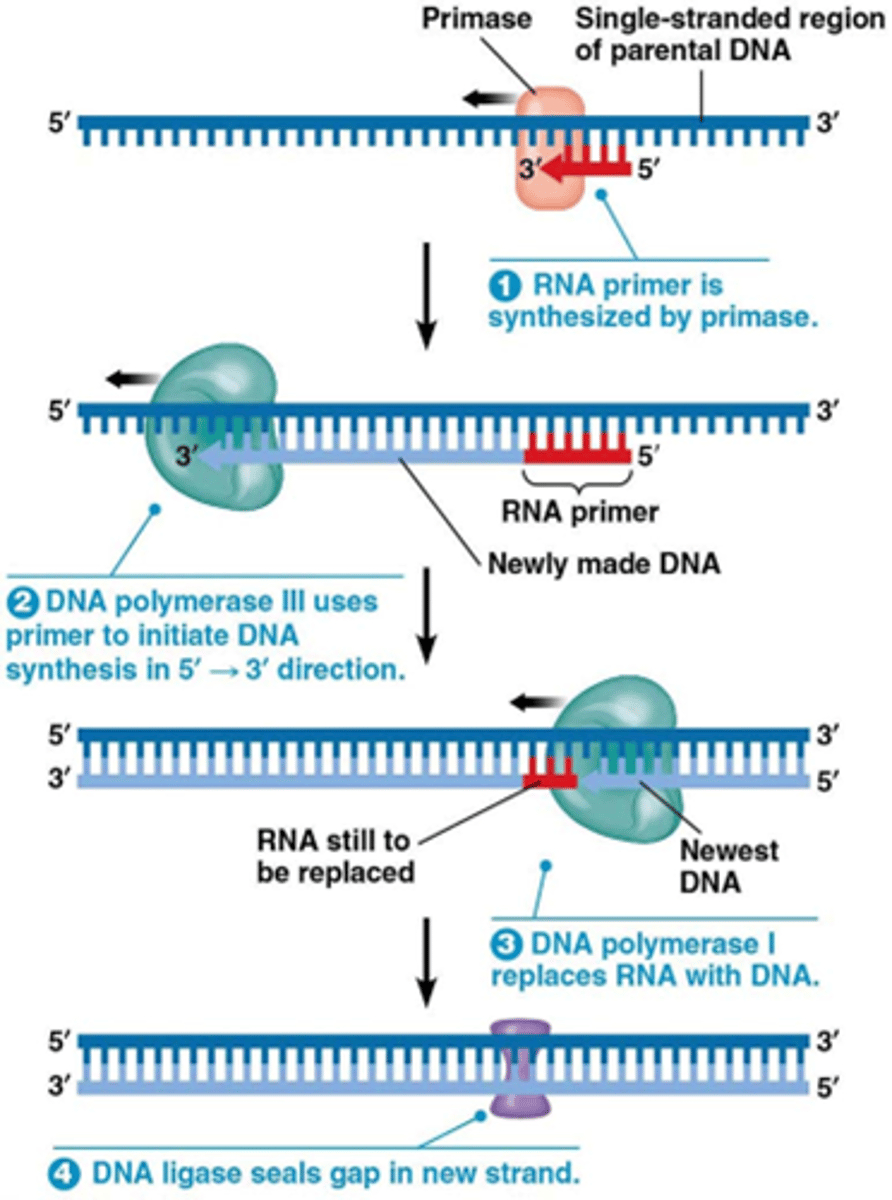
Wat is de rol van DNA ligase in de DNA synthese
DNA ligase verbindt de 5' en 3' uiteinden (katalyseert de vorming van een fosfodi-esterbinding, verbruik van ATP
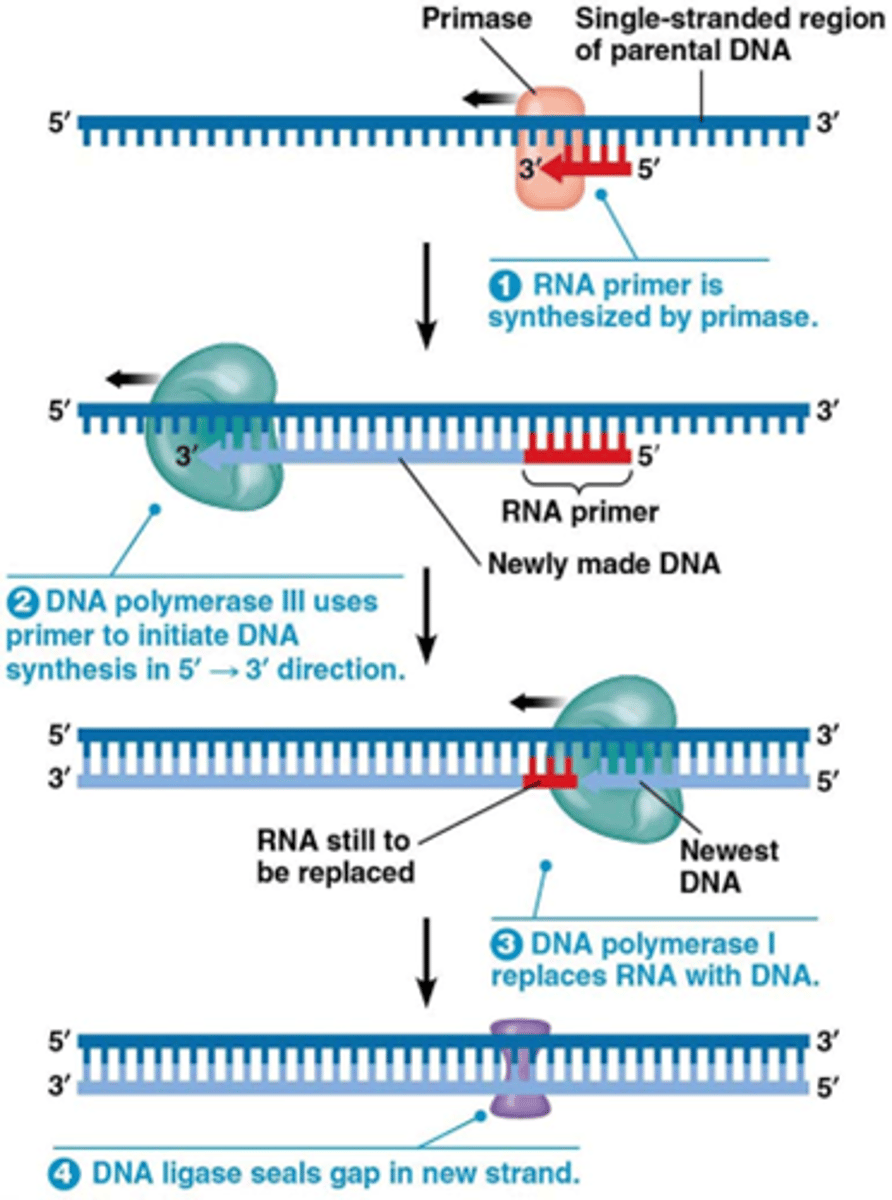
Eenmaal de DNA replicatie gestart is, hoe gaat de dubbele helix dan gebroken worden? (2 enzymes spelen een belangrijke rol)
DNA-helicasen, een enzyme dat de waterstofbruggen zal breken
-> er komen direct SSB's die zullen binden op het ssDNA
Ook belangrijke werking van topoisomerases zodat de supercoiling (en de tortionele spanning die ontstaat) verwijderd wordt
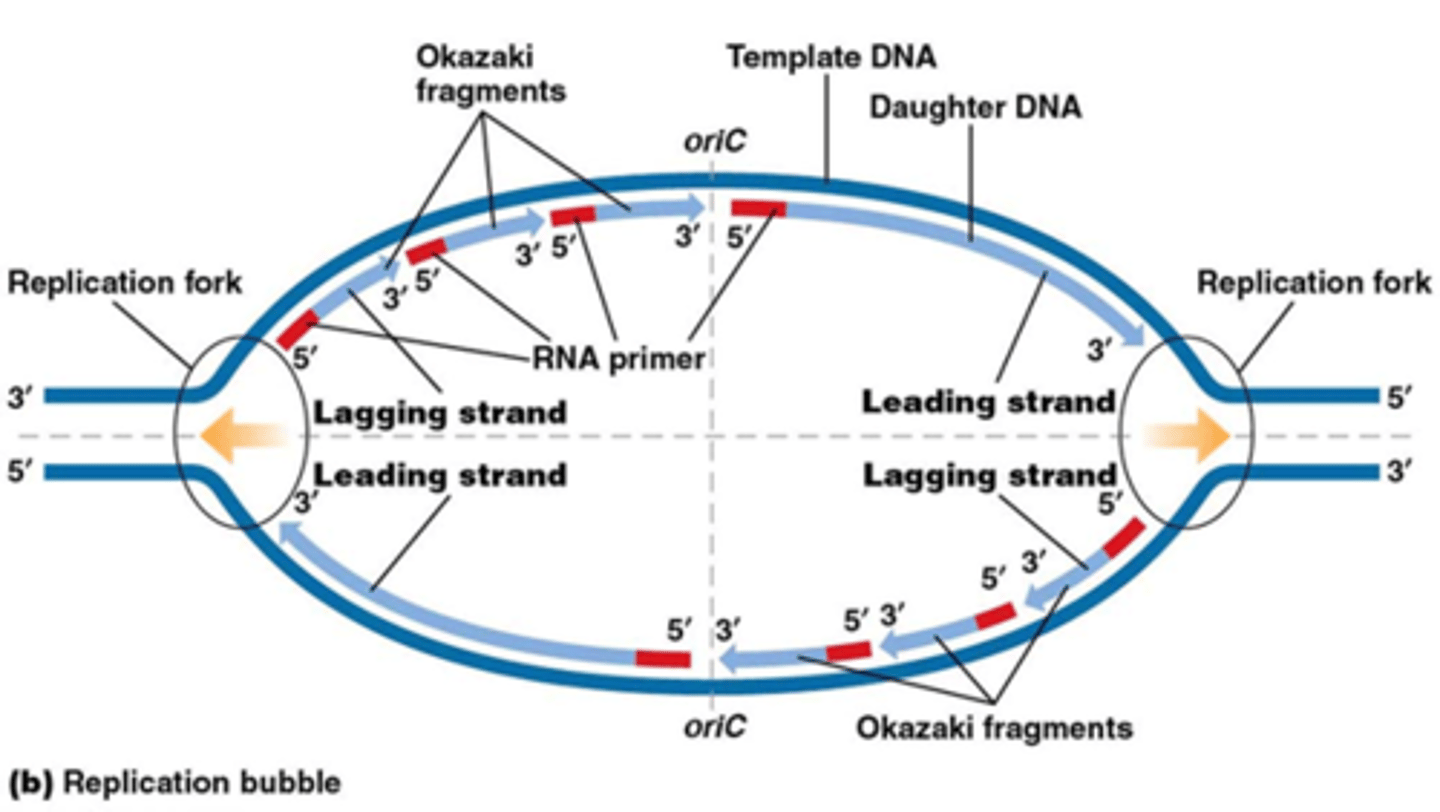
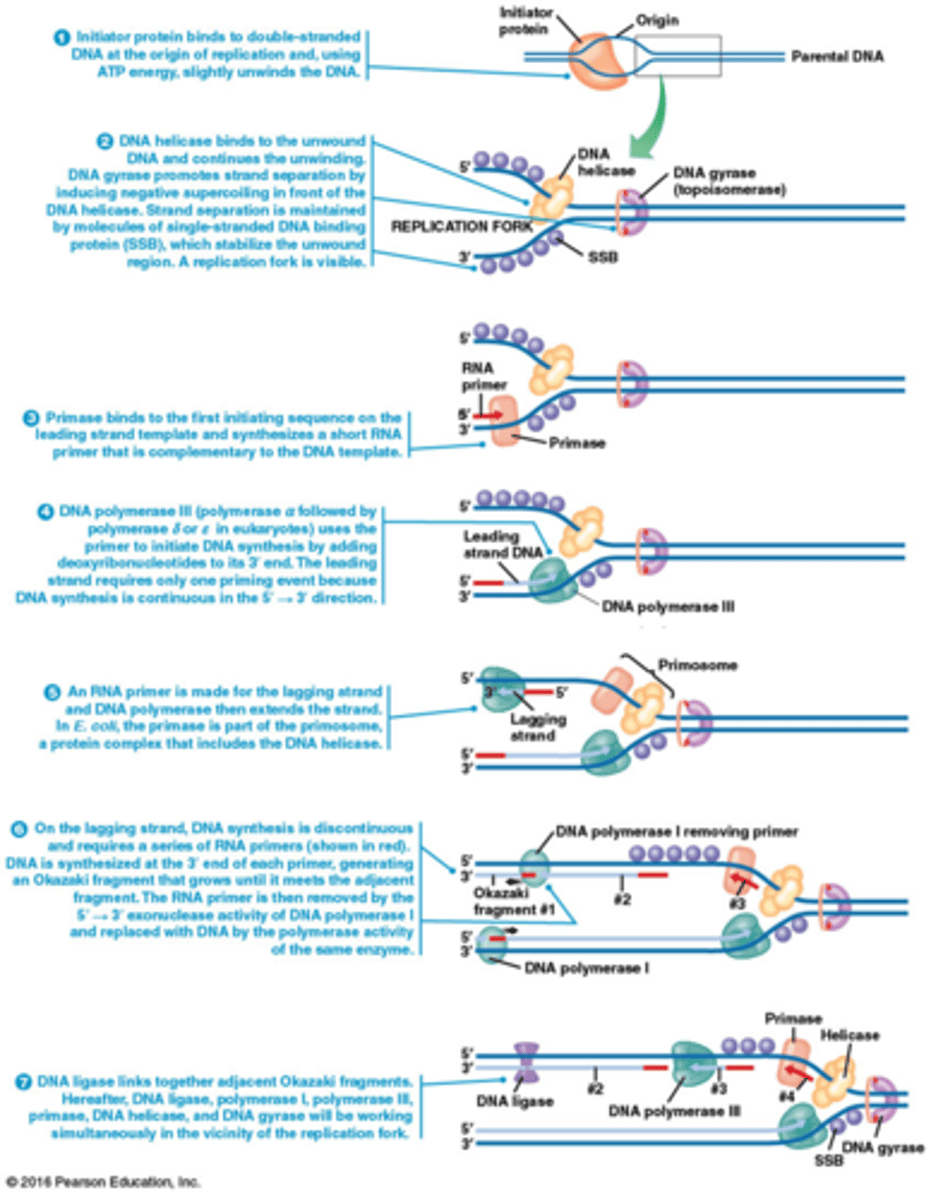
Hoe verloopt DNA replicatie in bacteria? samenvatting van de ovrige slides, geen nieuwe info eigenlijk
1 Initiatoreiwit bindt aan dubbelstrengs DNA bij de ORI en gebruikt ATP-energie om het DNA lichtjes te ontwinden.
2 DNA-helicase bindt aan het ontwonden DNA en gaat verder met het ontwinden. DNA-topoisomerasen helpen bij de scheiding door negatieve supercoiling voor helicase te creëren. Enkelstrengs DNA-bindende eiwitten (SSB) stabiliseren het ssDNA. Een replicatievork wordt zichtbaar.
3 Primase bindt aan de eerste initiërende sequentie op de leidende streng en synthetiseert een korte RNA-primer die complementair is aan het DNA.
4 DNA-polymerase III (of δ en ε in eukaryoten) begint met DNA-synthese door nucleotiden toe te voegen aan het 3'-uiteinde van de primer op de leidende streng. DNA-synthese is continu in de 5' → 3' richting.
5 Een RNA-primer wordt gemaakt voor de volgende streng, en DNA-polymerase verlengt deze streng. In E. coli is de primase deel van het primosoom, een eiwitcomplex dat helicase omvat.
6 Op de volgende streng is DNA-synthese discontinu en vereist het meerdere RNA-primers. Aan het 3'-uiteinde van elke primer wordt een Okazaki-fragment gesynthetiseerd. DNA-polymerase I verwijdert de RNA-primers en vervangt ze door DNA.
7 DNA-ligase verbindt aangrenzende Okazaki-fragmenten. Daarna werken DNA-ligase, polymerase I, polymerase III, primase, helicase en DNA-topoisomerasen samen bij de replicatievork.
https://mediaplayer.pearsoncmg.com/assets/secs-molecular-model-of-dna-replication
Hoe zorgt het lichaam ervoor dat de leading en lagging strand gelijktijdig repliceren?
Door interactie van eiwitten op beide strengen
Alle eiwitten die een rol spelen in DNA replicatie vormen het replisoom. Laat coördinatie toe tussen leidende en navolgende streng.
Klemprotëine (paars) nodig voor DNA polymerase op de streng te zetten!
klemproteine is geassocieerd met een clamp loader (een groter eiwit complex)
Clamp loader zorgt ervoor dat er een lus gevormd wordt
->DNA replicatie gebeurt gelijktijdig op de leading en op de lagging strand
https://www.youtube.com/watch?v=QMX7IpME7X8
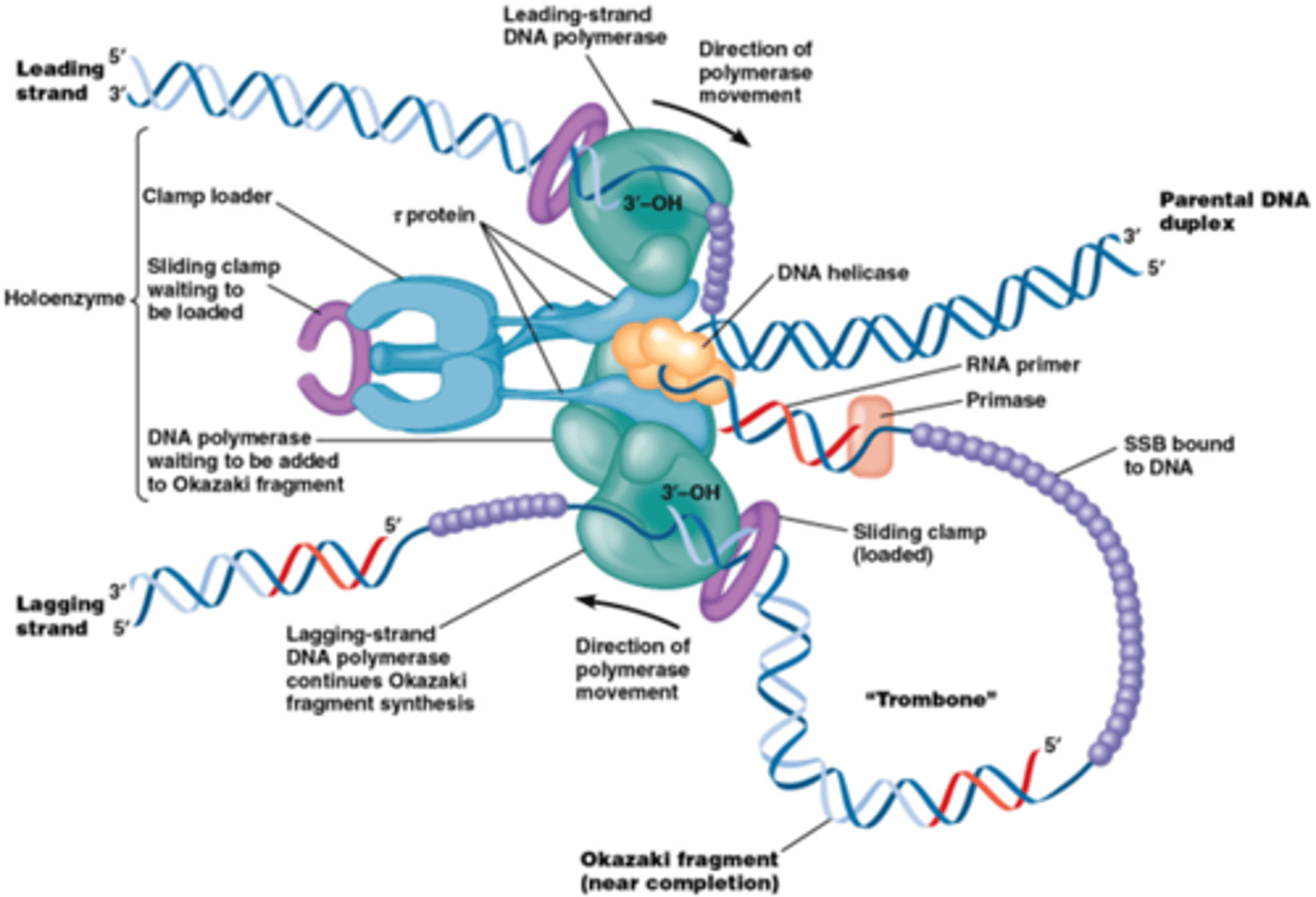
Wat zijn de verschillen in de DNA replicatie tussen prokaryoten en eukaryoten
•Gelijkaardig aan replicatie in prokaryoten
•Klemproteïne bij eukaryoten proliferating nuclear cell antigen, PCNA: recruteert DNA polymerase d
•Verschilpunt tov prokaryoot: verwijderen van RNA primers door endonuclease RNAse H (specifiek voor DNA-RNA duplex) en exonuclease FEN1
•Replicatie-fabrieken dicht bij kernmembraan
•Chromatine-remodelleer complexen: bv. laterale verschuiving van histonen complexen (repositionering van nucleosomen)
•Het probleem van de replicatie van chromosoomuiteinden. (zie foto en volgend kaartje!)
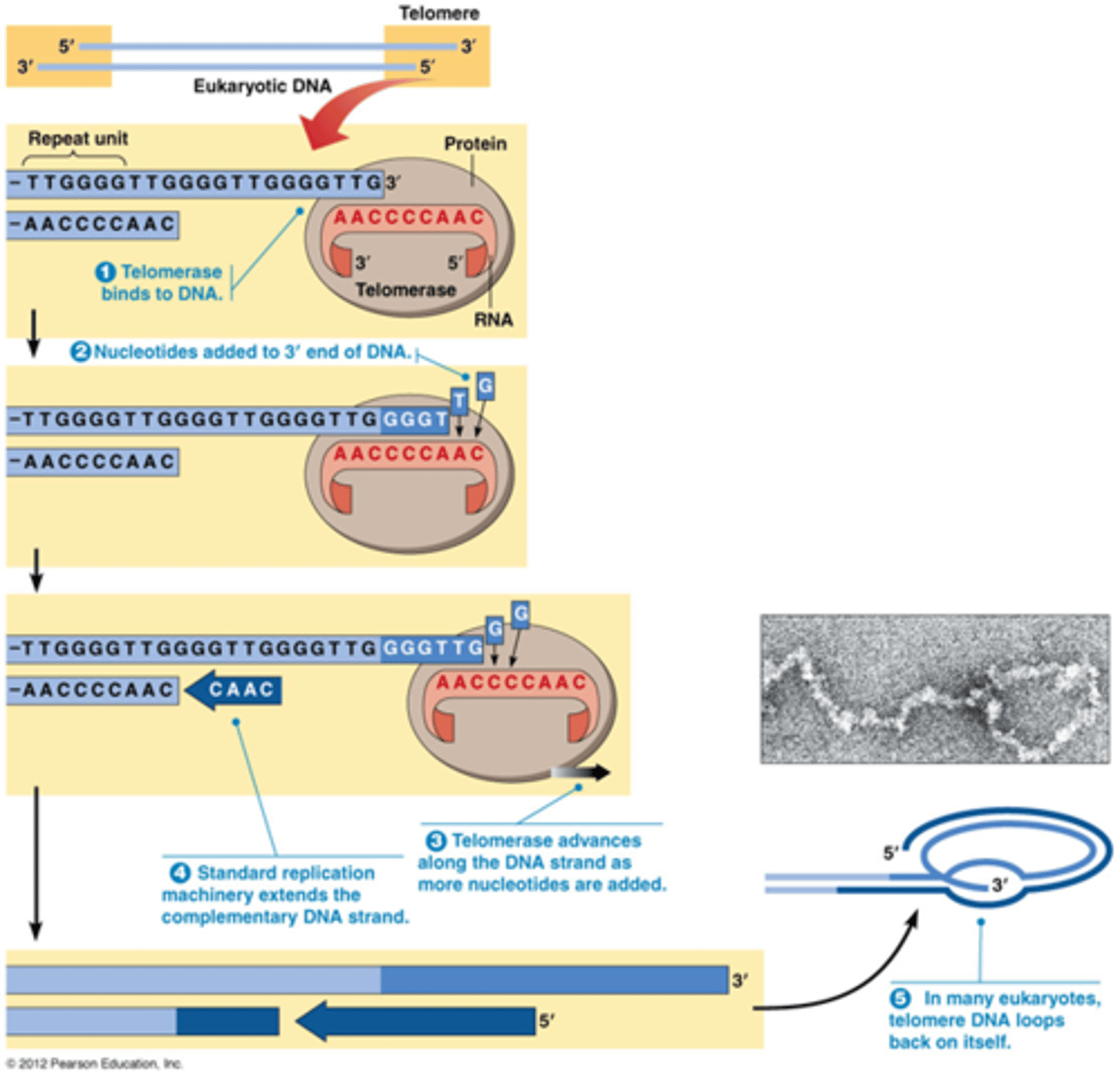
Het probleem van de replicatie van chromosoomuiteinden. (wat is het , wat is het effect, hoe lost het lichaam dit probleem op?)
-laatste RNA primer wordt verwijderd en kan niet opgevuld worden
->5' is altijd korter
->telomeren worden korter en korter doorheen u leven
(Telomeren binden eiwitten die chromosoomuiteinden beschermen tegen exonucleasen.)
->fusie van chromosomen
->DNA stopt met delen na 50-60 generaties (Hayflick limiet)
Gevolgen op lange termijn
-->Verminderde wondheling door verlies van bindweefselcellen.
--> Verhoogde gevoeligheid aan infecties door verlies van immuuncellen.
--> Zweren in darmkanaal door verlies van epitheelcellen.
oplossing: telomerase dat telomeren verlengt
Telomerase komt tot expressie in proliferende cellen (embryonale cellen, geslachtscellen, stamcellen en kankercellen). (genactivatie zie H20)
Telomerase: enzyme dat bestaat uit eiwit + RNA (= RNP).
RNA = matrijs voor verlenging telomeren.
Telomerase heeft reverse transcriptase activiteit (van RNA -> DNA synthetiseren)
3’ uiteinde van het telomeer is langer dan de complementair streng en vouwt terug op zichzelf
->Mutatie van telomerase is geassocieerd met versnelde veroudering.
-Kankercellen bevatten telomerase want die kunnen meer dan 50 keer delen
->Telomerase-inhibitoren voor kankertherapie?
17.2 DNA schade en foutenherstel
Wat is een mutatie?
Een verandering in het genotype als gevolg van een wijziging in het DNA.
De nieuwe vorm wordt een mutant genoemd
vaak zorgen mutaties niet voor veranderingen. Slechts 3% van het DNA codeert voor eiwitten, enkel als er net hier een mutatie optreedt zal het fenotype veranderen.
Hoe kunnen mutaties ontstaan?
volgende 6 kaartjes gaan in detail
Door endogene factoren
willekeurig omdat enzymen soms fouten maken
- DNA polymerase activiteit, fouten na proeflezing: ca. 1/10^7
- tautomeren van ringbasen -> verkeerde basenparing tijdens replicatie
- replicatie van een stukje DNA tweemaal (‘strand slippage’) in repetitief DNA
- spontane chemische modificaties aan basen zoals depurineringen, deamineringen
-spontane oxidatie van basen door ROS
door externe factoren
door milieufactoren of mutagene factoren. Bijvoorbeeld stralingen (UV- of X-stralingen) of chemische stoffen (ringbase-analogen, ringbase-modificerende agentia, intercalerende agentia)F
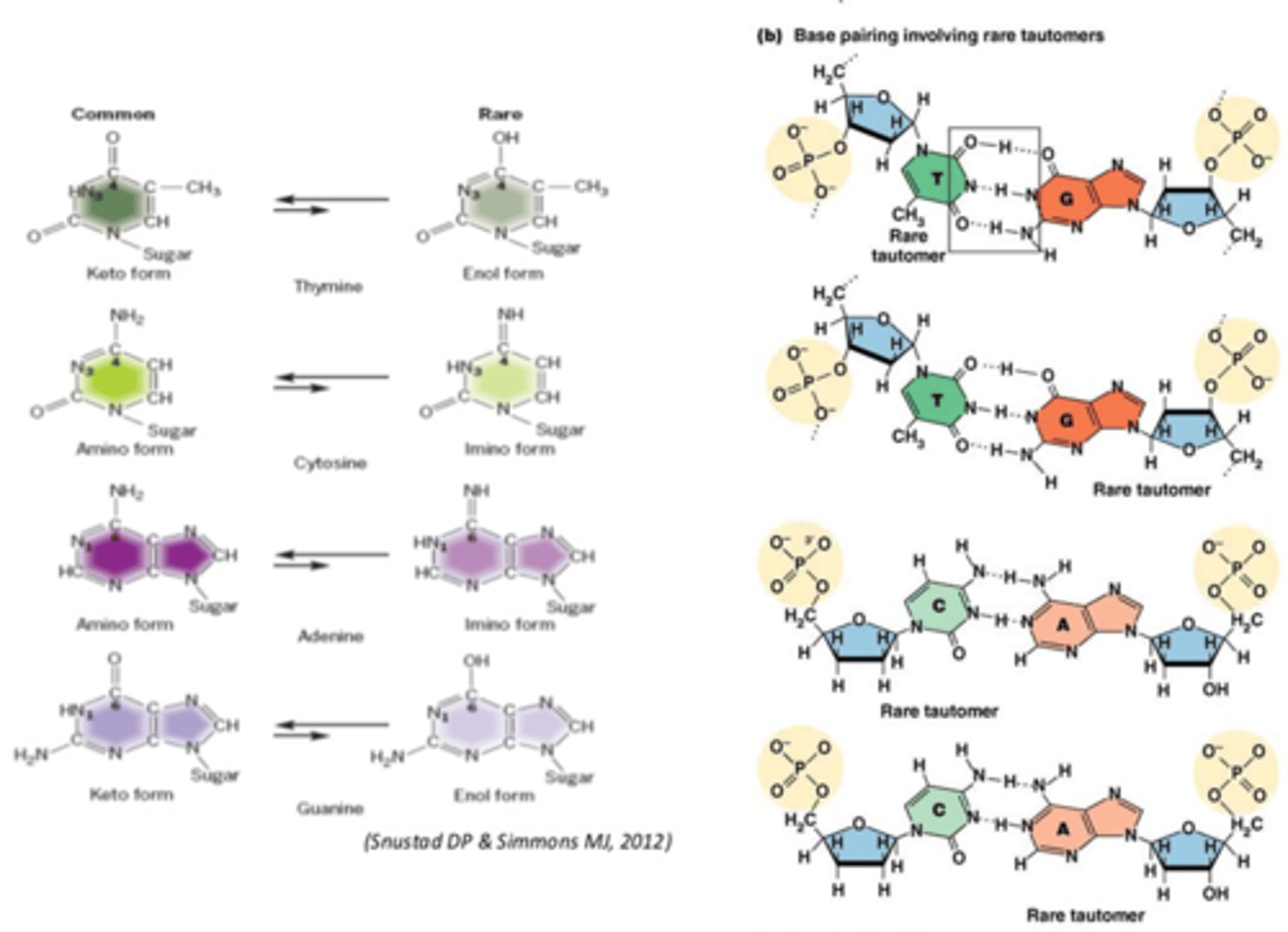
replicatie fouten door tautomeren van ringbasen
Tautomeer = zeldzame resonantiestructuur van ringbasen
->omkeerbare verplaatsing van H-atoom (O wordt OH)
•Tautomeren leidt tot DNA mismatch
•Geen Watson-Crick basenparing (zie rechts foto)
•Leidt tot mutatie tijdens replicatie
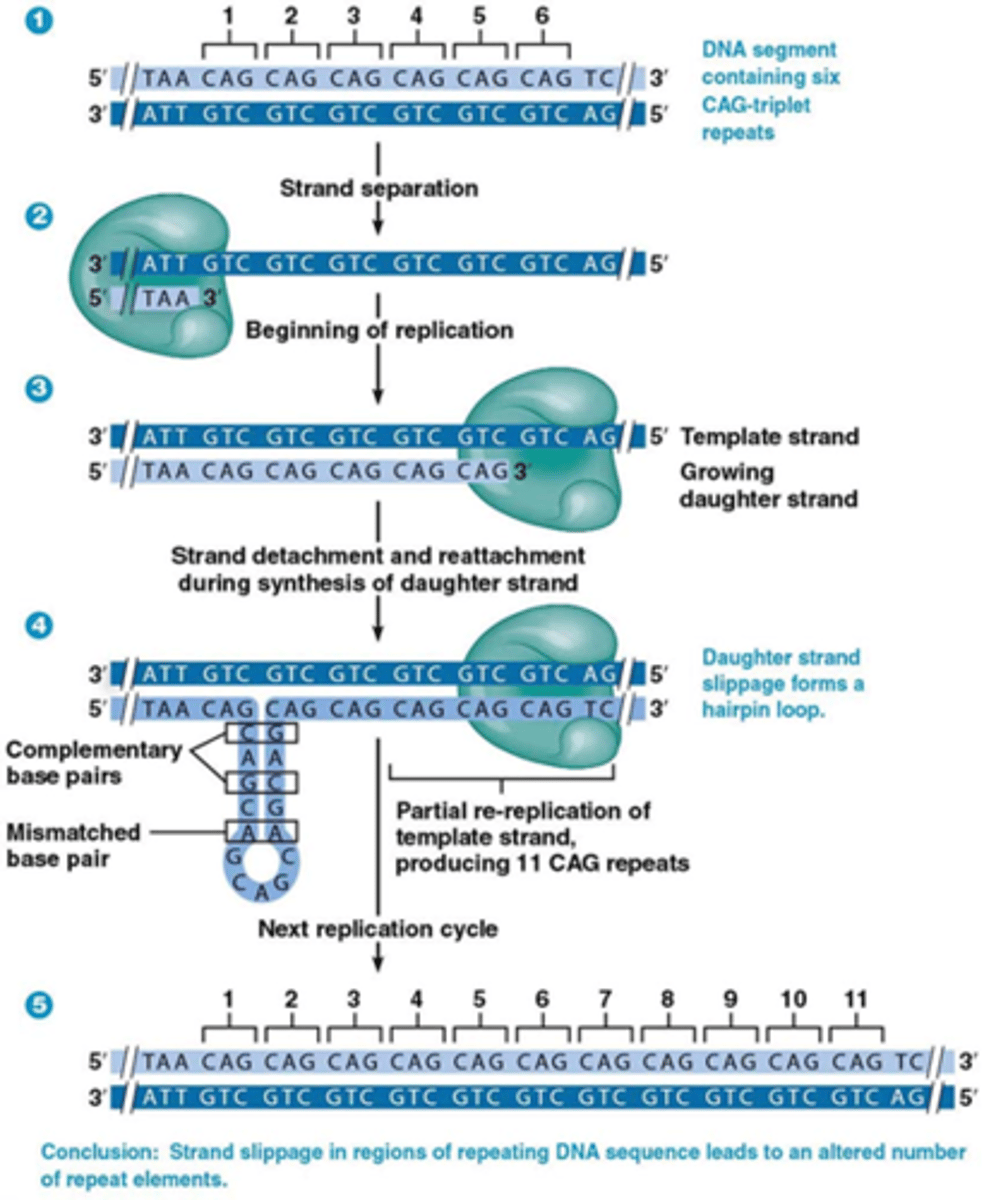
Replicatiefouten in repetitief DNA, bv. trinucleotiden-repeats
•CAG triplet codeert voor glutamine
•Dissociatie van DNA polymerase (‘strand slippage’): DNA polymerase gaat even af de template streng en er daarna terug op
•Vorming van ‘hairpin’ in nieuwgevormde streng, allemaal CAG's die met elkaar waterstofbruggen gaan vormen
•Overeenkomstige matrijs tweede keer gerepliceerd
->proteine heeft meer glutamines
->huntington's disease (autosomaal dominante* ziekte waardoor je dementie en parkinsonachtige symptomen hebt, iedereen sterft ongeveer 15 jaar na de eerste symptomen (rond 30-50 jaar oud) die iedereen met het gen krijgt)
*de mutatie doet zich dus voor in geslachtscellen!
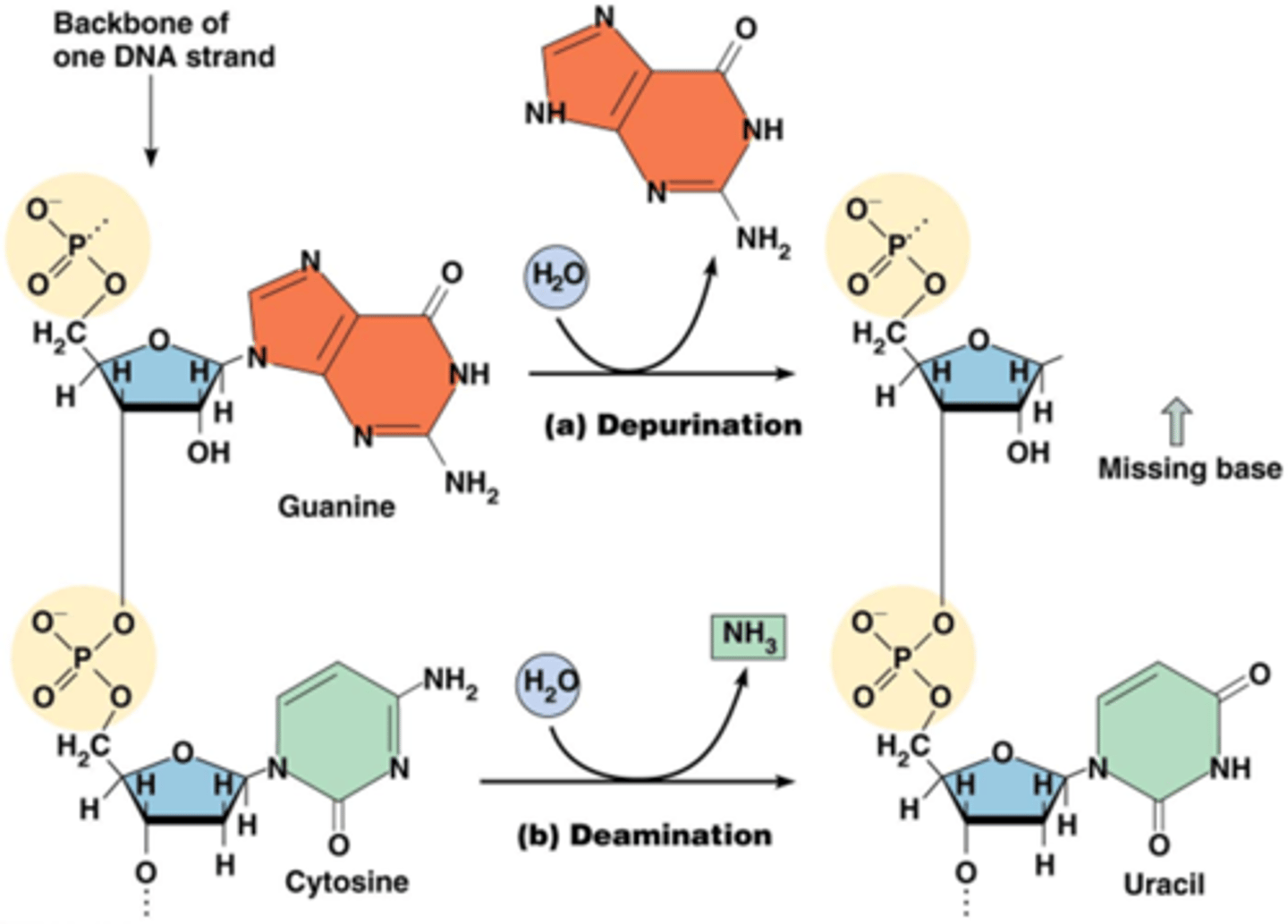
Replicatiefouten tgv spontane depurineringen en deamineringen
•Depurineringen: verlies van een purine base (adenine of guanine ringbasen), spontane hydrolyse van een glycoside binding, 1000den keer/cel/dag omdat de glycoside binding niet heel stabiel is
-> een base-vrij deoxyribose-fosfaat (=‘AP’ site)
•Tijdens de replicatie zal het DNA polymerase bij een ‘apurine’ meestal een ‘A’ toevoegen aan de dochterstreng
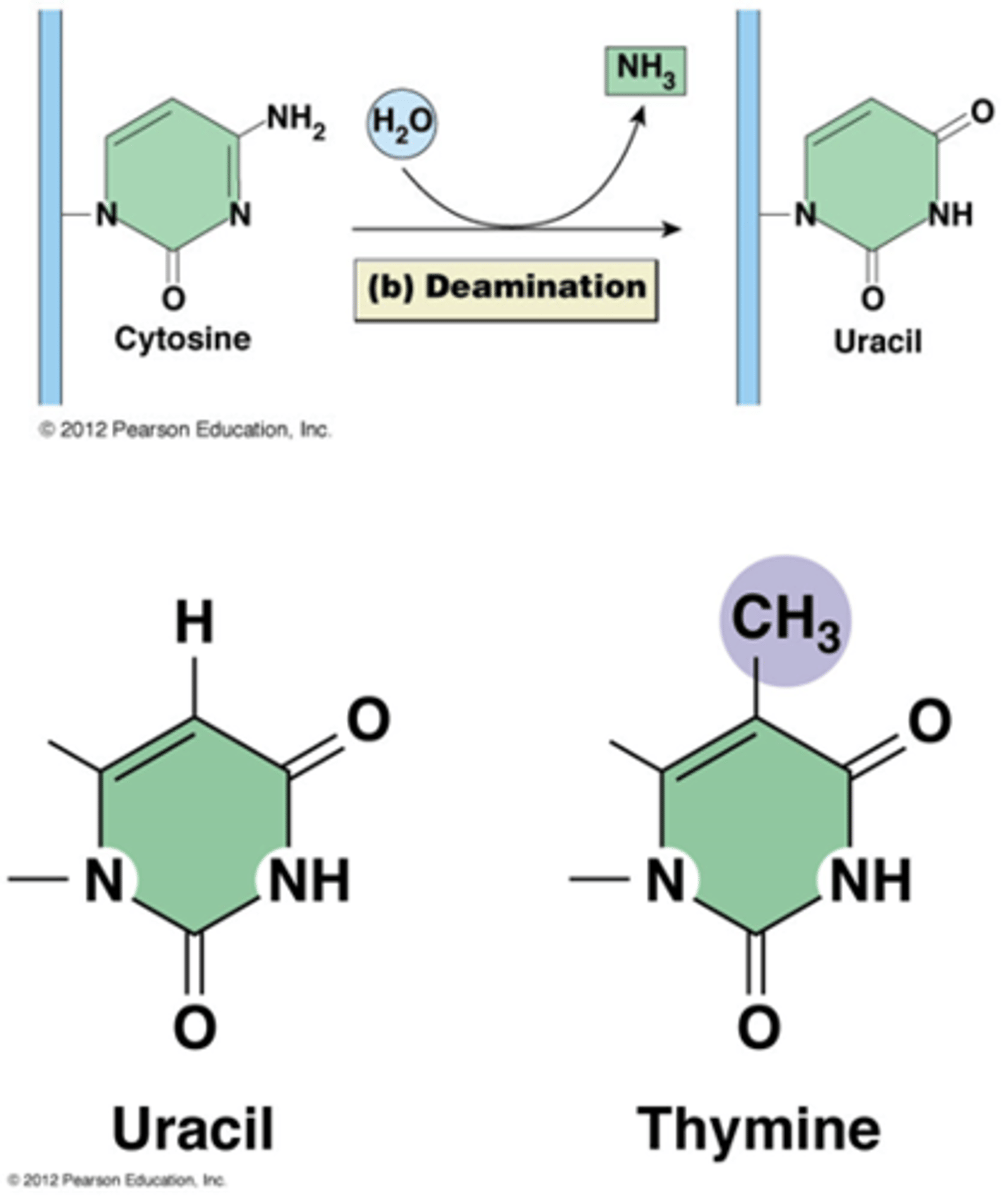
•Deamineringen: spontane hydrolyse van aminogroep, ca. 100/cel/dag (C->U->A )(cytosine is uracil met een aminegroep)
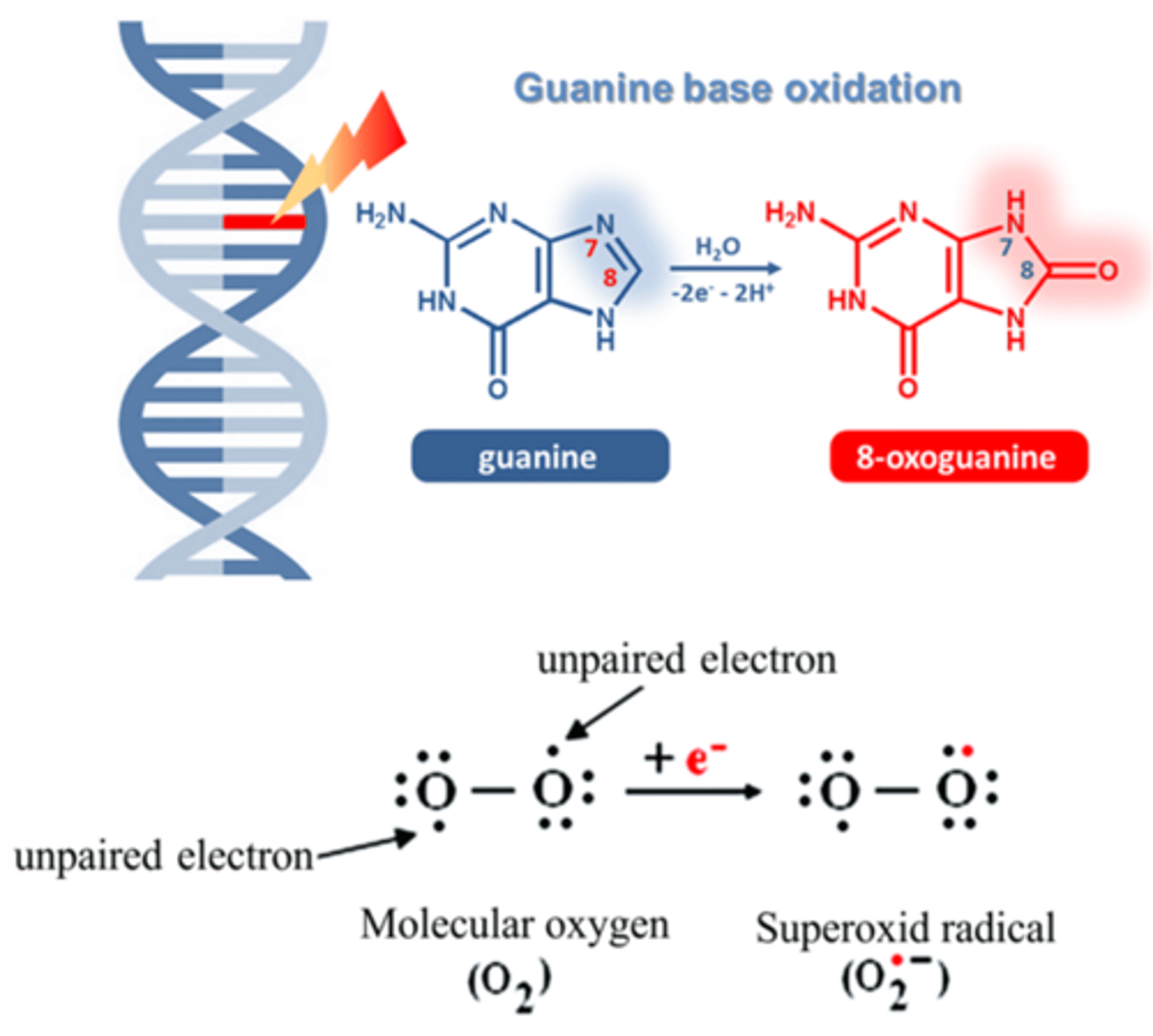
Replicatiefouten tgv spontane oxidatie van basen door ROS
-Reactieve zuurstofsoorten (ROS) leidt tot oxidatie van basen (guanine naar oxoguanine)
-ROS: zuurstofsoorten waarbij een elektronenpaar aanwezig is dat een elektron mist waardoor het heel reactief is bv. waterstofperoxide, superoxide anion
Welke chemische mutagenen induceren mutaties?
Ringbase-analogen Bv. 5’-bromo-deoxyuridine = een uracil-analoog dat ingecorporeerd gaat worden
->een tautomeer ontstaat
->verkeerde basenparing (mutatie)
Base-modificerende agentia
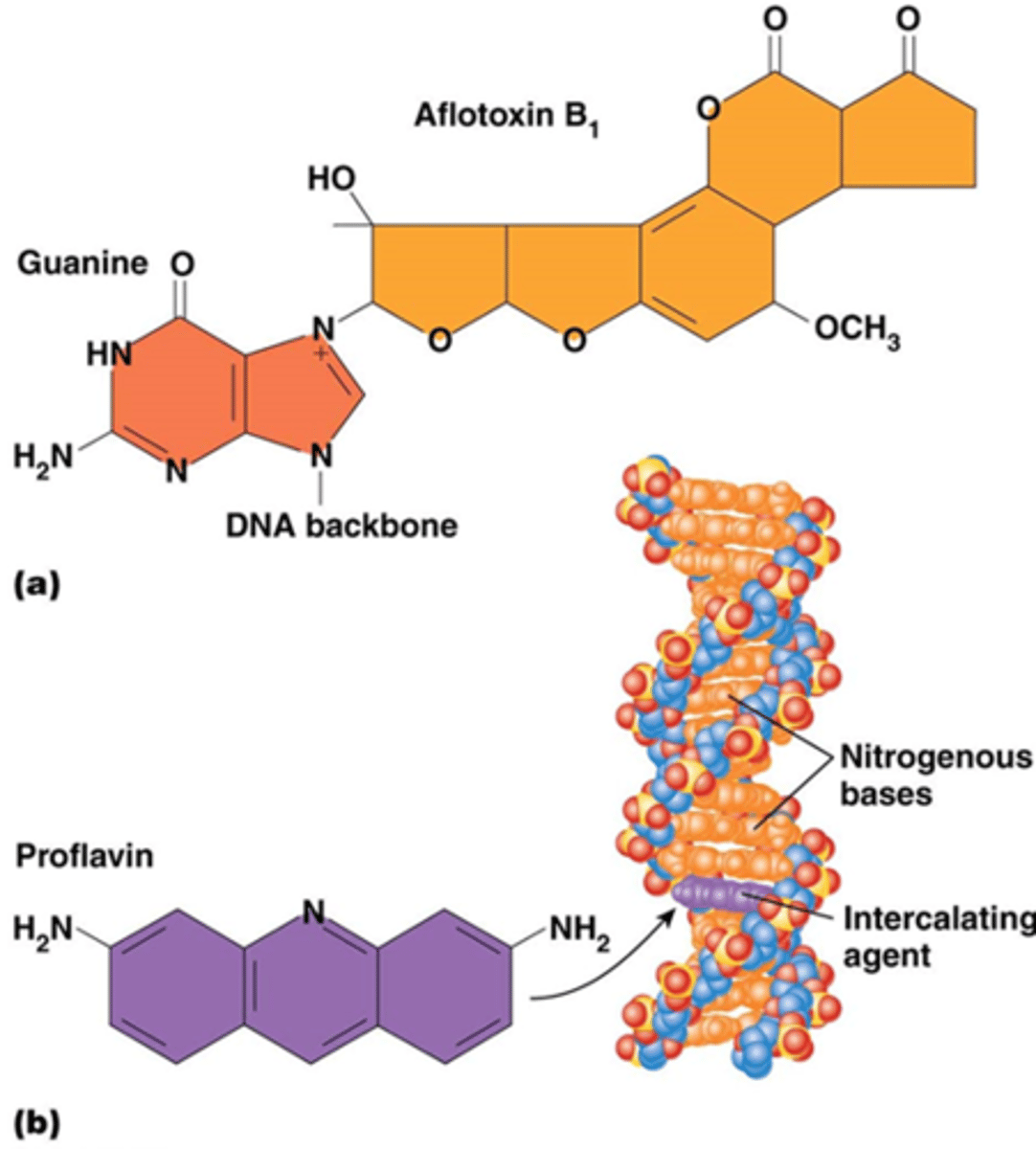
Bv. aflatoxine: leidt tot depurinering tgv verzwakking binding met deoxyribose. Sommige agentia blokkeren DNA polymerase. (zie foto)
Intercalerende agentia induceren deleties of invoegingen (inserties) in DNA (bv. ethidium bromide of proflavine)
Welke straling induceert mutaties
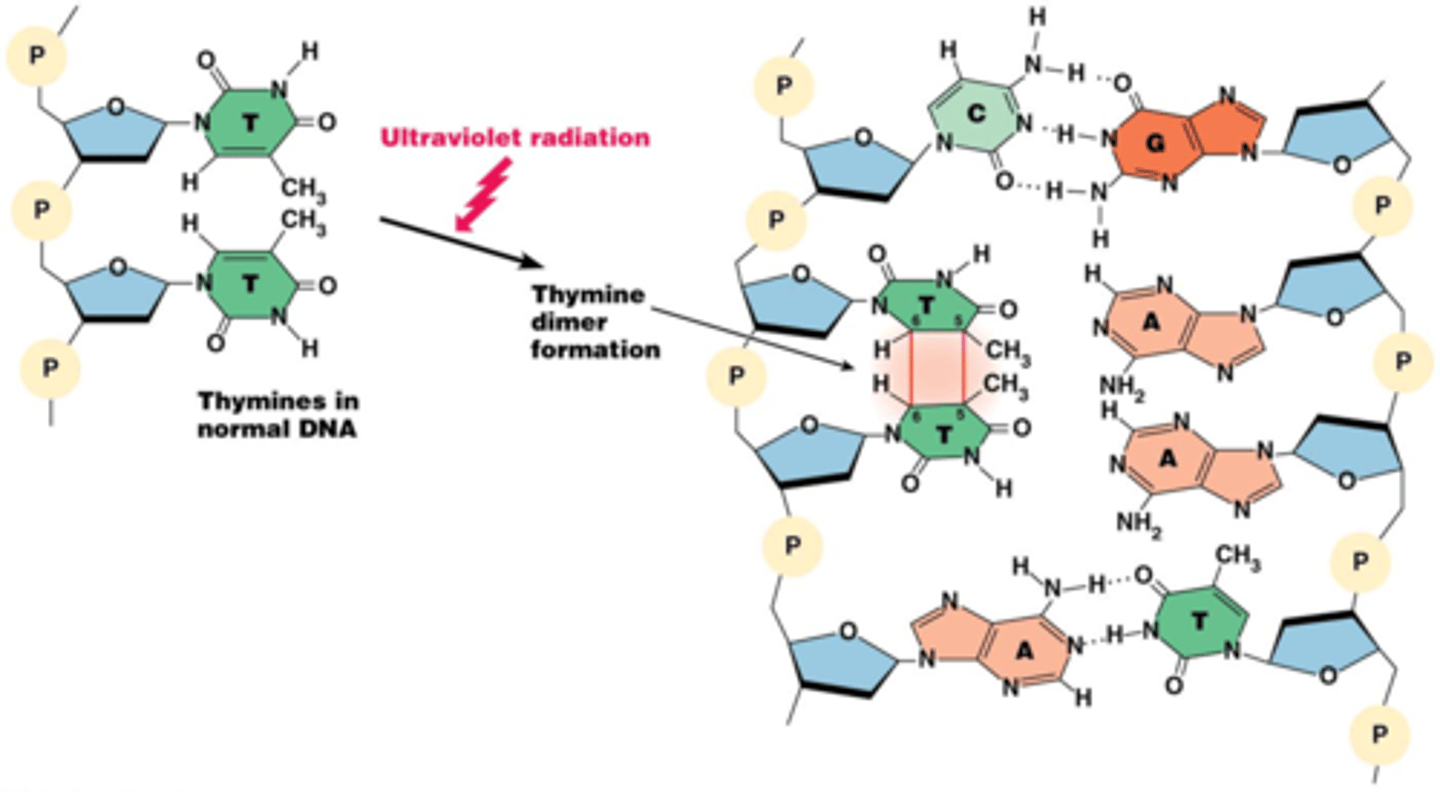
UV-stralen leidt tot thymine-dimeren (2 thymines covalent gebonden binnen dezelfde streng boven elkaar). Thymine-dimeren blokkeren replicatie-machinerie en kunnen leiden tot DNA breuken
X-stralen and radioactieve straling zijn ioniserende stralingen die nog schadelijker zijn voor DNA
->ontstaan vrije radicalen
We hebben veel verschillende soorten mutaties gezien op verschillende niveaus. Op welke 3 niveaus?
niveaus: atomair niveau, moleculair niveau, ...
-op niveau van de cel waarin ze voorkomt
-op niveau van DNA: gen, chromosoom, ...
-op niveau van het gevormde proteïne: het effect dat deze mutatie dus heeft.
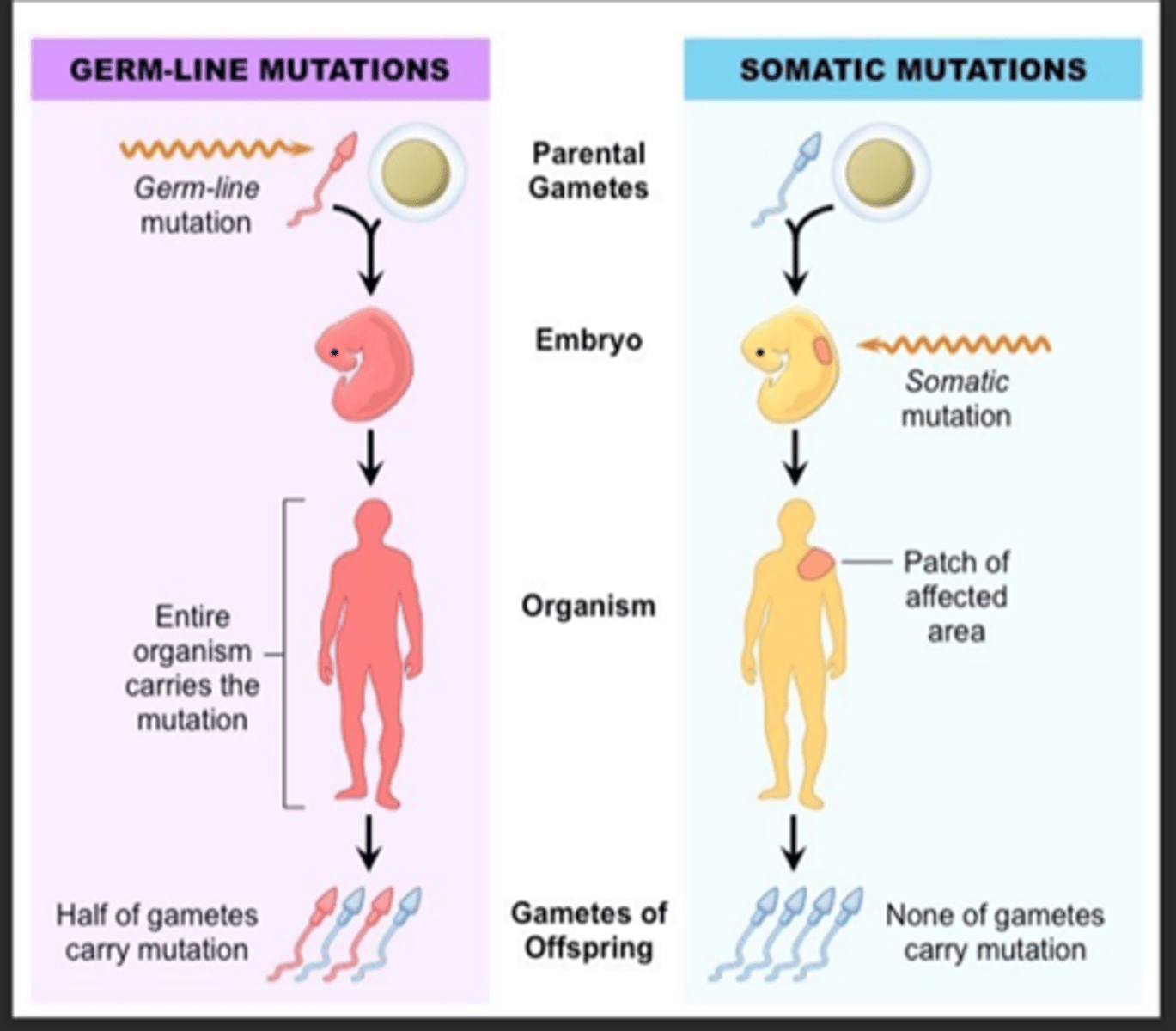
Tussen welke 2 soorten mutaties maken we onderscheid op niveau van de cel? Wat is het grote verschil ertussen?
-somatische mutaties (in lichaamscellen) zijn niet erfelijk
bv kanker: een lichaamscel is dan gemuteerd tot een kankercel
-germinale mutaties (in kiemcellen en gameten/geslachtscellen) zijn erfelijk
Hoe worden mutaties hersteld? geef ook alle voorbeelden die we hebben gezien!
door DNA-herstelmechanismen
(1)Fotolyase
(2)Base excision repair (BER)
(3)Nucleotide excision repair (NER)
(4)Mismatch repair
(5)Translesie synthese (geen echt foutenherstel)
(6)Herstel van dsDNA breuk
-via niet-homologe ‘end-joining’ (NHEJ)
-via ‘Synthesis-Dependent-Strand-Annealing’ (SDSA)
-via homologe recombinatie (HR)
afhankelijk van waar je zit in de celcyclus zullen andere mechanismen gebruikt worden
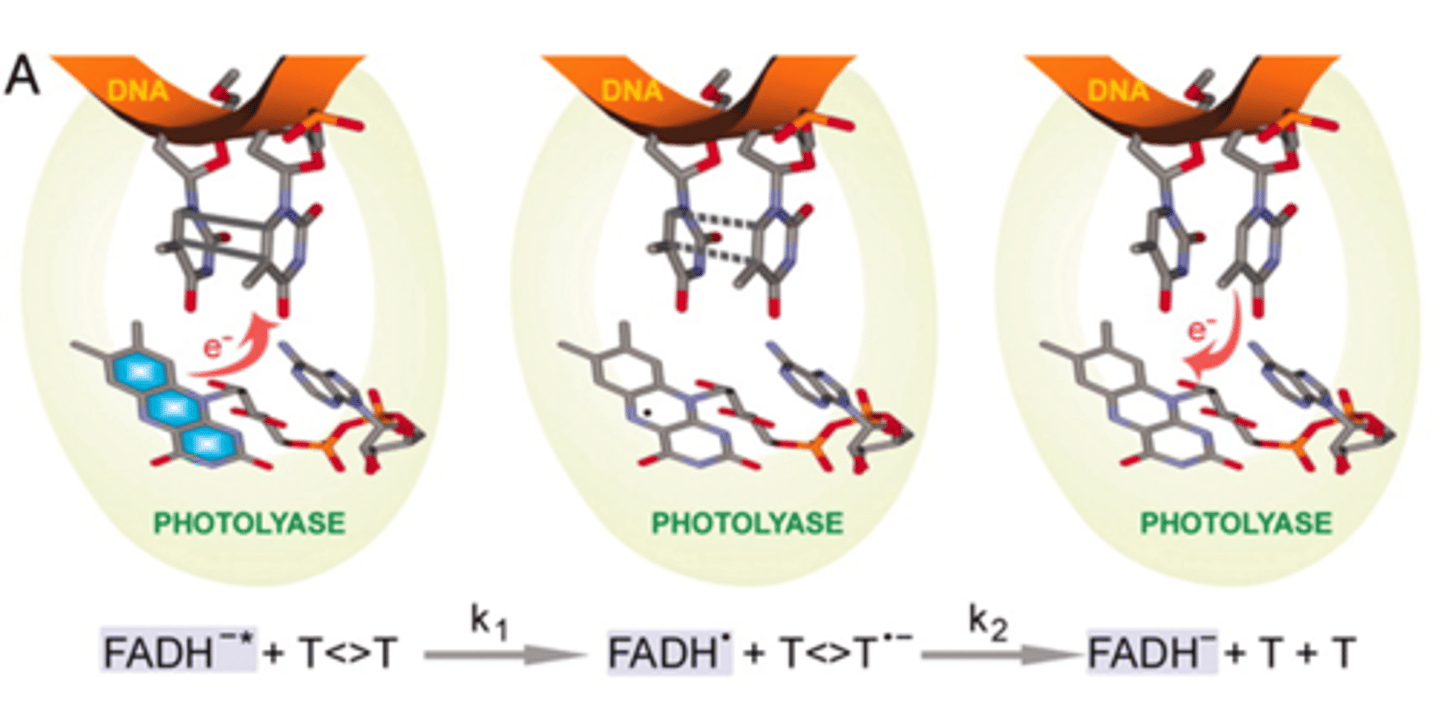
Fotolyase (thymine dimeren)
Alleen bij prokaryoten!
-fotolyase is een enzyme dat instaat is om thyminedimeren (T=T) kan verbreken
-Gereduceerde FADH- (geactiveerd door licht en functioneert als een electron donor
->transiënt een elektron afgeven ->verbinding wordt verbroken en juist hersteld
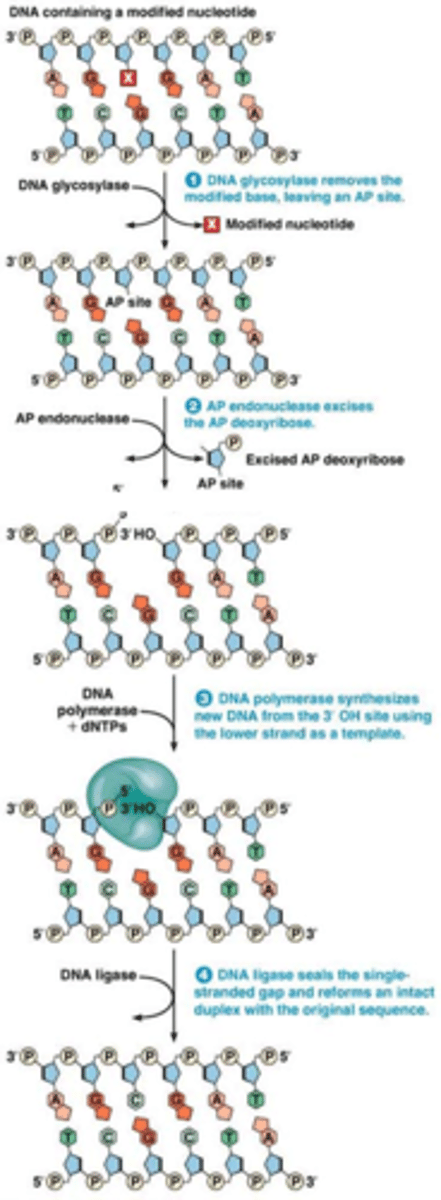
Base Excision Repair (BER)
Herstel gewijzigde of missende ringbasen:
1.DNA glycosylase verwijdert veranderde base (bijv. na deaminering). Deze stap is niet nodig bij verlies van base (bijv. na depurinering). de site die overblijft is een AP site
2.Endonuclease breekt de fosfodiësterbinding en lyase verwijdert deoxyribose fosfaat
3.DNA polymerase voegt het missend nucleotide toe op basis van complementariteit
4.DNA ligase verbindt de uiteinden
Waarom bevat DNA thymine ipv uracil?
Als er uracil in DNA zou zitten zou deze niet herkend kunnen worden door de base excision pathway en dus nooit hersteld kunnen worden
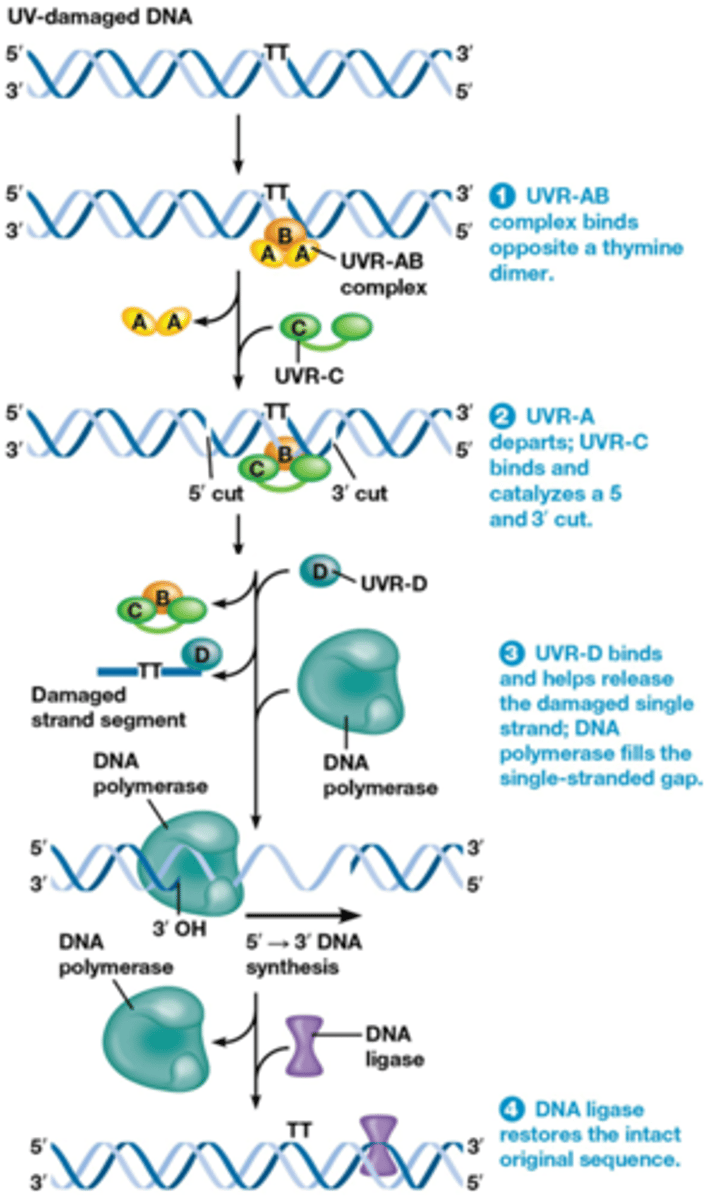
Nucleotide Excision Repair (NER), waar leidt het tot als er mutaties in zijn?
Thymine dimeren herstellen
1. UVR-AB herkent de afwijking (3D structuur is niet normaal door de TT)
-UVR-AB zal binden op de lichtblauwe streng (zonder TT)
2. Dissociatie van de UVR A, binding van UVR C (een endonuclease)
-UVRC zal knippen in beide strengen -> dissociatie van een complex
3. Recrutering van UVR D (een helicase) die zal de hybridisatie verbreken ->opening
-Polymerase synthetiseert DNA complementair met de streng
4. Ligase vult het gat op -> het is hersteld
Nucleotide Excision Repair’ (NER) ook gebruikt voor herstel van transcriptie-stop door gemodificeerde basen: ‘transcription-coupled repair’ -> legt uit waarom actieve genen sneller hersteld worden dan heterochromatine
Mutaties in NER eiwitten leidt tot Xeroderma pigmentosa: ilano, een 8 jarig belgisch jongetje dat huidtumoren krijgt in de zon: xprotectilano.be
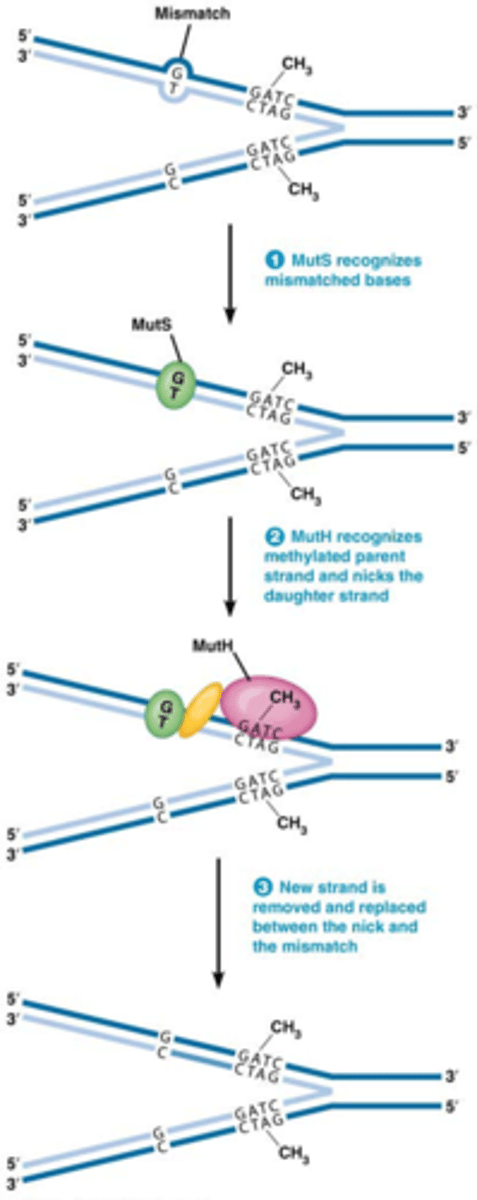
Mismatch repair
Foutenherstel na replicatie + proeflezing (1:10.000.000)
1. MutS herkent de GT mismatch
-Eiwit bindt
2. mutH wordt gerecruteerd en herkent de parentale streng*
-MUT H zal de dochterstreng verbreken
-MUT H zal exonuclease recruteren zodat die die DNA streng verder afbreekt
3. Opening zal opgevuld worden door DNA polymerase
Ligase zal het gat dichten
*Hoe weet het eiwit wat de parentale streng is?
bij bacterien=methylering van adenine
Bij eukaryoten= okazakifragmenten
Wat gaan prokaryoten doen bij ernstige schade aan het DNA?
Bij ernstige, niet onmiddellijk herstelbare beschadiging van de matrijs streng waardoor DNA polymerase geblokkeerd is maar helicase en topoisomerase verder gaat
->enkelstrengig DNA
-> SSB's en RecA binden hierop
->bypass polymerase komt uitzonderlijk tot expressie
Wat gaan eukaryoten doen bij ernstige schade?
translesie synthese:
Door DNA polymerisatie eta tot expressie te laten komen wat gespecialiseerd is in thymine dimeren te herstellen
-DNA polymerisatie η (eta) komt tot expressie bij zwaar beschadigde matrijs
-het zorgt er wel voor dat de replicatie verder gaat van de dochterstreng ookal is de matrijsstreng niet hersteld
->Dus TT in matrijs is niet hersteld!
->geen echt foutenherstel!
->kan wel correcte dochterstreng maken! hoe exact ga je toch aan chatgpt moeten vragen
Een dubbelstrengige breuk kan op 3 manieren, welke?
-via niet-homologe 'end-joining' (NHEJ)
-via 'Synthesis-Dependent-Strand-Annealing' (SDSA)
-via homologe recombinatie (HR)
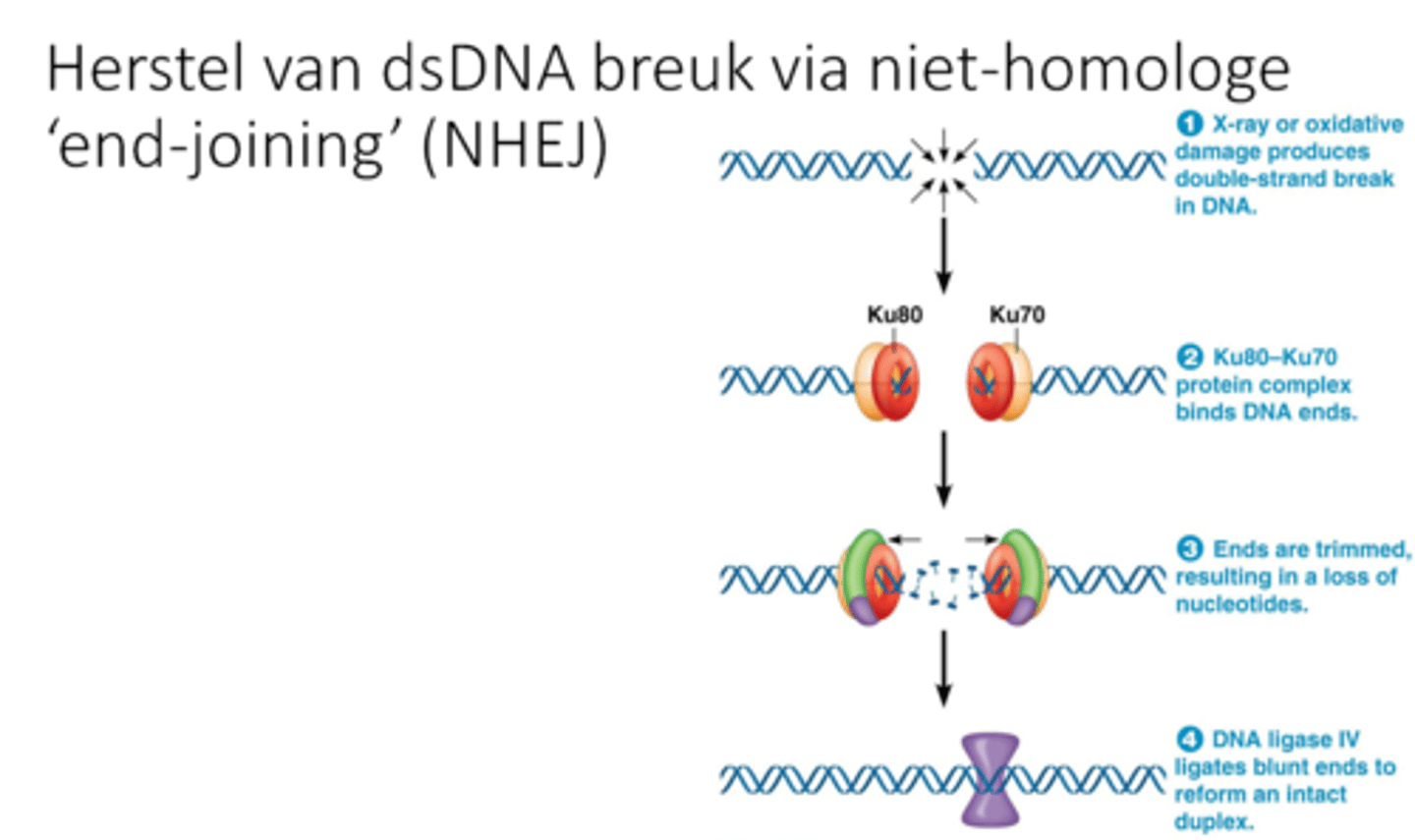
Herstel van dsDNA breuk via niet-homologe ‘end-joining’ (NHEJ)
Wat is het, in welk stuk van de celcyclus gebeurt het en wanneer gebeurt het ook nog foutief?
1. Dubbelstrengige breuk door X rays
Herkent door Ku80 en Ku70
2. Ze binden aan het uiteinde
3. Recruteren exonucleasen
-Exonuclease maakt mooie uiteindes* maar hierbij gebeuren er deleties
4. DNA ligase gaat opnieuw binden
->niet fouten vrij (=error-prone repair) door die deleties als dat in een exon is!
Dit gebeurt enkel in de G0 (uit de celcyclus, bv gedifferentieerde cellen) en G1 fase
Treedt ook op bij verlies van telomeren en kan tot chromosoomfusie leiden
-Telomeren worden korter bij elke celdeling
->Cel denkt dat er een breuk is, herstellen start
->chromosomen fuseren, dit is rampzalig voor de cel!
*Breuk kan symmetrisch (een rechte knip) of assymetrisch gebeuren
Bij assymetrisch: moeilijke enkelstrengige uiteindes die afgebroken moeten worden door exonuclease
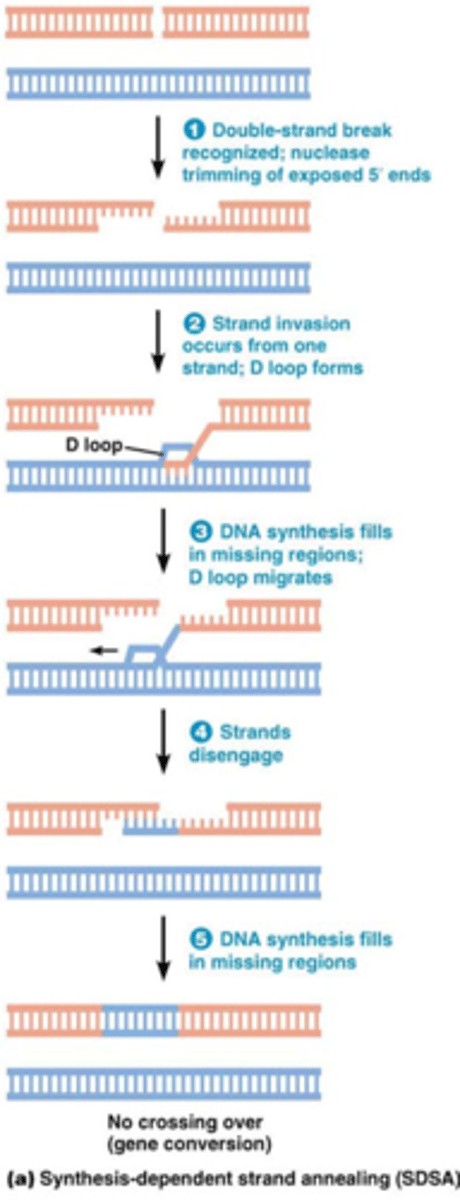
Herstel van dsDNA breuk via 'Synthesis-Dependent-Strand-Annealing' (SDSA)
Foutenvrij herstel als er een breuk optreed in homologe sequenties bv 2 zusterchromatiden (identiek aan elkaar, als er geen fouten in DNA replicatie zijn)
1. 5’ exonuclease de uiteinden trimmen
->enkelstrengige DNA uiteindes met vrij 3' uiteinde
-SSB zal binden, in dit geval Rad51 (niet getekend)
2.DNA streng van de ene zusterchromatide gaat in de andere zus incorporeren en complementair binden
-Er ontstaat een D (displacement) loop -> DNA streng wordt verplaatst
3. DNA polymerase gaat dna herstellen aan het 3' uiteinde complementair aan de blauwe streng
4. Dissociatie van de 2 strengen
5. DNA polymerase gaat DNA synthetiseren
-ligase gaan de gatenopvullen
Foutenvrij omdat blauwe streng identiek is aan de oranje streng, de blauwe streng is onveranderd!
Dit noemt men ook genconversie (geen crossing over)
Dit proces gebeurt enkel in en na de S fase want je hebt homologe zusterchromatiden nodig!
Herstel van dsDNA breuk via homologe recombinatie (HR)
verschil met SDSA: Beide strengen zijn betrokken bij de invasie
gelijkenis: ook foutenvrij
1. Nuclease trimt de uiteinden
2. Beide strengen invaseren elkaar en binden complementair met elkaar
3. twee 3’ uiteinden en DNA polymerase gaat beginnen synthetiseren
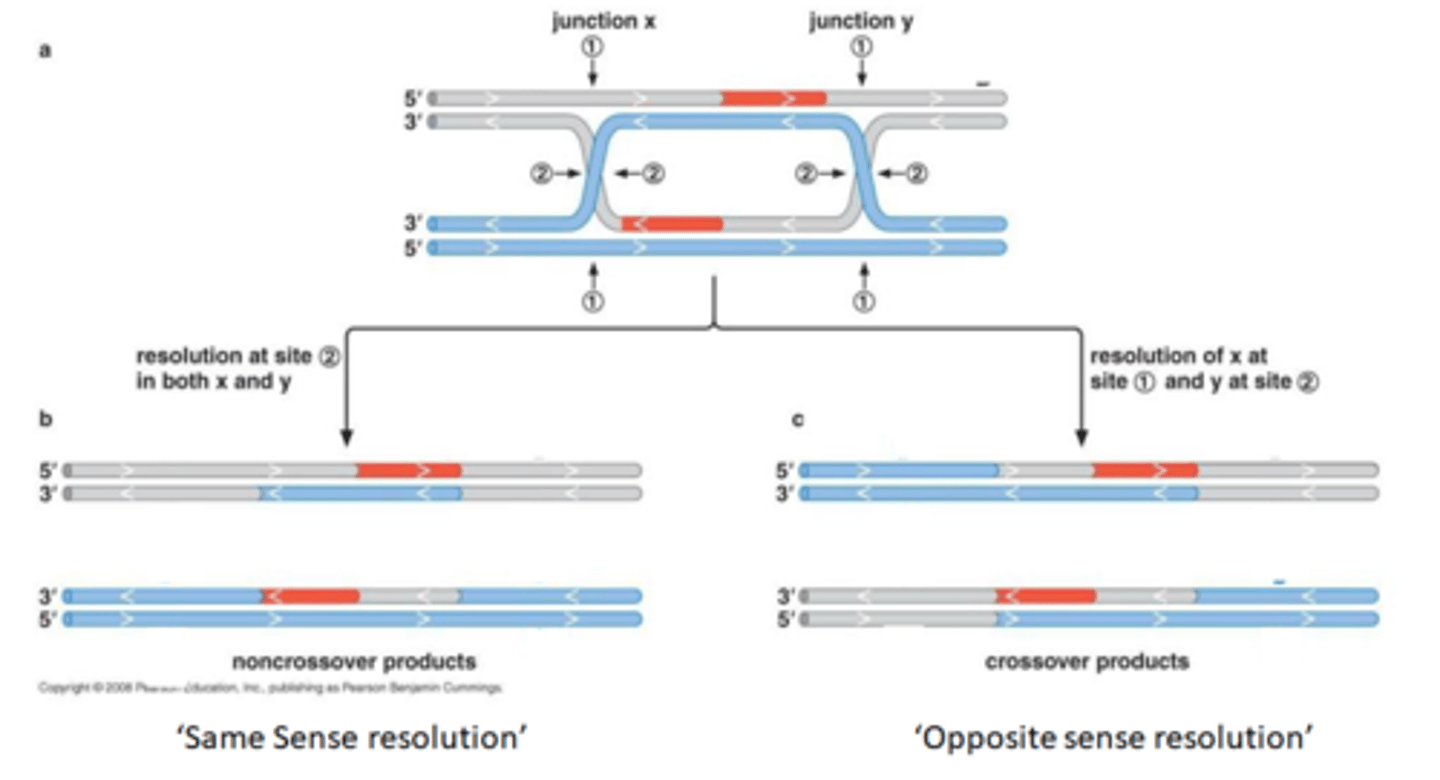
4. Holiday junctions worden gevormd: ze gaan kruisen waardoor ze niet meer uit elkaar kunnen gaan, ze zijn verstrengeld met elkaar door 2 fosfodiester bindingen die ontstaan
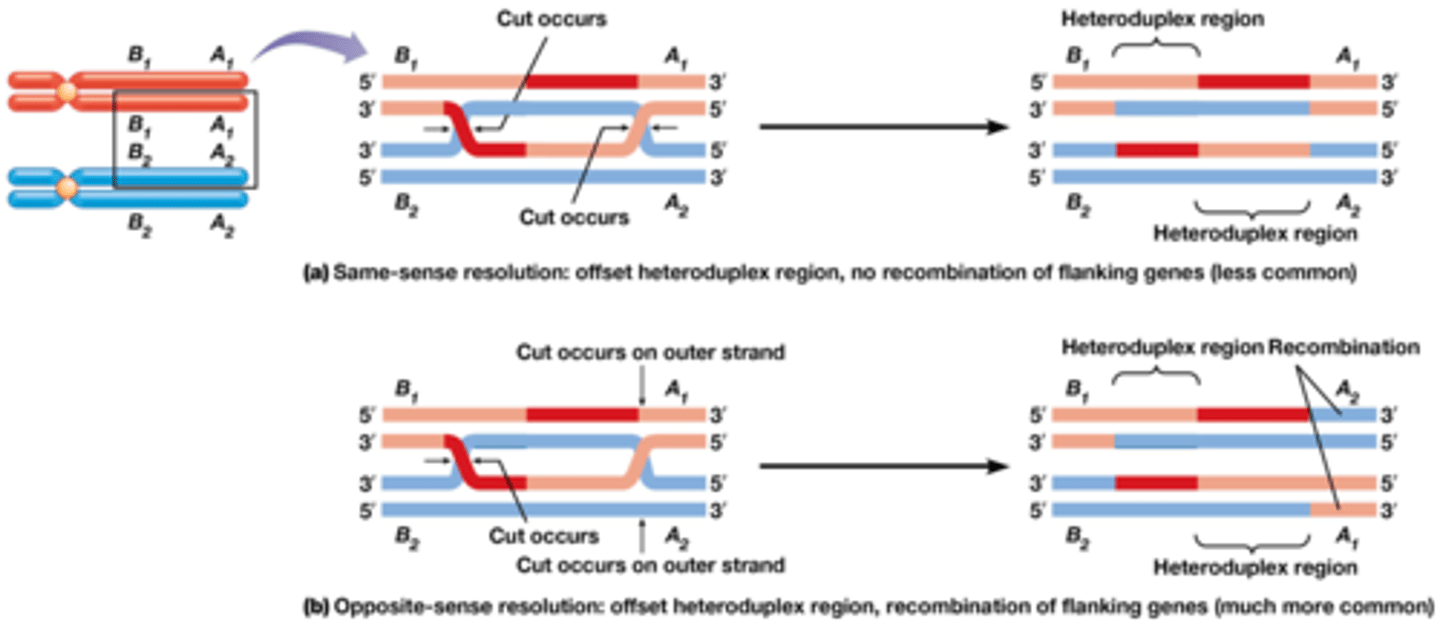
5. Twee oplossingen voor holiday junctions (nog extra foto volgend kaartje)
rood is nieuw gesynthetiseerd DNA, om ze uit elkaar te krijgen moeten knipjes gebeuren
-knip op 2(junction X en Y) zodat beide tegelijkertijd geknipt worden: same sense resolution ->gene conversie en geen crossing over
-knip op 1 bij X en op 2 bij Y zodat er een grote uitwisseling is tussen grijs en blauw ->crossing over
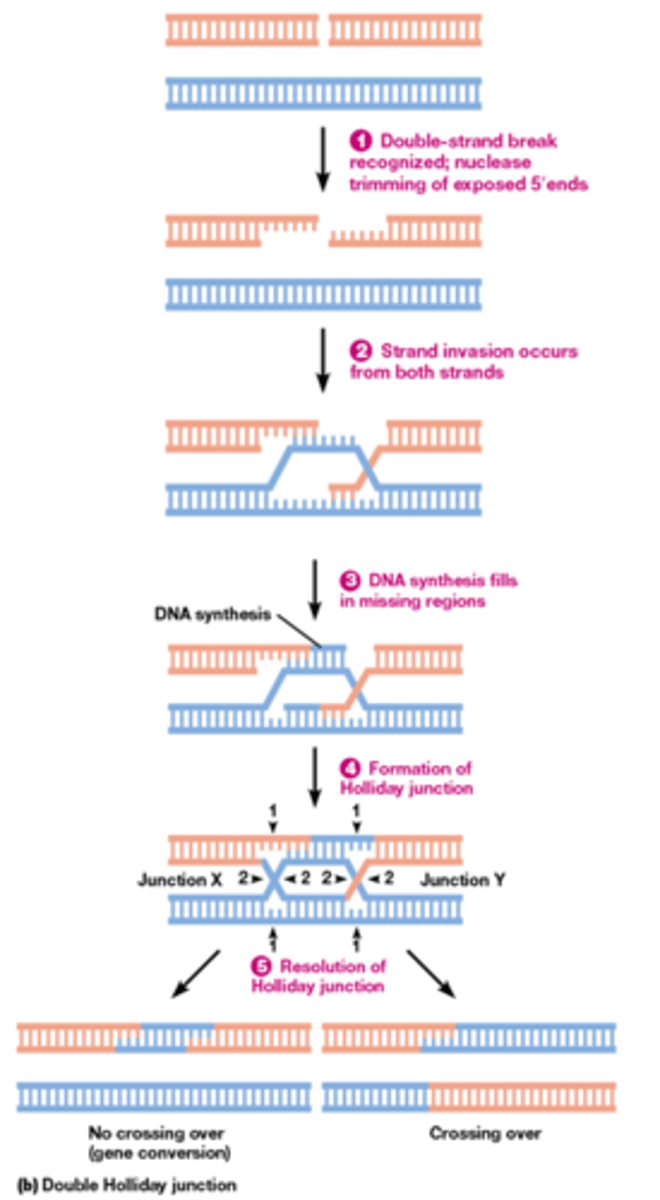
extra foto oplossing holiday junctions
5. Twee oplossingen voor holiday junctions
rood is nieuw gesynthetiseerd DNA, om ze uit elkaar te krijgen moeten knipjes gebeuren
-knip op 2(junction X en Y) zodat beide tegelijkertijd geknipt worden: same sense resolution ->gene conversie en geen crossing over
-knip op 1 bij X en op 2 bij Y zodat er een grote uitwisseling is tussen grijs en blauw ->crossing over
Wat doet de cel wanneer de DNA schade onherstelbaar is ?
apoptose= geprogrammeerde celdood
->de cel wordt vernietigd en verwijderd
->meer in H24
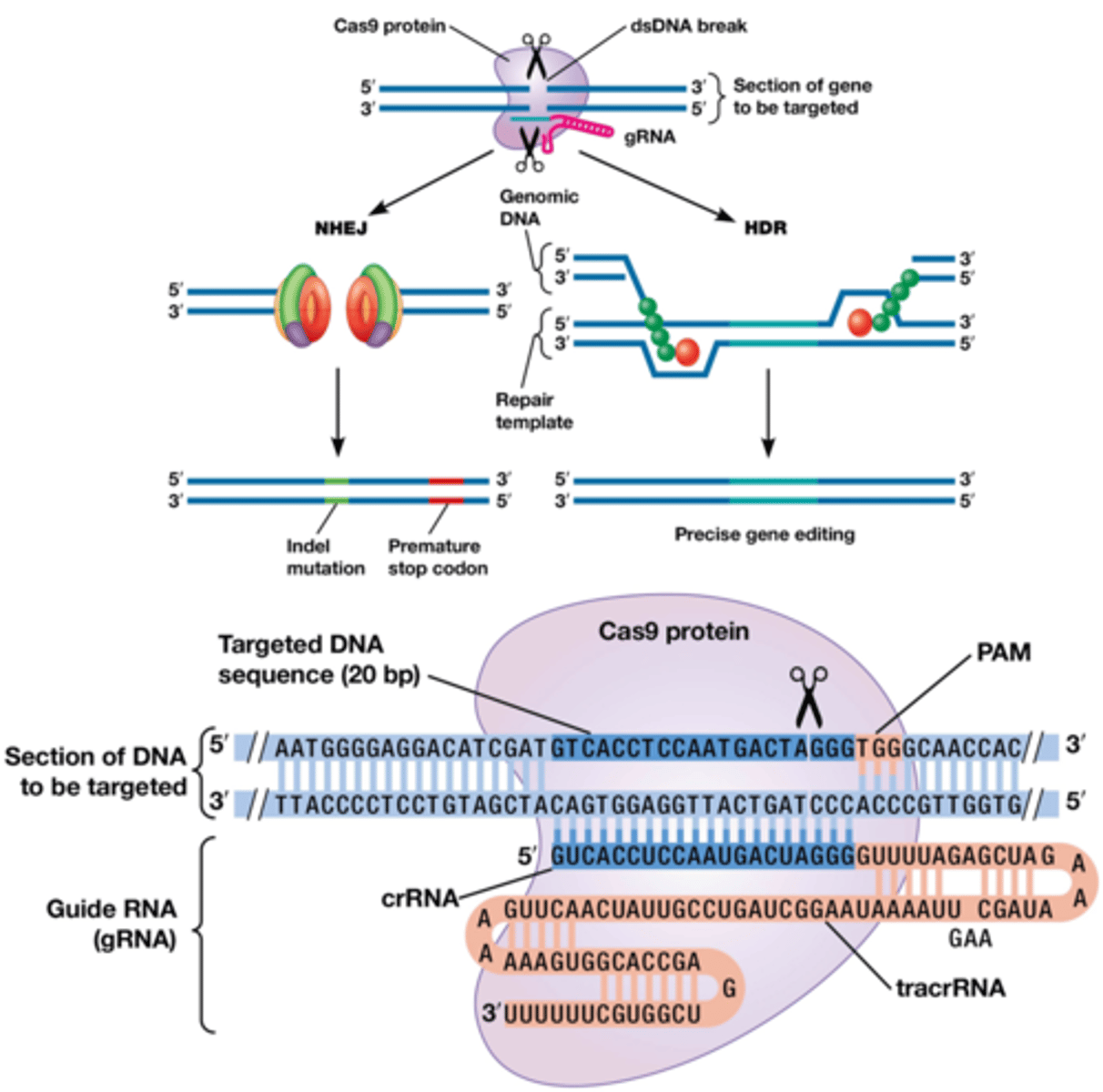
Wat is CRISPR/CAS?
CRISPR-Cas9: Gerichte DNA-bewerking waardoor we een mutatie kunnen inbrengen, genen uitschakelen, of zelfs nieuwe genen invoegen.
Cas9 Eiwit: Een endonuclease dat dubbelstrengige DNA-breuken kan maken op een specifieke locatie in het DNA. Dit gebeurt op de 'target'-sequentie (de targetsequentie moet naast XGG liggen, dit noemt men de PAM-sequentie)
Werking met gRNA: Cas9 wordt geleid door een guide RNA (gRNA), dat bestaat uit een
-crRNA (CRISPR RNA) (donkerblauw): 20 nucleotide lange sequentie en het bepaalt de bindingsplaats van Cas9 door complementaire basenparing met het target-DNA.
-tracrRNA (trans-activating CRISPR RNA): activeert het Cas9 eiwit
Herstelmechanismen van de cel:
-NHEJ (Non-Homologous End Joining): Kan kleine deleties veroorzaken, die leiden tot een stopcodon en het uitschakelen van het eiwit.
-HDR (Homology-Directed Repair): Hierbij wordt een hersteltemplate toegevoegd om de breuk nauwkeurig te herstellen. Deze template kan ook worden aangepast om specifieke mutaties in te brengen.
Probleem: Cas9 kan onbedoeld binden aan 'off-targets' als er slechts een klein verschil (bijv. 1 nucleotide) is.
->genoom editing op humane embryo's is verboden: mutaties op geslachtscellen zijn namelijk erfelijk!
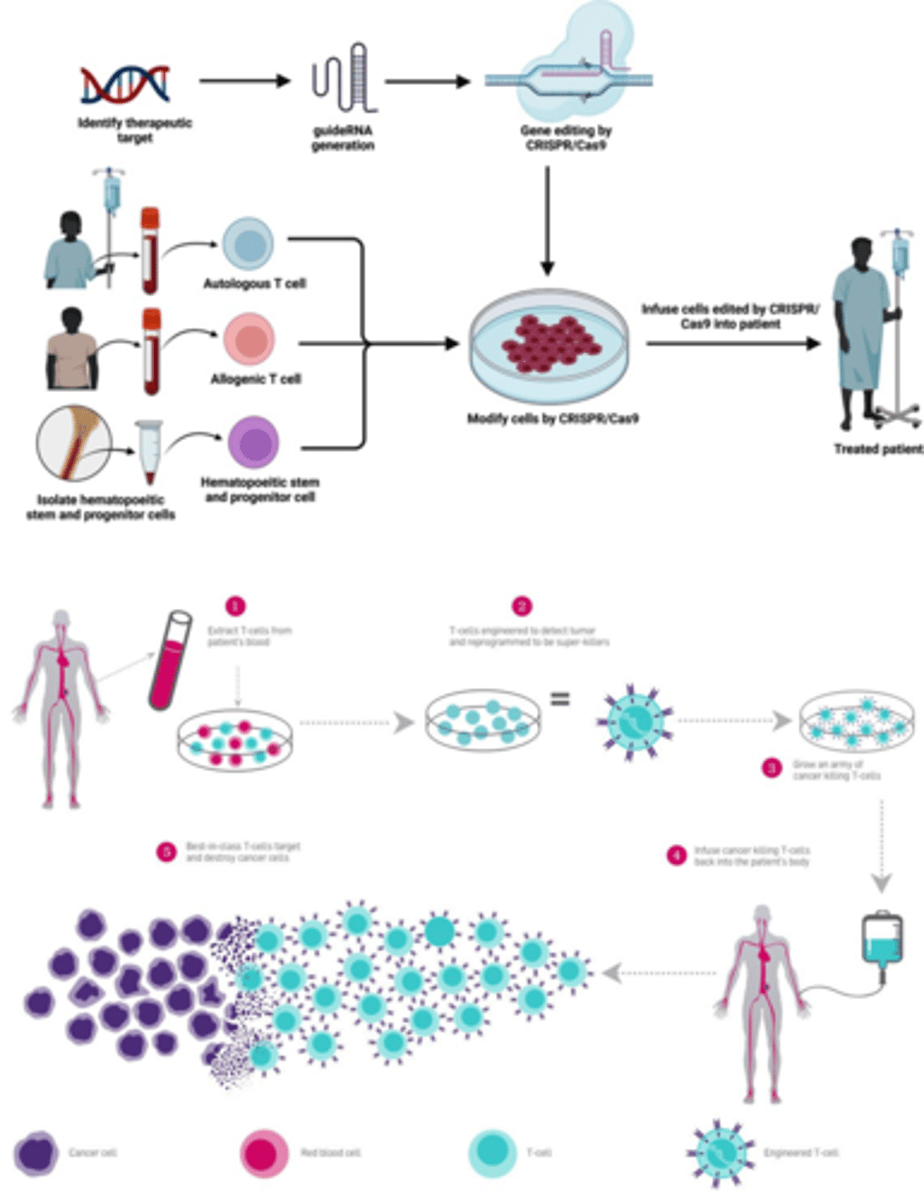
Voor wat wordt CRISPR nu al gebruikt?
-sikkelcelanemie
-leukemie (CAR T celtherapie) kinda uitbreiding maar wel super cool natuurlijk
Stap 1:
Extractie van T-cellen uit het bloed van de patiënt
Het proces begint met het afnemen van bloed van de patiënt. Hieruit worden T-cellen (een type witte bloedcel dat betrokken is bij het immuunsysteem) geïsoleerd.
Stap 2:
T-cellen worden genetisch gemodificeerd
De T-cellen worden in het laboratorium genetisch gemodificeerd met behulp van CRISPR-Cas, zodat ze tumorcellen kunnen herkennen. Ze worden herprogrammeerd door het inbrengen van een Chimeric Antigen Receptor (CAR), waardoor ze krachtige effectorcellen worden die specifiek kankercellen kunnen identificeren en elimineren.
Stap 3:
Vermeerdering van de gemodificeerde T-cellen
Stap 4:
Infusie van de gemodificeerde T-cellen terug in het lichaam van de patiënt
De nieuw geproduceerde T-cellen worden via een infuus in het lichaam van de patiënt gebracht.
Stap 5:
De T-cellen vernietigen doelgericht kankercellen
De gemodificeerde T-cellen richten zich specifiek op de tumorcellen en vernietigen deze, terwijl ze gezonde cellen in het lichaam zoveel mogelijk sparen.
17.3. Homologe recombinatie en mobiele genetische elementen
Wat is crossing over?
Homologe recombinatie van 2 niet-zusterchromatiden van homologe chromosomen
=uitwisseling van stukjes zusterchromatiden
->genetische variatie
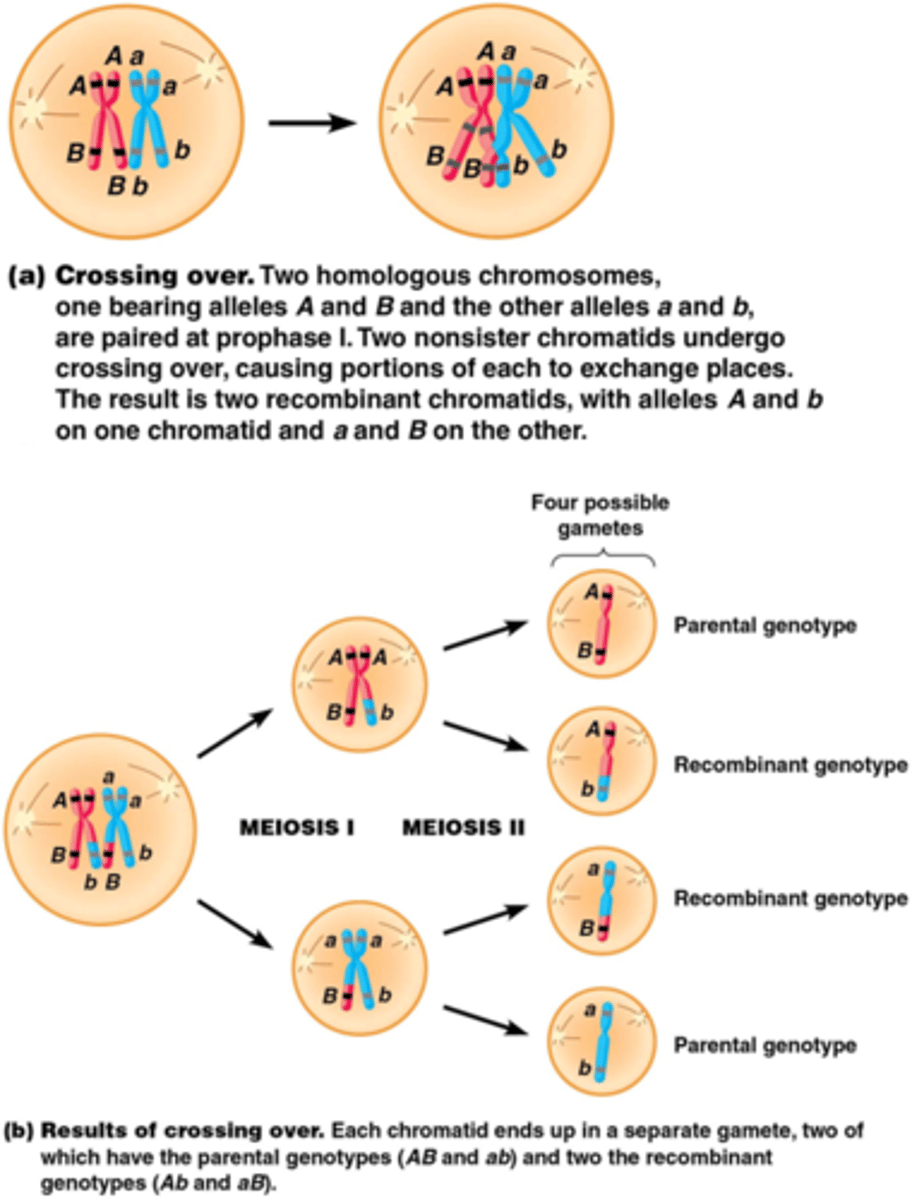
Hoe wordt homologe recombinatie geinitieerd tijdens meiose
tijdens de meiose komt een endonuclease (Spo11) tot expressie die breuken creëert
Zelfde principe als de vorige slide over homologe recombinatie, hier gebeurt het gewoon tussen 2 niet-zusterchromatiden
1. Spo11 knipt DNA
Nuclease trimt de 5' uiteinden
2. Rad51 bindt
3. Beide strengen invaseren elkaar en binden complementair met elkaar
4. eerst een D loop daarna dubbele Holiday junctions worden gevormd: ze gaan kruisen waardoor ze niet meer uit elkaar kunnen gaan, ze zijn verstrengeld met elkaar door 2 fosfodiester bindingen die ontstaan
5. heteroduplex regio: dubbelstrengige DNA molecule met 2 strengen van verschillende oorsprong (blauw met oranje)
6. DNA polymerase en ligase om het te herstellen
7. Twee oplossingen voor dubbele holiday junctions: volgend kaartje
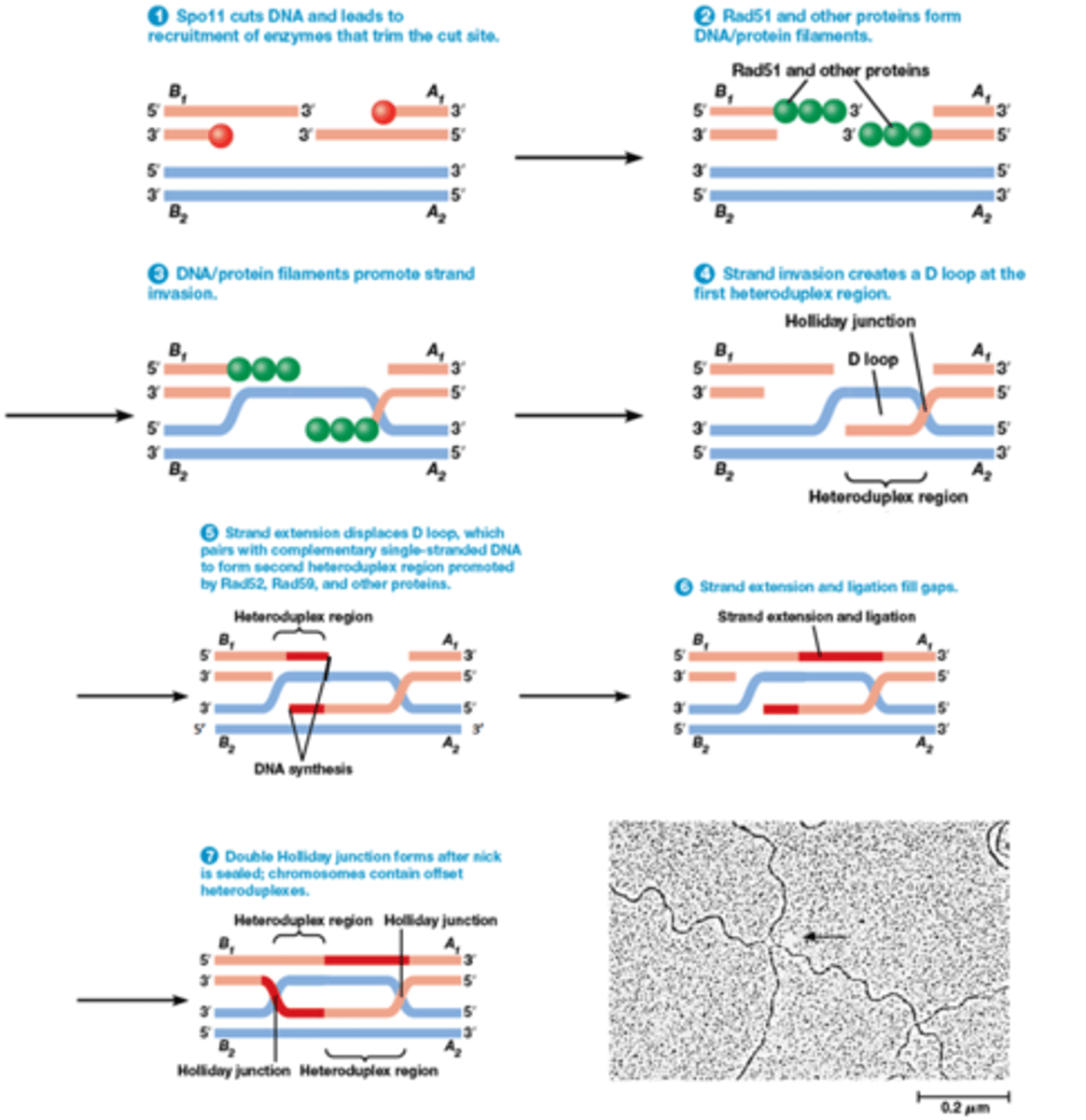
Welke 2 oplossingen zijn er voor de holliday junctions bij de meiose?
same sense
-> klein stukje verandert
opposite sense (meestal)
->grote verandering
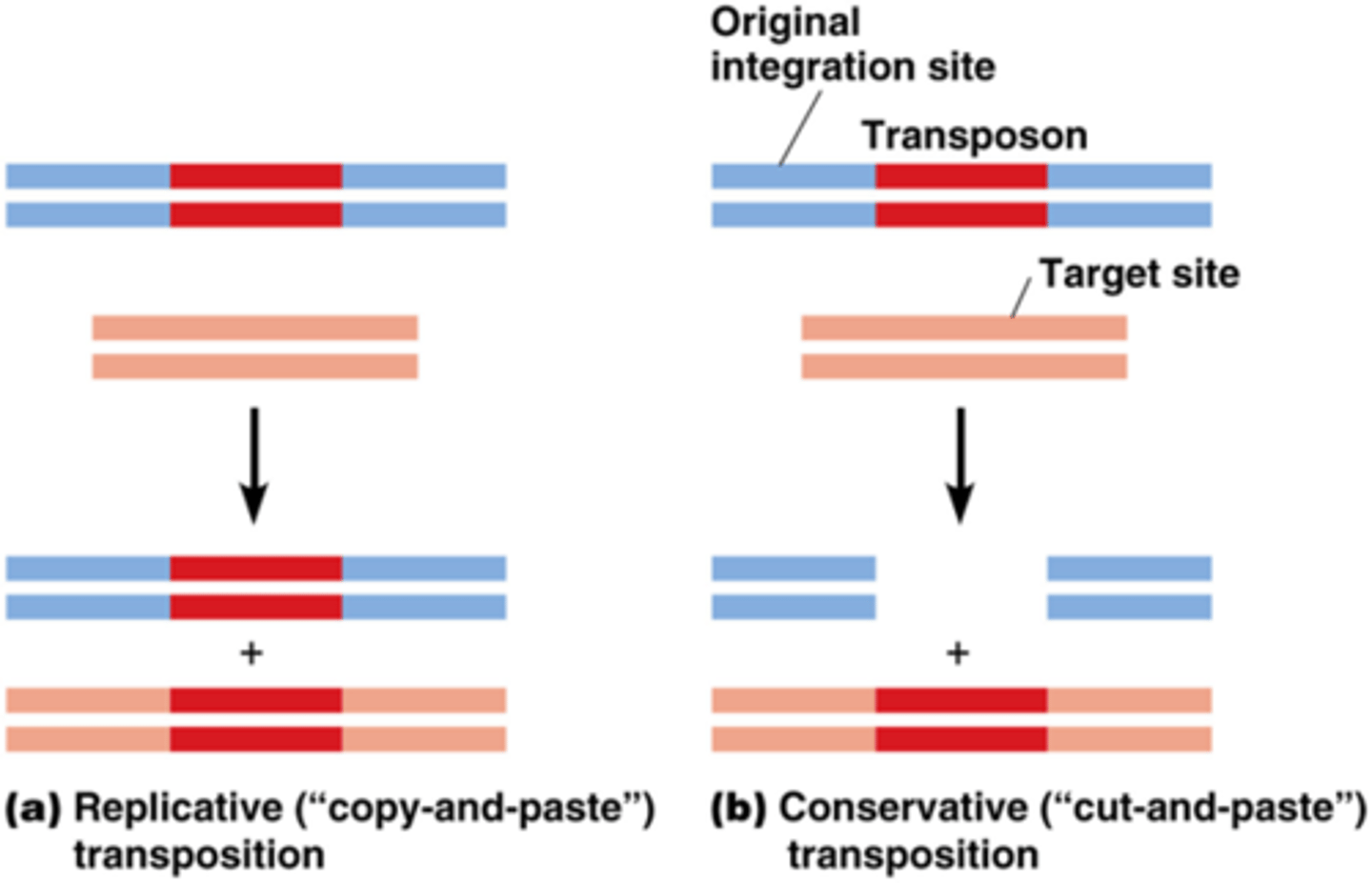
Crossing over is DNA uitwisseling tussen homologe chromosomen, hoe werkt DNA uitwisseling via transposons
gewoon verplaatsing, copy paste (gekopieerd) en cut and paste (eruit geknipt)
-> replicatieve en conservatieve transpositie
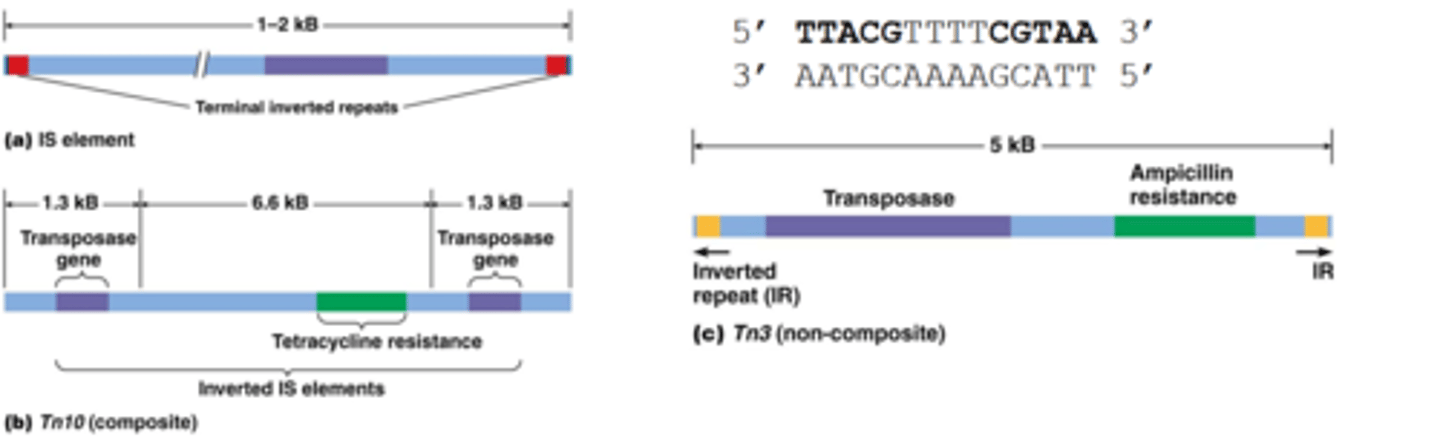
Welke DNA transposons hebben we in bacteria?
-IS (insertie element) bevat aan de uiteindes inverted repeats bevat in het midden een transposase (een enzyme dat DNA uitknipt en insereert ergens anders)
-inverted repeat is een nucleotidensequentie: TTACG dat wordt herhaald maar reverse complement: CGTAA, als er geen nucleotiden tussen de herhaling zitten is het een palindroom
2 soorten transposons in bacteria:
Samengesteld (composite transposon): 2 IS elementen met daartussen een ander gen dat meestal codeert voor anitbiotica resistensie
Niet samengesteld (noncomposite transoson): geen IS elementen en uiteindes zijn inverted repeats, het gen is geflankeerd door het ampiciline resistentie gen
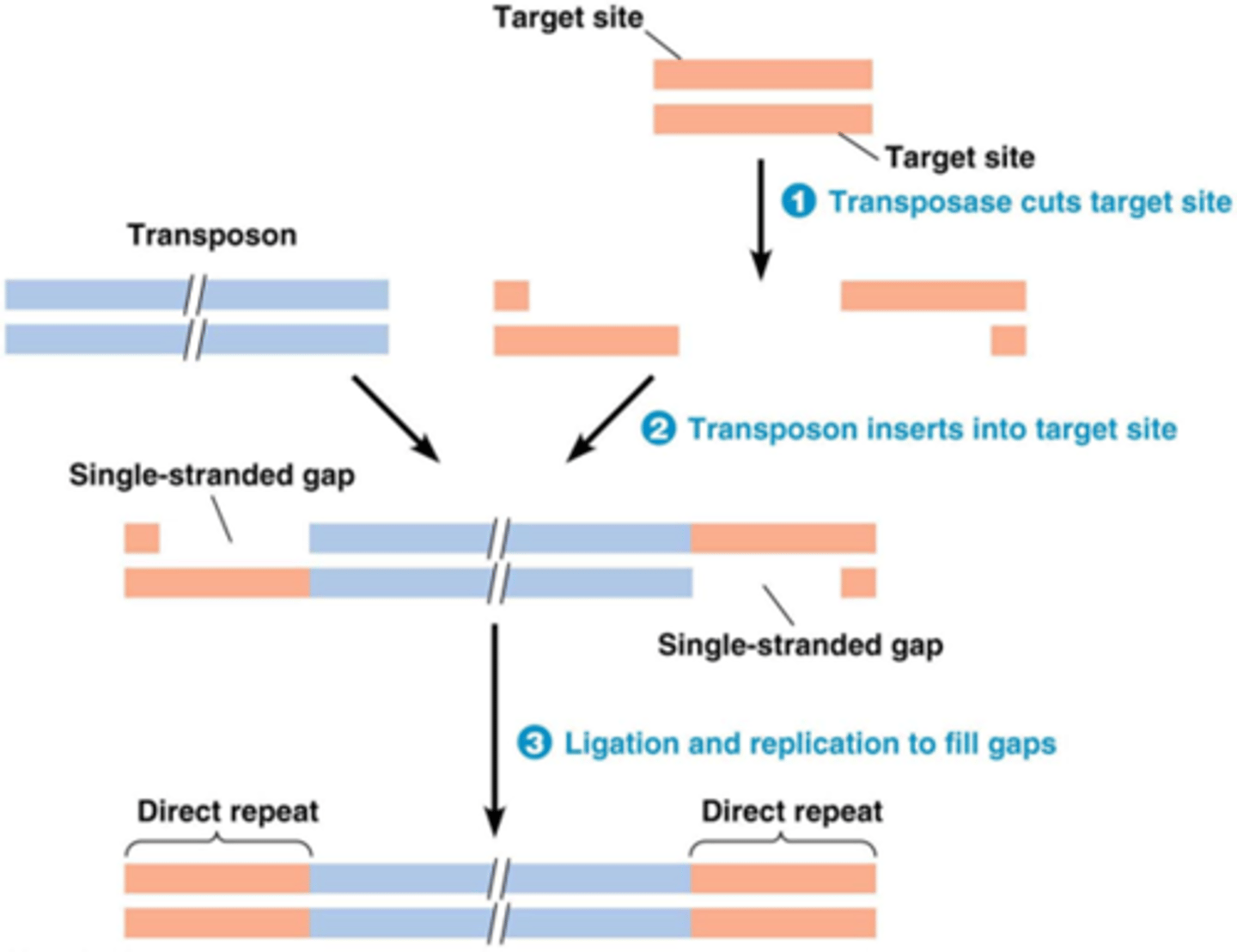
Hoe gebeurt de insertie van het DNA transposon in een nieuwe locatie?
Waartoe leidt de insertie van een DNA transposon?
1. Katalyse door transposase:
- knippen van beide DNA strengen op een verschillende plaats ->vorming van ‘sticky ends’= enkelstrengige stukken DNA)
2. Insertie van transposon
3.DNA repair door bacteriële enzymen: DNA polymerase + ligase
->‘target-site’ duplicatie
wat voor transposons hebben eukaryoten?
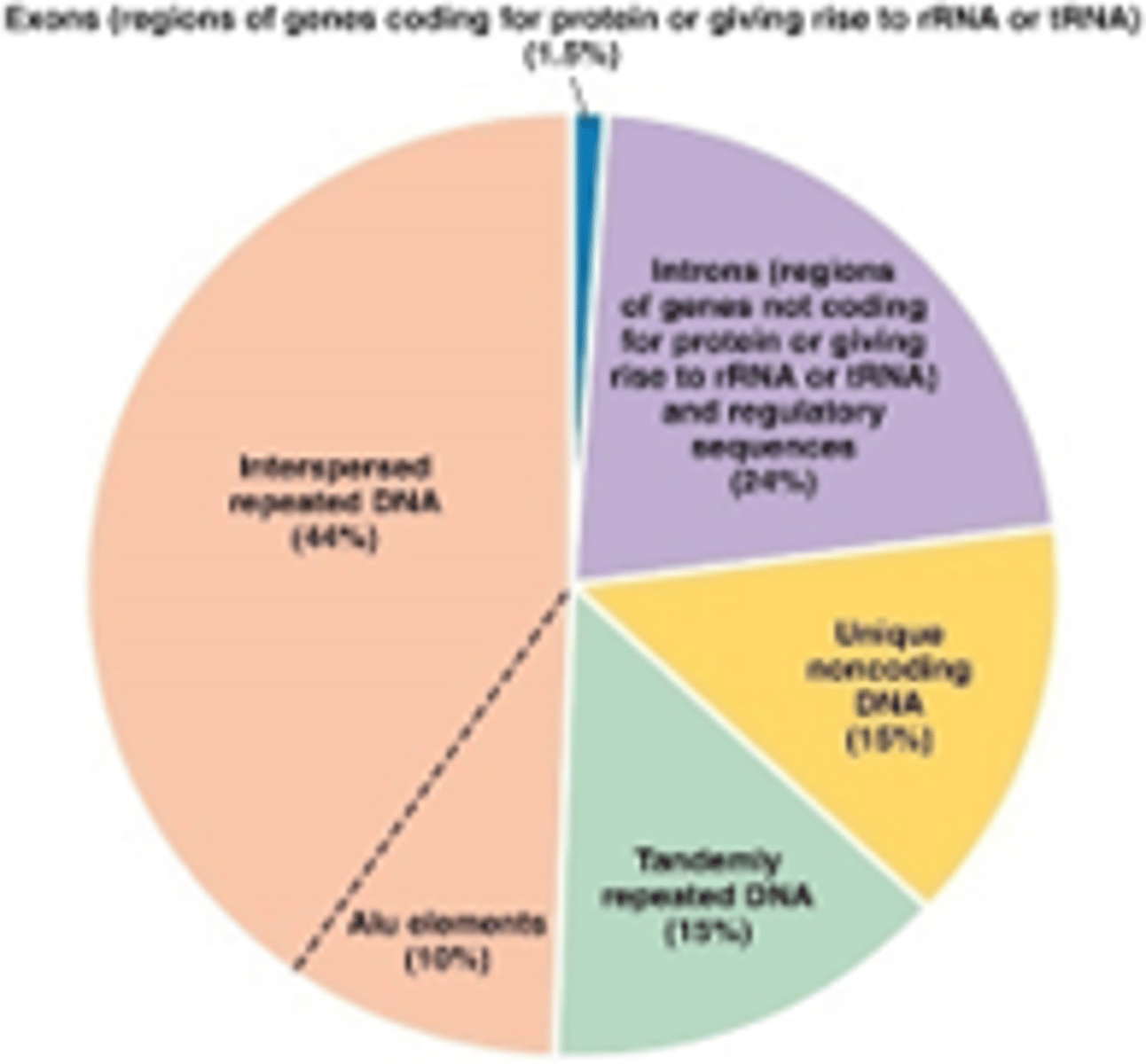
humaan genoom bestaat uit ca. 3% DNA transposons en >40% retrotransposons
Retrotransposons zijn mobiele genetisch elementen dat zich kunnen verplaatsen in het genoom via replicatieve transpositie met een RNA intermediair als tussenstap
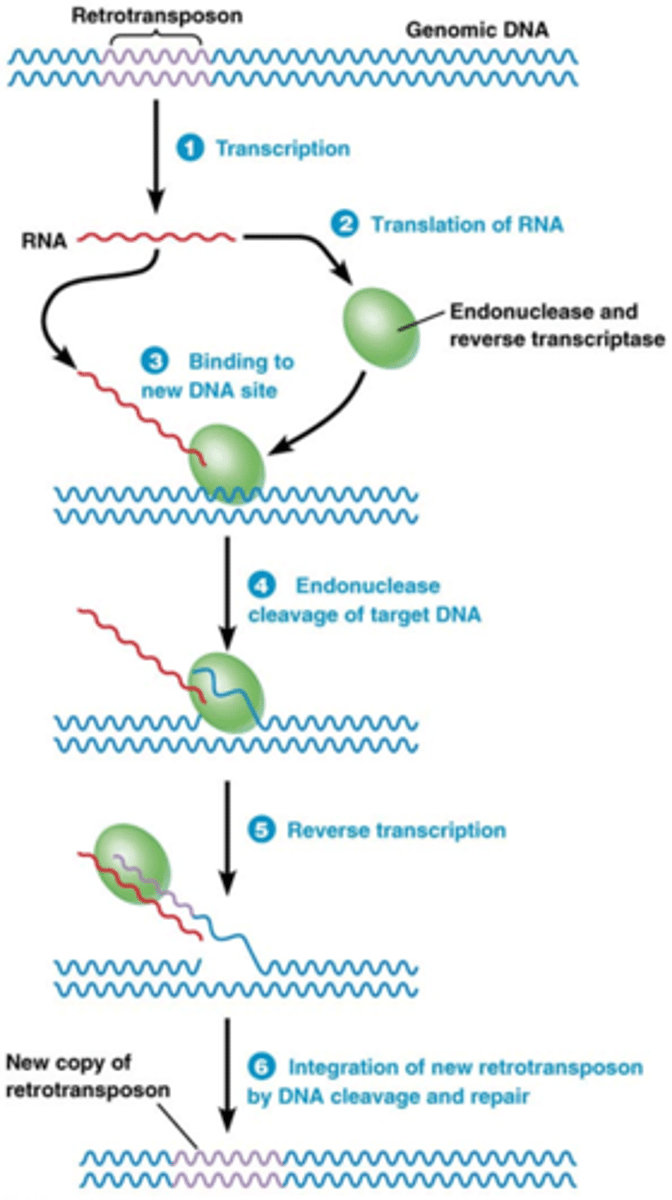
Hoe werkt de replicatieve transpositie?
1. Transcriptie door RNA polymerase II van de gastheer
2. Translatie: synthese van een eiwit met 2 activiteiten: endonuclease (breekt fosfodiester bindingen) en reverse transcriptase (van RNA DNA maken) activiteit
3. Binding op de nieuwe locatie in het genoom
4.Endonuclease knipt in 1 van de DNA strengen (breking fosfodiesterbinding)
5. RNA hybridiseert met een stukje van het enkelstrengig DNA, dient als primer voor de synthese van de eerste DNA streng door reverse transcriptase
->paars: DNA dat gesynthetiseerd is vanuit het RNA door reverse transcriptase
6. Knipping van de tweede DNA streng op de plaats van integratie, synthese van de tweede DNA streng, integratie en DNA repair (niet in detail want veel te ingewikkeld)
tot wat kan retrotranspositie leiden?
afhankelijk van de plek van de insertie van het retrotransposon in het genoom, het celtype en het type transposon kan het bijdragen tot de genetische variabiliteit.
het kan leiden...
Tot de bevordering van homologe recombinatie dat kan leiden tot exonshuffling of exonduplicatie (wordt duidelijk volgend hoofdstuk)
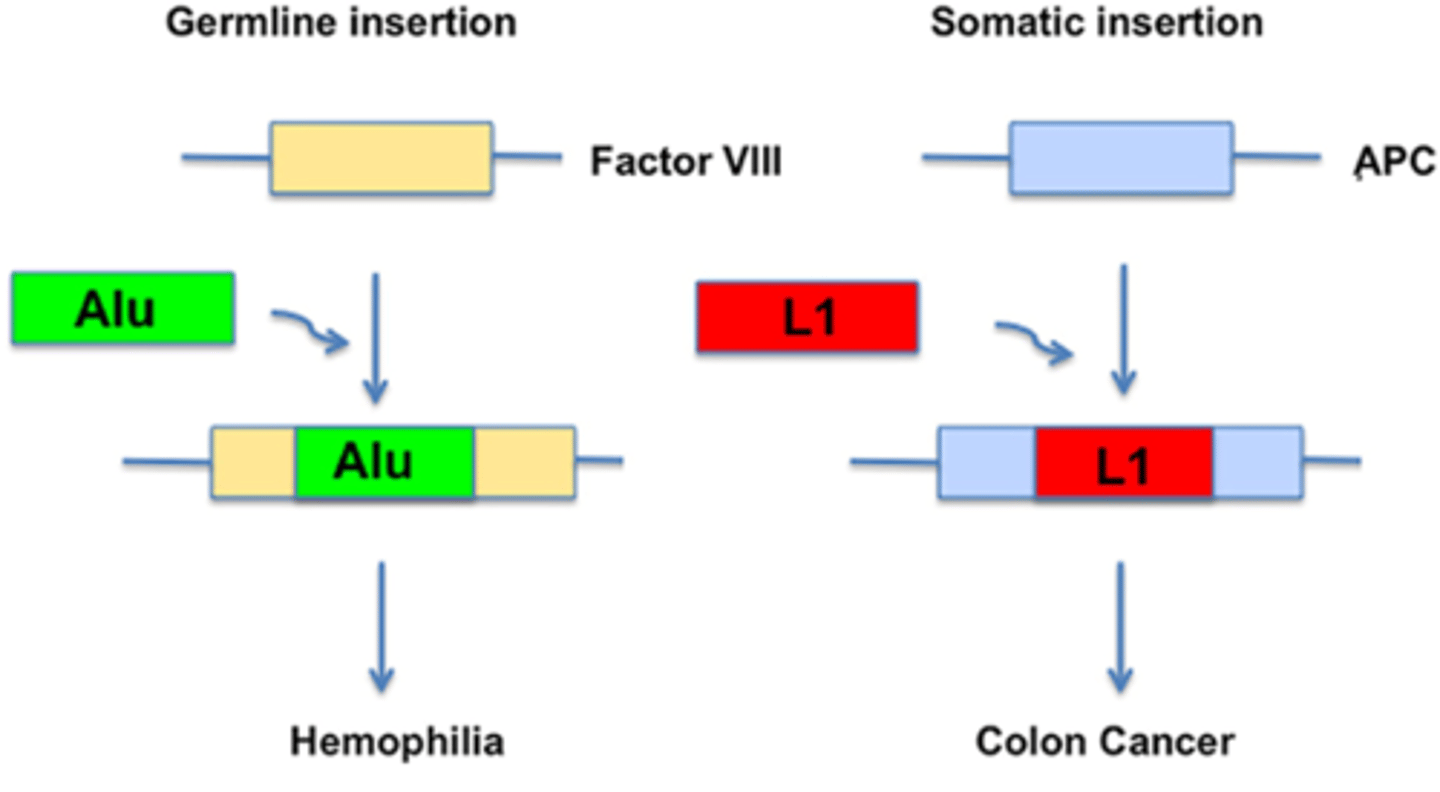
Tot de inactivatie van een gen:
Hemofilie is het effect van een retrotranspositie, alu repeat is geinsereerd in factor 7 in de geslachtscellen
->erfelijke bloedstollingsziekte: hemofilie
Als je L1 insereert in het APC gen krijg je darmkanker omdat het gen niet meer actief is