bio MOL chap 2.1 ADN: composition, strucutre et compactage
1/52
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
53 Terms
Noyau cellulaire = ?
Organite eucaryote
Matériel génétique
Réplication ADN
Transcription ARN
Noyau : exceptions
Anucléées → hématies
Binucléées → hépatocytes, cell cancer
Plurinucléées → 2 origine :
syncytium
plasmodes (ex:ostéoclaste)
Composition du noyau
Enveloppe nucléaire
Nucléoplasme
Chromatine
Nucléole
Matrice nucléaire
Enveloppe nucléaire
2 membranes
Continuité RER
Pores nucléaires
Trafic nucléocytoplasmique
Nucléoplasme
Milieu aqueux
Support structures
Transport molécules
Régulation réactions
Chromatine = ADN + prot
ADN + protéines
États condensation
Euchromatine : centre, accessible, reserve enz
hétérochromatine : compacte, périphérique
chromosome = unté de la chromatine
Nucléole
Transcription ARNr
Assemblage (biogenèse) ribosomes
Matrice nucléaire
Réseau (fibre nucléaire) polymère lamines = filament intermédiaire type V
Soutien cell : noyau structure + envloppe
Organisation chromatine/chromosome/transcription/réplication
Laminopathies
Mutation gène LMNA
Lamine A/C
Déformation noyau
progérine = forme tronqué lamine
ADN composition
Polymère de nucléotides = Base azotée + Pentose + Phosphate
Support info génétique
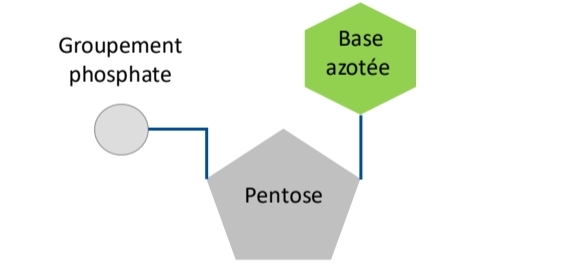
Bases azotées
Puriques → A, G
Pyrimidiques → C, T (U ARN)
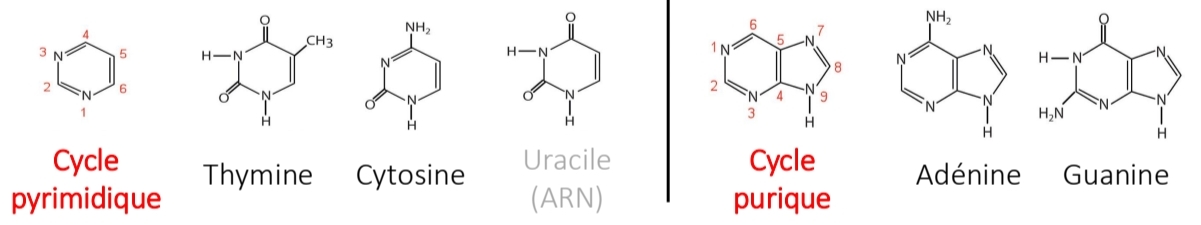
Pentose ADN
Désoxyribose
C2’ = H
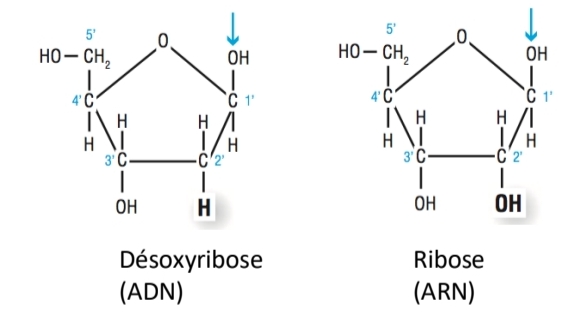
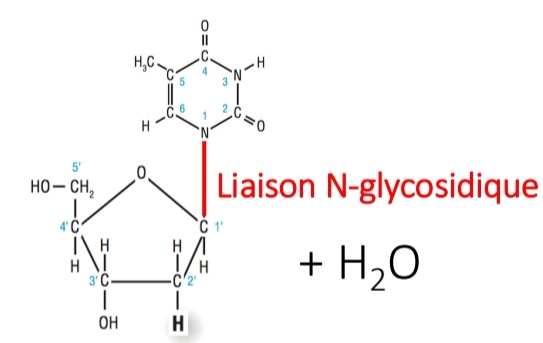
Nucléoside vs Nucléotide
Base + pentose
Liaison N-glycosidique
vs
Nucléoside + phosphate
Liaison phosphoester
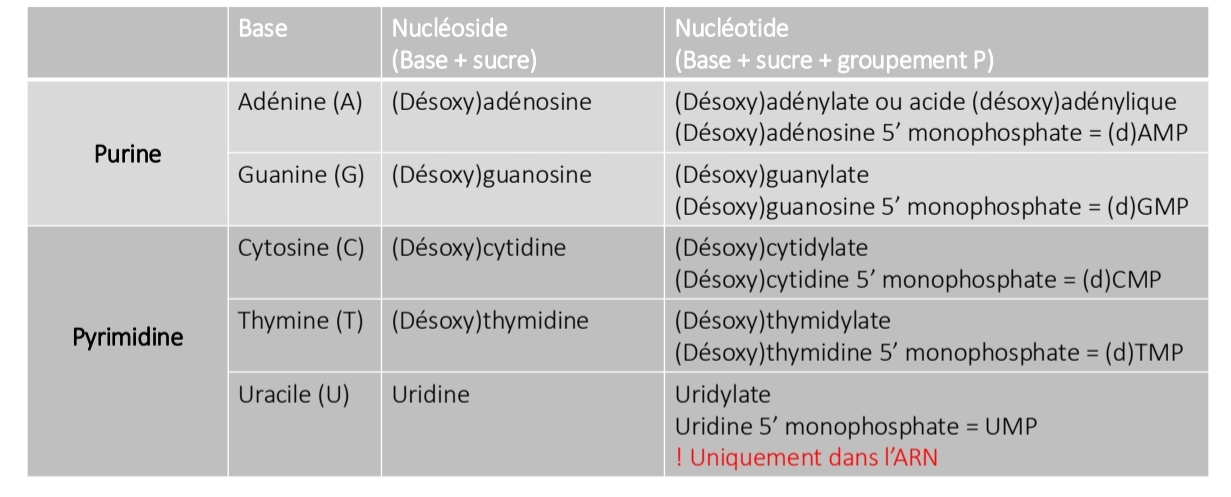
Liaisons ADN
Phosphodiester 3’–5’ |e| nucléotide
Chaîne polynucléotidique
Polarité ADN
Extrémité 5’ P
Extrémité 3’ OH
Lecture 5’ → 3’
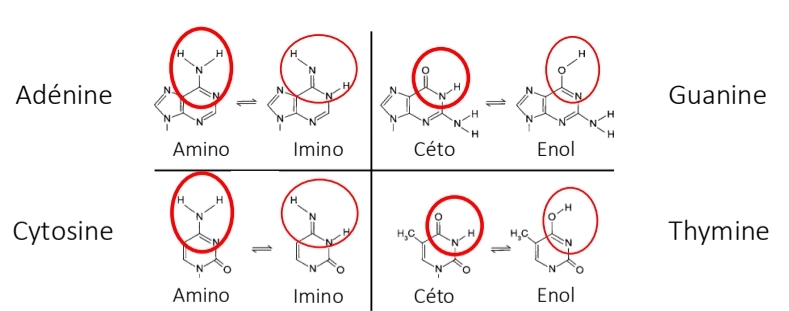
Tautomères des bases
isomère ≠ ds la position et liaison des atome (mm formule chimique)
Amino* / imino
Céto* / énol
pH dépendant : déplace H+ + 2ble liaison autre place
*mileu physio
Double hélice : propriétés
Antiparallèle
Complémentaire
Hélicoïdale
Antiparallélisme
1 brin 5’→3’
1 brin 3’→5’
Complémentarité
A = T (2 liaisons H)
G ≡ C (3 liaisons H)
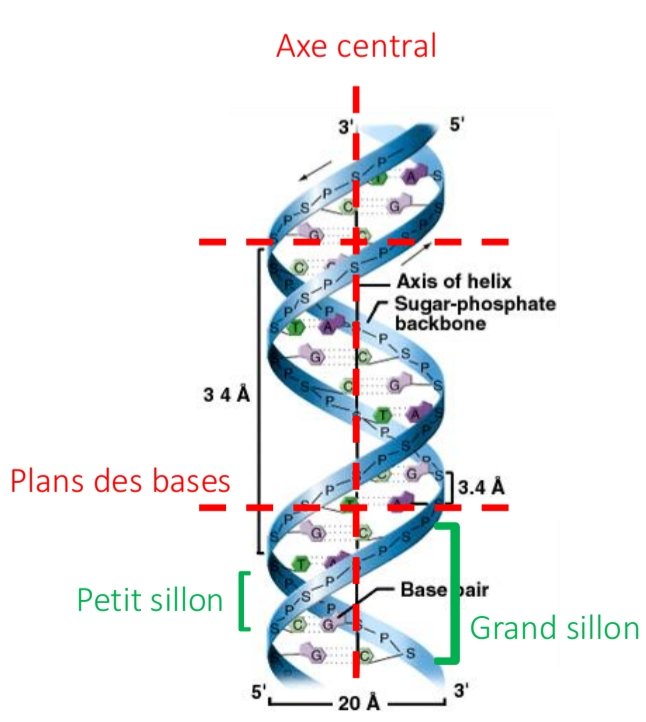
Forme B de l’ADN
configuration hélicoïdale
2ble Hélice droite
plan des base ⟂ axe hélice
Forme majoritaire
Grand sillon + Petit sillon
Règles de Chargaff
Quantité de purines ≈ quantité de pyrimidines (A+G ≈ T+C)
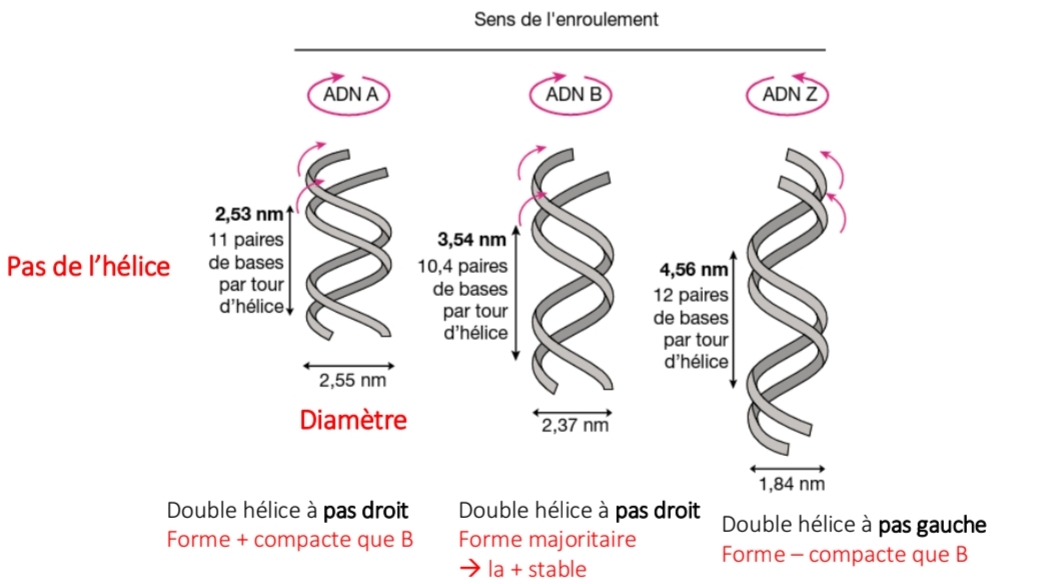
Conformations ADN
ADN B → majoritaire
↓ {ionique} = condition intracell
ADN A → déshydratation
↓ {eau} ↑{sel} ADN Z → pas gauche
↑{sel} + alcool
in vivo qd cytosine méthylé
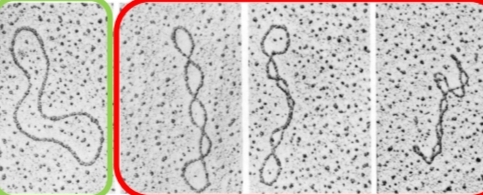
Topoisomères
Même séquence
Topologie différente (3D globale )
Enlacements différents
étar relaché → + stable
état superenroulé → superhélice
superenroulement + : ds sens
superenroulement - : ds sens inverse
CSQ nbr enroulement ↓ , ↓ torsion, ↑accessibilité
Topoisomérases : rôle + étape
enz controle structure topologique ADN : interchange les topoisomère ADN
Capables d'introduire ou d'éliminer des super-enroulements
IMPORTANT : réplication, réparation,transcription
coupure ADN nv ester 1ε& sucre/PO4
passage segm- ADN
ligation
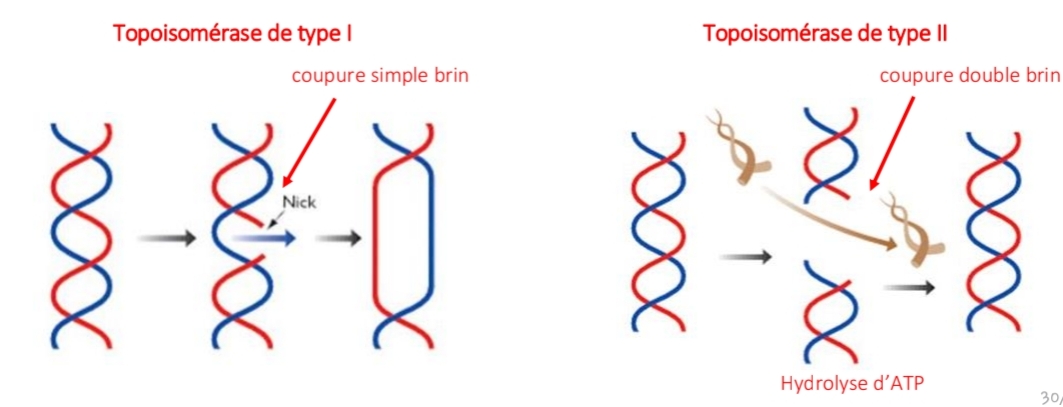
Topo I vs Topo II
Topo I → 1 brin
Topo II → 2 brins + ATP
Topo procaryotes
Type 1 : Topo I - III→ relâche ADN
Type 2 : Topo II/DNA gyrase - V→ introduit sous-enroulements
Topo eucaryotes
Type 1 : topo I-III → relâche
Topo II → enlève supertours
Topoisomérases : clinique
Inhibiteurs de la gyrase bactérienne: Antibiotiques
→ inhibe réplication
Inhibiteurs des topoisomérases humaines: Anticancéreux
→voir dias 32
Propriété de ADN
Dénaturation ADN
methode+facteur influence
Séparation brins : 2 simple // liaisons H
Température ↑
pH ↑
Agents chimiques
Température fusion :50 % ADN dénaturé
facteur influence dénaturation
• Température
• % de G≡C (plus stable que A=T)
• longueur de la chaine
• pH
• composition ionique de la solution
Propriété de ADN
Renaturation ADN
Refroidissement
Brins complémentaires
Processus réversible
Génome
Chromosome
gène
Locus
Allèle
Total ADN organisme
ADN condensé : 1 chromosome = 1 ADN
Segment ADN qui transcrit ARNpm (→ protéine)
Position gène sur chromosome
Autre versions gène : Séquence nucléotide ≠
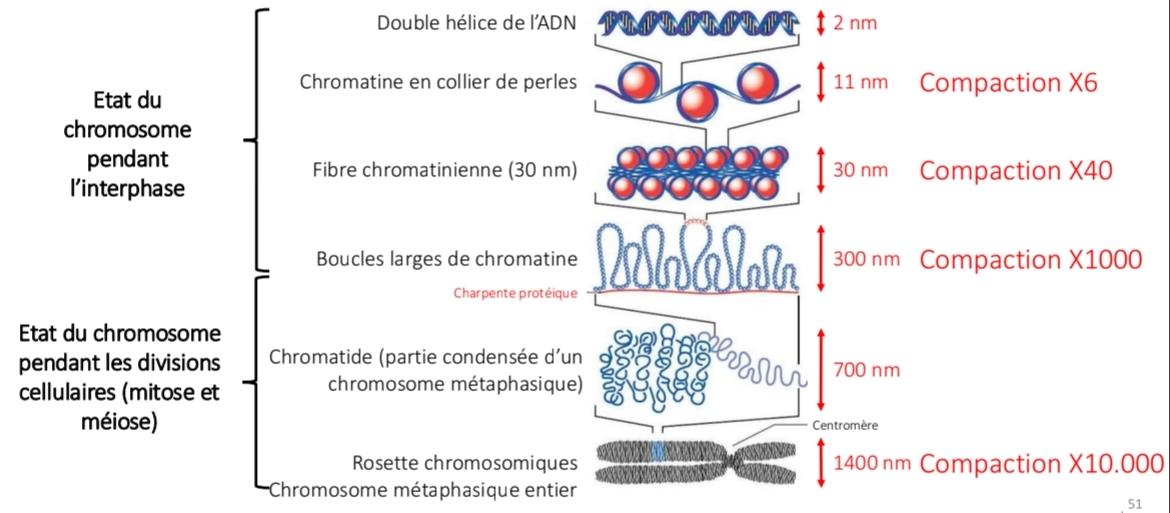
Nucléosome
146 pb ADN
Octamère histones
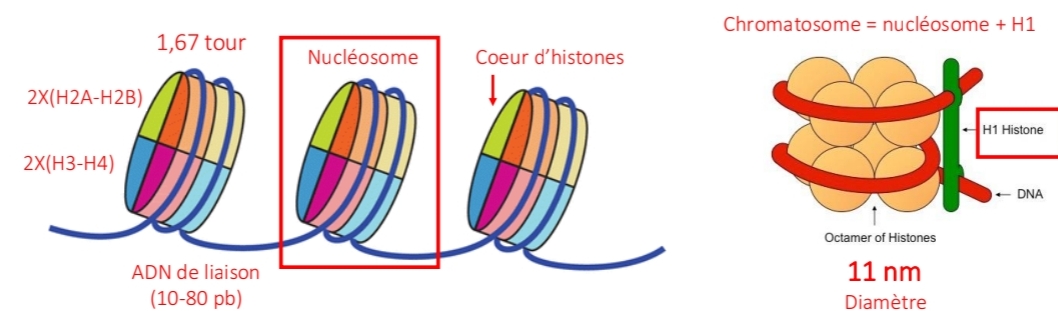
Histones
H2A, H2B, H3, H4
H1 (liaison)
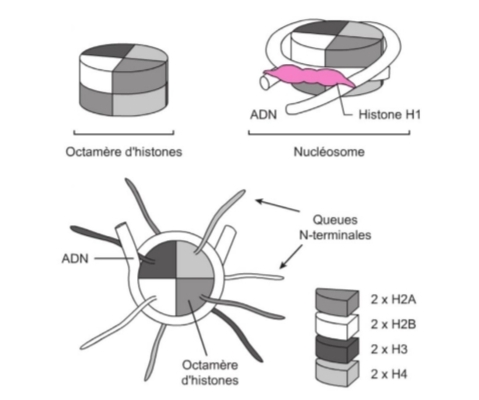
Nucléofilament
Collier de perles
11 nm
Compaction ×6
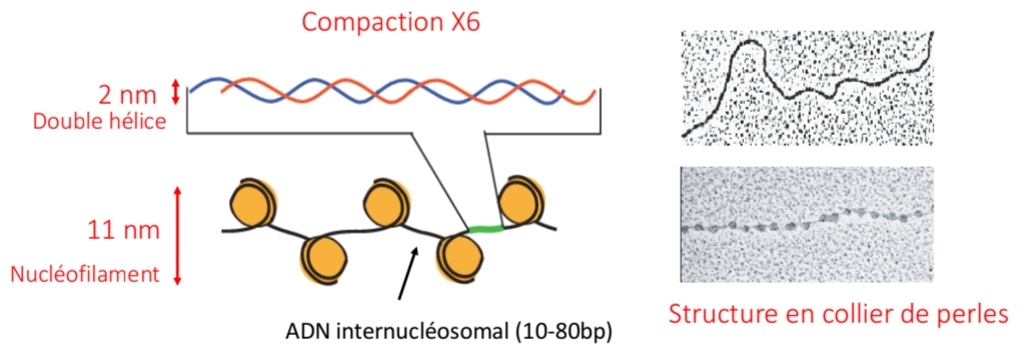
Fibre chromatinienne
30 nm
Solénoïde
Compaction ×40

Boucles chromatine
300 nm
Fixées échafaudage
Compaction ×1000
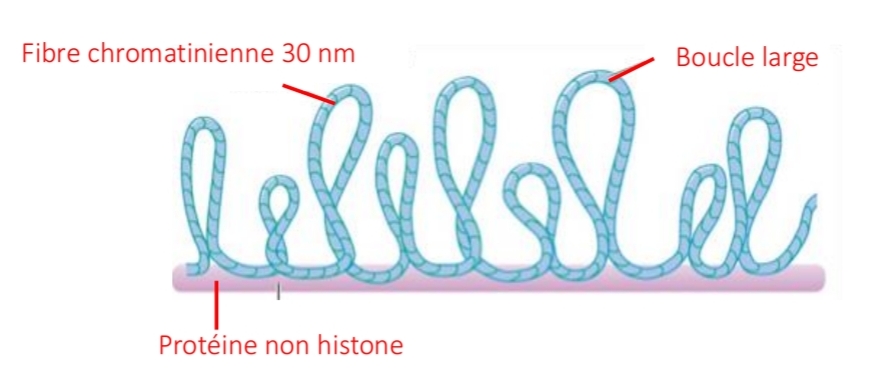
Rosettes chromosomiques
Superenroulement boucles
Chromatide
Compaction ×10 000
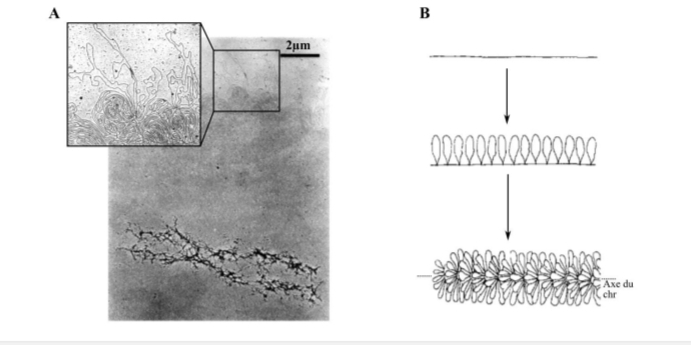
résumer : compation de l’ADN
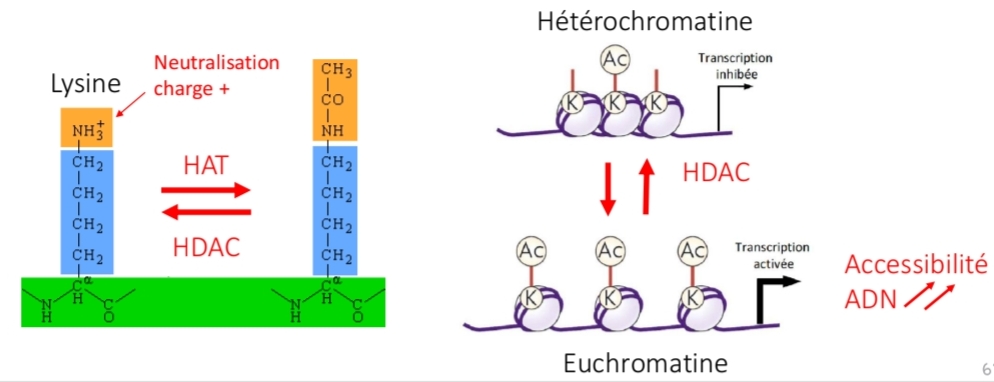
Chromatine : états
Euchromatine → ouverte “nucléofilament”
Hétérochromatine → fermée en périphérie “fibre chromatidienne”
conctitutif : tj inactive
facultatif : active ss condition
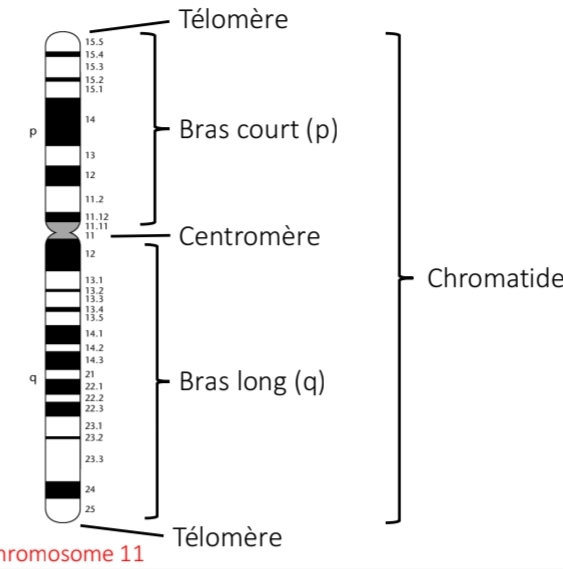
Caryotype + carte génétique + formule chromosomique
Nombre chromosomes
Structure
Métaphase
+
représentation de la disposition des gènes ou des marqueurs sur un chromosome
+
Xp21
chromosome/bras/region/bande
Autosomes / gonosomes
Autosomes → non sexuels
Gonosomes → X / Y
Territoires chromosomiques
Position fixe noyau
interphase/metaphase
Anomalies chromosomiques
nbre/structure des chromosome
interaction ADN-Histone
nn spécifique : de seq ADN avc histone
nn covalente :
→ liaison ionique |e| chargz + (lys/ARG) et - (acide libre de gpm- PO4)
→ lien H ds Nter base azoté (Ser/Thr/Tyr)
nn polaire |e| chaine L histone et désoxyribose
Régulation compaction ADN
organisation chromatine Dynamique : Ftranscription
ouvre fibre chromatidienne + déplace histone
→ modif post-trad des histone modif charge 2ter = structure
→ remodelage chromatine avc déplacement/remplacement histone influence accessibiliter
Réversible fin transcription
Modifications histones
Acétylation
Méthylation
Phosphorylation
Ubiquitination
Acétylation histones
grpm- acétyle sur Nter d’un histone
enz : HAT / HDAC
Neutralise charge + Nter H
→ ↓ interaction
→Accessibilité ADN ↑
✨️ expression génétique
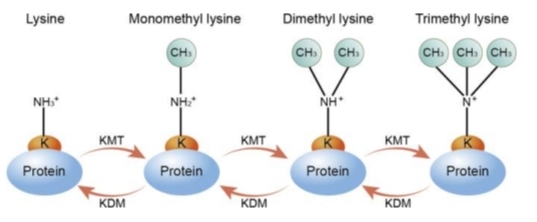
Méthylation histones
1-3 grpm- méhyl sur Lys/Arg de H
enz HMT / HDM
↑V H , charge
Activation ou répression : selon nbre/position
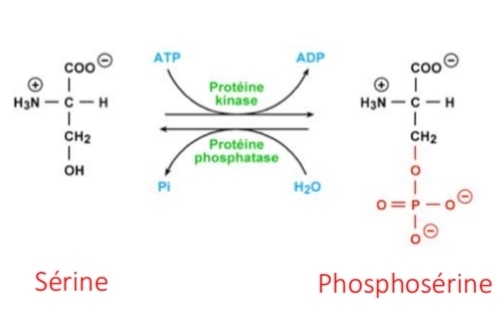
Phosphorylation histones
+PO4 sur Ser/Thr
enz : Kinases + ATP >< phosphatase
ajout charge −
→ Chromatine relâchée
→Accessibilité ADN ↑
Ubiquitination histones
Mono → régulation
Poly → dégradation
lys
dynamique et reversible
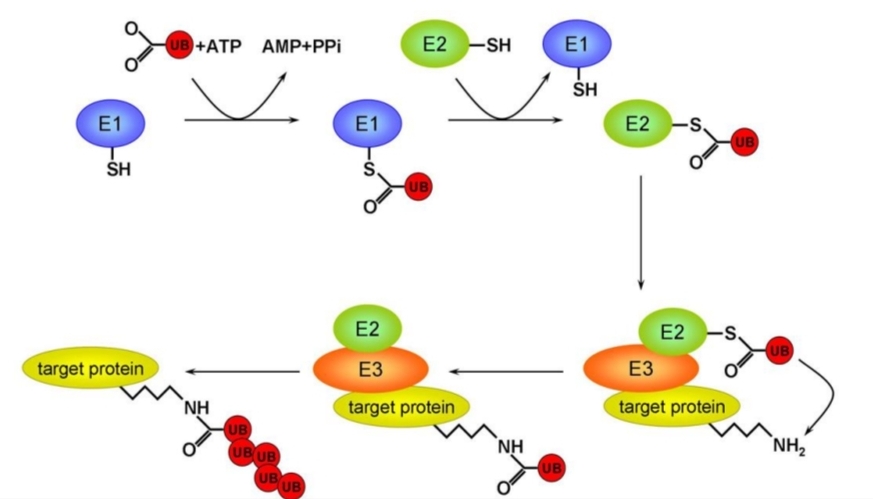
Writers / Erasers / Readers
Les histones portent des marques
✍ writers les posent
🧽 erasers les enlèvent
👀 readers les lisent
Bromodomaines
Chromodomaines
👉 Les readers décident si le gène sera lu ou bloqu
Ajout
Retrait
Lecture marques
Remodelage chromatine
ATP-dépendant
changem- conform nv nucléosome
lie pas directement ADN
Déplacement histones
5 famille
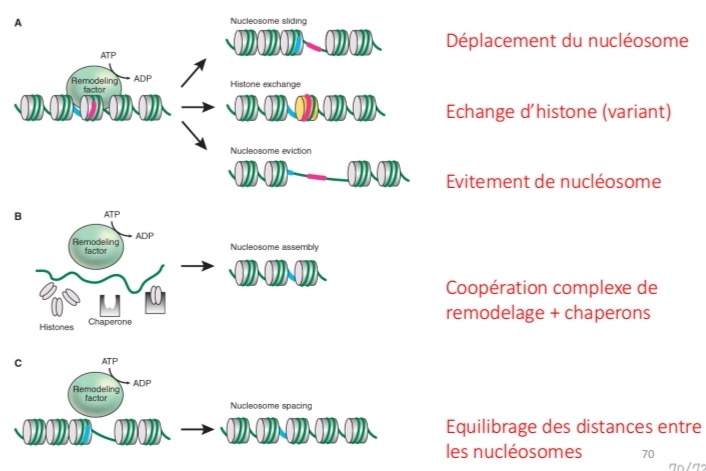
résumer
