GW BGZ 2022 Taak 3 - De perfecte kopie van ons genoom, … of toch niet?
1/33
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No study sessions yet.
34 Terms
Wat is DNA-replicatie en waarom is het essentieel?
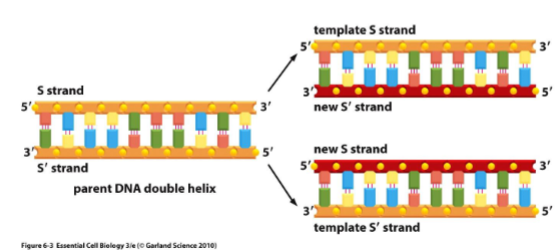
DNA-replicatie is het proces waarbij een cel haar volledige DNA nauwkeurig kopieert vóór mitose of meiose. Dit garandeert dat elke dochtercel een complete set genetische informatie ontvangt.
Het proces is semiconservatief, wat betekent dat elke nieuwe dubbele helix bestaat uit:
één oude (template) streng
één nieuwe streng
Replicatie gebeurt tijdens de S-fase van de celcyclus en verloopt in eukaryoten zeer gecontroleerd om fouten te voorkomen.
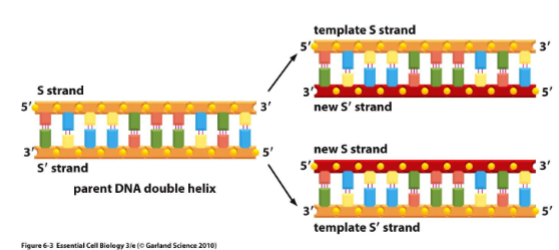
Waarom noemt men DNA-replicatie semiconservatief?
Omdat bij elke replicatie één van de twee oude strengen behouden blijft en als sjabloon dient voor de synthese van een nieuwe complementaire streng.
Zo ontstaan uiteindelijk twee DNA-moleculen:
Oud + Nieuw
Oud + Nieuw
Dit verhoogt de nauwkeurigheid en stabiliteit van genetische overdracht.
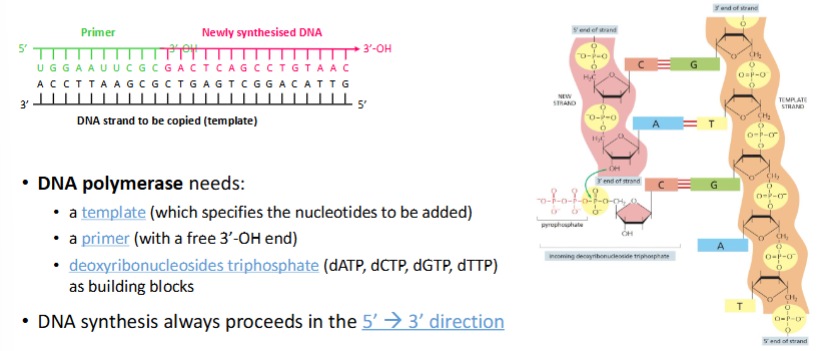
Wat heeft DNA-polymerase nodig om nucleotiden toe te voegen?
DNA-polymerase heeft nodig:
een template (die de toe te voegen nucleotiden specificeert)
een primer (met een vrij 3'-OH-uiteinde)
Complementair met template
Gemaakt van RNA
Nodig omdat DNA-polymerase een begin nodig heeft → kan hij niet uit DNA bestaan
Dan moet een enzym uit het niets beginnen
deoxyribonucleosiden trifosfaat (dATP, dCTP, dGTP, dTTP) als bouwstenen
Geactiveerde vorm van nucleotides
Fosfordiesterbinding, met H en P
Andere twee fosfaten afgesplitst → energie die nodig is om de nucleotide te koppelen
DNA-synthese verloopt altijd in de 5' → 3' richting
Nieuwe nucleotiden altijd aan de 3’ kant toegevoegd
Welke drie hoofdfasen kent DNA-replicatie?
A. Initiatie
Herkenning van de origin of replication (ORI)
Helicase opent het DNA
SSB-eiwitten stabiliseren enkelstrengen
Primase maakt RNA-primers
B. Elongatie
DNA-polymerase III verlengt nieuwe streng in 5’ → 3’
Leading strand: continu
Lagging strand: discontinu via Okazaki-fragmenten
DNA-polymerase I verwijdert primers + vult DNA in
DNA-ligase verbindt fragmenten
C. Terminatie
Replicatie stopt wanneer forks elkaar ontmoeten of het DNA-einde bereiken
Fouten worden gecontroleerd door proofreading
Wat is het verschil tussen leading strand en lagging strand?
Leading strand: continu gesynthetiseerd in 5’ → 3’ richting, slechts één primer nodig.
Lagging strand: discontinu, in Okazaki-fragmenten, meerdere primers nodig.
Het verschil ontstaat doordat DNA-polymerase alleen 5’ → 3’ kan werken terwijl de twee strengen antiparallel zijn.
Lagging strand “loopt achter” bij de voortschrijdende replicatievork en moet later door ligase worden samengevoegd.
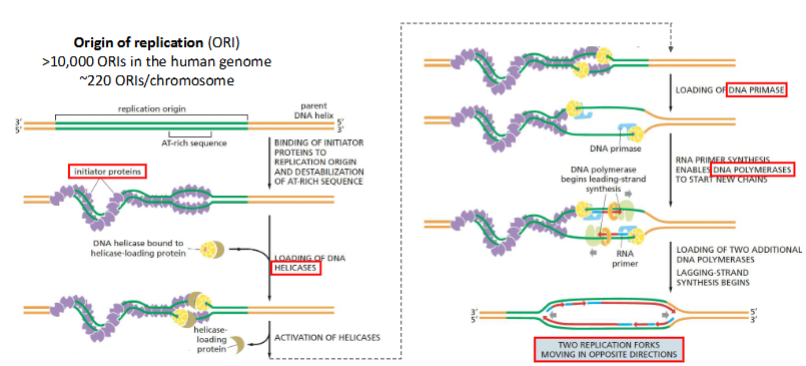
Wat gebeurt er tijdens de opening van de dubbele helix bij DNA-replicatie?
De dubbele DNA-helix is stabiel door waterstofbruggen tussen basenparen.
Replicatie begint op specifieke plaatsen, de replicatieoorsprongen (origins of replication).
Initiator-eiwitten herkennen deze oorsprongen en binden hierop, waarna ze lokale waterstofbruggen breken om de strengen uit elkaar te trekken.
A-T-rijke regio’s worden vaak gebruikt omdat A-T-paren slechts twee waterstofbruggen hebben, terwijl G-C-paren drie hebben.
Bij elke oorsprong ontstaan twee replicatievorken die in tegengestelde richtingen bewegen → bidirectionele replicatie.
Deze vorken zijn Y-vormig en zorgen ervoor dat de strengen beschikbaar komen als sjabloon voor nieuwe DNA-strengen.
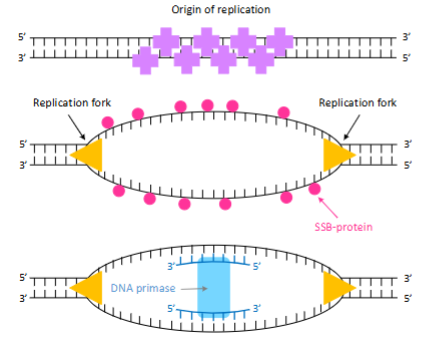
Waarom zijn RNA-primers nodig en hoe worden ze gemaakt?
DNA-polymerase kan geen nieuwe streng starten, het kan alleen nucleotiden toevoegen aan een bestaand 3’-OH einde.
Daarom maakt primase, een speciaal RNA-polymerase, een kort RNA-primer van ongeveer 10 nucleotiden als startpunt.
De primer baseert zich op de complementaire DNA-streng.
Leading strand: slechts één primer nodig aan het begin.
Lagging strand: meerdere primers nodig voor elk Okazaki-fragment, omdat DNA in stukjes wordt gesynthetiseerd.
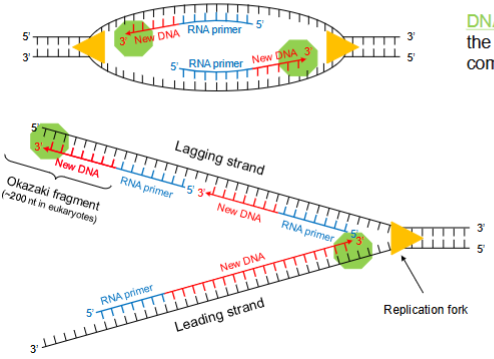
Hoe verloopt de DNA-synthese vanaf een primer?
DNA-polymerase III voegt nucleotiden toe aan het 3’-uiteinde van de primer.
De nieuwe streng wordt complementair aan de template streng.
DNA-polymerase werkt alleen 5’ → 3’, waardoor de synthese op de strengen verschillend verloopt:
Leading strand: continu synthese.
Lagging strand: discontinu synthese, in Okazaki-fragmenten.
Energie: nucleotiden zijn dNTP’s met drie fosfaatgroepen; bij binding komt energie vrij voor de fosfodi-esterbinding.
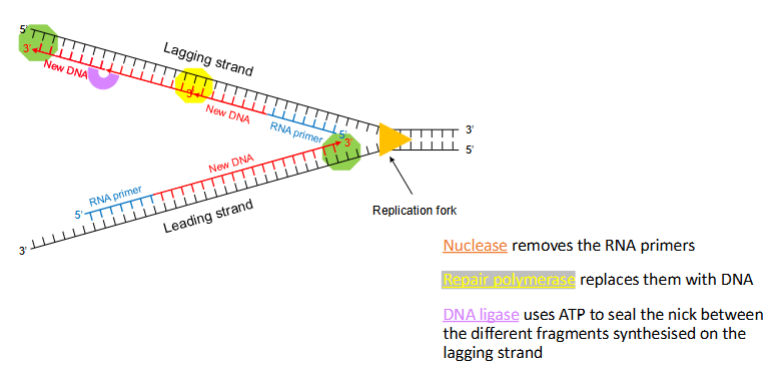
Wat gebeurt er met de RNA-primers na synthese van DNA?
DNA-polymerase I verwijdert RNA-primers via 5’ → 3’ exonucleaseactiviteit.
Het enzym vult de vrijgekomen gaten op met DNA.
Zo wordt het DNA volledig continu gemaakt, zonder RNA-stukken.
Dit is cruciaal om stabiel DNA te verkrijgen dat geen RNA bevat in het uiteindelijke molecuul.
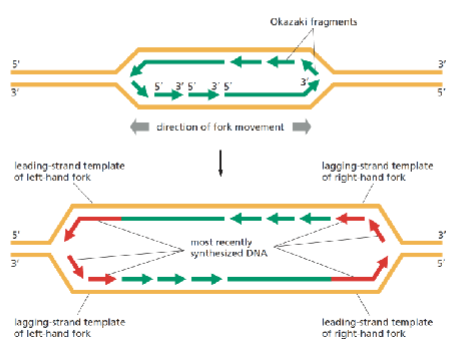
Hoe worden Okazaki-fragmenten samengevoegd tot één DNA-streng?
Na vervanging van de RNA-primers blijven korte DNA-fragmenten (Okazaki-fragmenten) over.
DNA-ligase vormt fosfodi-esterbindingen tussen de fragmenten.
Resultaat: een continue, ononderbroken DNA-streng.
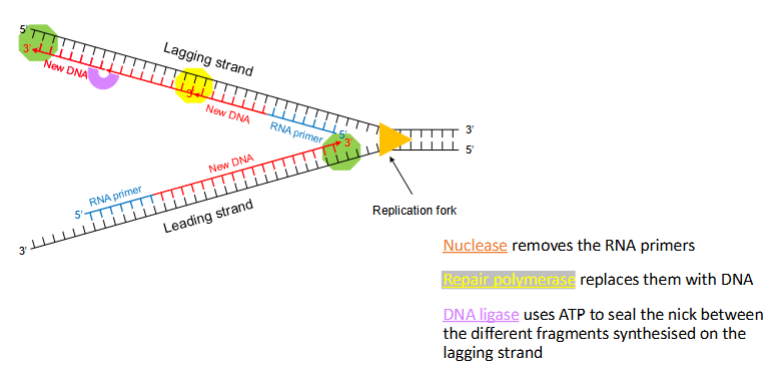
Welke enzymen spelen een rol in DNA-replicatie en wat is hun functie?
Enzym | Functie |
|---|---|
Initiator-eiwit | Herkent replicatie-oorsprong en opent DNA |
Helicase | Scheidt DNA-strengen door waterstofbruggen te verbreken |
Primase | Maakt RNA-primers als startpunt voor DNA-synthese |
DNA-pol III | Verlenging van DNA-streng vanaf primer (5’ → 3’) |
DNA-pol I | Verwijdert RNA-primers en vervangt ze door DNA |
Ligase | Verbindt Okazaki-fragmenten tot continue streng |
DNA-pol II | Herstel DNA-schade en vullen van gaten (niet betrokken bij replicatie) |
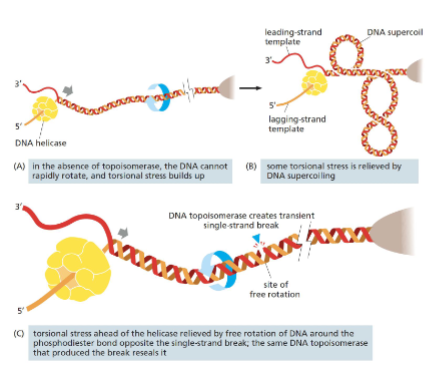
Wat is het probleem van supercoiling tijdens DNA-replicatie en hoe wordt dit opgelost?
Tijdens het openen van de dubbele helix door helicase ontstaat torsiespanning in het DNA vóór de replicatievork.
Dit veroorzaakt supercoiling, waardoor helicase niet verder kan bewegen en replicatie zou stoppen.
Oplossing: DNA-topoisomerasen.
Topoisomerase I:
Maakt een enkelstrengsbreuk zodat de streng kan draaien en spanning vermindert.
Herstelt de breuk automatisch, ATP niet nodig.
Topoisomerase II:
Maakt een dubbele strengbreuk om verstrengelde DNA-moleculen door elkaar te laten glijden.
Vereist ATP, omdat het beide strengen moet openen en sluiten.
Functie: voorkomt blokkades bij replicatie en ontward DNA bij celdeling.
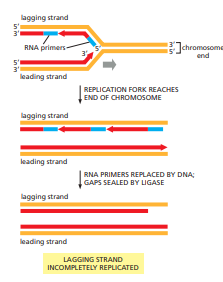
Wat is het “end-replication problem” bij lineaire chromosomen?
DNA-polymerase kan DNA alleen 5’ → 3’ verlengen en heeft een primer nodig om te starten.
Op de lagging strand is het laatste RNA-primer aan het uiteinde niet vervangbaar door DNA, omdat er geen vrij 3’-OH einde is.
Resultaat: elk chromosoom wordt bij replicatie korter.
Bacteriën hebben dit probleem niet (cirkulair DNA), maar eukaryoten wel (lineaire chromosomen).
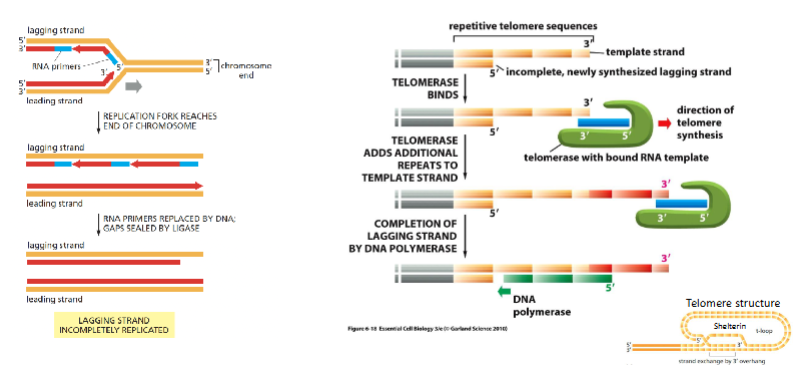
Hoe voorkomt telomerase verlies van DNA bij chromosoomuiteinden?
Telomerase is een enzym met een eigen RNA-sjabloon.
Het voegt herhaalde DNA-sequenties (telomeren) toe aan het 3’-uiteinde van het DNA (bijv. TTAGGG bij mensen).
Hierdoor kan DNA-polymerase later de complementaire streng kopiëren.
Het resultaat: chromosomen behouden hun lengte en genetische informatie gaat niet verloren.
Functies van telomerase:
Verlengt telomeren.
Beschermt chromosoomuiteinden tegen herkenning als DNA-breuken.
Voorkomt verlies van genetische informatie bij celdeling.
In welke cellen is telomerase actief en waarom is dit belangrijk?
Cellen met actieve telomerase | Waarom actief? |
|---|---|
Kiemcellen | Voor voortplanting, behouden chromosoomlengte |
Stamcellen | Voor continue deling en weefselregeneratie |
Kankercellen | Vaak heractivering → onbeperkte deling mogelijk |
Somatische lichaamscellen: lage/geen telomerase → telomeren verkorten bij elke deling → draagt bij aan celveroudering.
Kankercellen: abnormale telomerase-activiteit voorkomt telomeerverkorting → draagt bij aan immortalisatie van de cel.
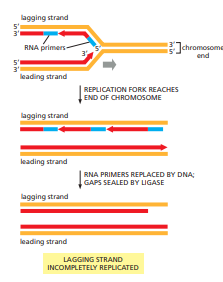
Waarom ontstaan Okazaki-fragmenten en wat gebeurt ermee?
Op de lagging strand, die antiparallel loopt, kan DNA alleen in korte stukjes worden gesynthetiseerd in de juiste 5’ → 3’ richting.
Daarom maakt DNA-pol III telkens korte DNA-fragmenten, elk voorafgegaan door een primer.
Vervolgens:
DNA-pol I verwijdert RNA-primer (5’→3’ exonuclease)
Vult aan met DNA
DNA-ligase verbindt de fragmenten tot één doorlopende streng
Wat zijn de functies van DNA-polymerase I, II en III?
DNA-pol I
Verwijdert RNA-primers (5’→3’ exonuclease)
Vult aan met DNA
Reparatie van kleine beschadigingen
DNA-pol II
Reparatie-enzym
Proofreading (3’→5’ exonuclease)
DNA-pol III
Hoofd-replicatie-polymerase
Hoge snelheid en procesiviteit
Proofreading (3’→5’ exonuclease)
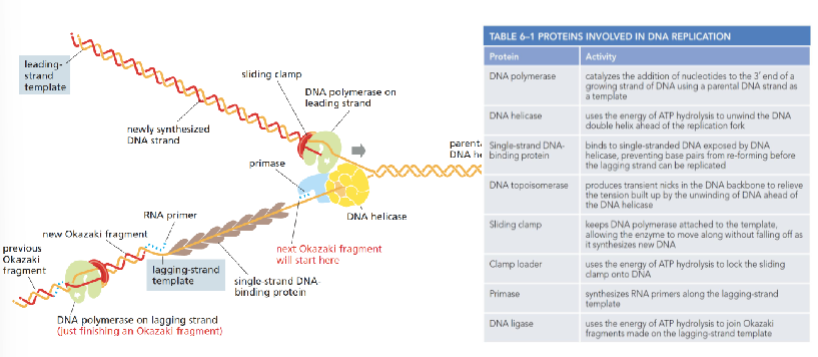
Welke eiwitten werken samen bij het replicatiemechanisme?
DNA-polymerase
katalyseert de toevoeging van nucleotiden aan het 3'-uiteinde van een groeiende DNA-streng met behulp van een ouderlijke DNA-streng als template
DNA-helicase
gebruikt de energie van ATP-hydrolyse om de dubbele DNA-helix vóór de replicatievork af te wikkelen
Enkelstrengs DNA-bindend eiwit
bindt aan enkelstrengs DNA dat is blootgesteld door DNA-helicase, waardoor wordt voorkomen dat basenparen zich opnieuw vormen voordat de achterblijvende streng kan worden gerepliceerd
DNA-topoisomerase
produceert tijdelijke inkepingen in de DNA-ruggengraat om de spanning te verlichten die is opgebouwd door het afwikkelen van DNA vóór de DNA-helicase
Glijdende klem
houdt DNA-polymerase aan de template vast, waardoor het enzym kan bewegen zonder eraf te vallen terwijl het nieuw DNA synthetiseert
Klemlader
gebruikt de energie van ATP-hydrolyse om de glijdende klem op DNA te vergrendelen
Primase
synthetiseert RNA-primers langs de achterblijvende-streng template
DNA-ligase
gebruikt de energie van ATP-hydrolyse om Okazaki-fragmenten te verbinden die op de achterblijvende-streng template zijn gemaakt
Welke stappen vervangen RNA-primers door DNA?
DNA-pol I verwijdert RNA-primer (5’→3’ exonuclease)
DNA-pol I vult het ontstane gat op met DNA
DNA-ligase sluit de laatste fosfodiesterbinding
Welke herstelmechanismen zijn er tijdens de replicatie?
Monitoring (selectie van juiste base)
Proofreading (3’→5’ exonuclease)
Single-Strand Repair (SSR)
Mismatch repair (MMR)
Exision Repair (NER en BER)
Double-Strand Break Repair (NHEJ en HR)
Wat is de controle van basenparing (monitoring)?
Tijdens het toevoegen van een nieuw nucleotide:
Controleert DNA-polymerase of de inkomende nucleotide correct past bij de sjabloonstreng.
Alleen bij een juiste match verandert het enzym van vorm en maakt het de binding (vorming van een fosfodiësterbinding).
Hierdoor worden verkeerde koppelingen (zoals G–T of C–A) meestal direct voorkomen.
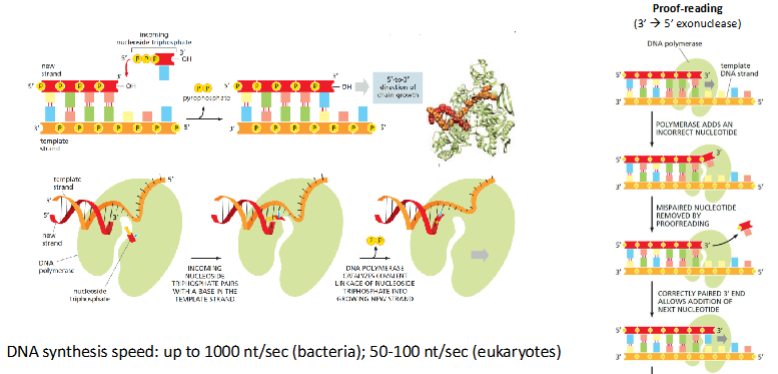
Wat is proofreading (monitoring)?
Als er tóch een fout wordt gemaakt, kan DNA-polymerase dit onmiddellijk herstellen.
Van 3 → 5’
Het enzym controleert het laatst toegevoegde nucleotide voordat het verdergaat:
Als het goed gepaard is → gaat de synthese verder.
Als het fout is → knipt het enzym de verkeerde base weg met een aparte exonuclease-activiteit en probeert opnieuw.
De polymerisatie en proofreading gebeuren tegelijk binnen hetzelfde enzym, maar in verschillende domeinen van het molecuul.
Wat is het basismechanisme van single-strand DNA-reparatie?
DNA-schade beperkt tot één streng kan worden hersteld met de onbeschadigde streng als sjabloon.
Stappen:
Herkenning en verwijderen van beschadigde nucleotiden
Nucleases knippen de beschadigde nucleotide uit.
Er ontstaat een gap in de streng.
Vullen van de gap
Repair DNA-polymerase bindt aan het 3’-einde en synthetiseert nieuwe nucleotiden complementair aan de onbeschadigde streng (5’→3’ richting).
Sluiten van de nick
DNA-ligase sluit het suiker-fosfaat-achtervlak.
Voorbeelden van schade: depurinatie, deaminatie (C→U), oxidatie of alkylatie van basen.
Wat is mismatch repair (MMR) en waarom is het belangrijk?
Herstelt fouten die tijdens replicatie zijn gemaakt en proofreading hebben overleefd.
Herkenning: mispaired nucleotiden (bv. G-T, A-C).
Proces: stukje van de nieuwgesynthetiseerde streng wordt verwijderd en correct aangevuld door DNA-polymerase.
Resultaat: foutfrequentie verlaagd tot ~1 op 10⁹ nucleotiden.
Belang voor de mens: mutaties in MMR-genen verhogen risico op kanker, vooral colonkanker.
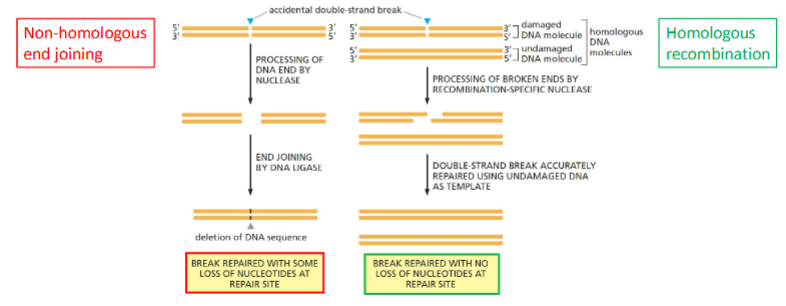
Welke twee hoofdstrategieën bestaan er voor double-strand break repair (DSB)?
Non-Homologous End Joining (NHEJ)
Snel, “noodreparatie”.
Breuken direct aan elkaar gelijmd → kans op verlies van nucleotiden/mutaties.
Vooral actief in niet-delende cellen of G1-fase.
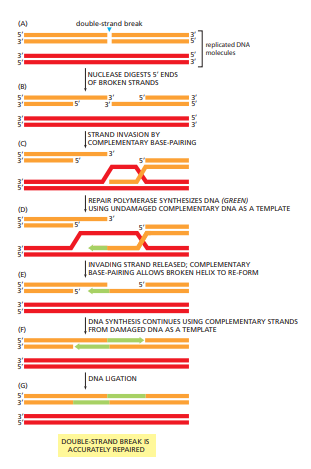
Homologous Recombination (HR)
Nauwkeuriger, foutloos.
Gebruikt de zusterchromatide als sjabloon.
Alleen mogelijk in S- en G2-fase.
Specialistische enzymen herkennen de breuk, vinden de zusterchromatide en bouwen de juiste nucleotiden in.
Gevaar: dubbelstrengsbreuken kunnen leiden tot chromosoomfragmentatie, verlies van genen, celsterfte of kanker.
Wat is excisie-herstel (excision repair) en welke stappen omvat het?
Overkoepelende term voor herstelmechanismen waarbij beschadigde nucleotiden worden herkend en verwijderd.
Stappen:
Herkenning van beschadigde nucleotiden
Excisie van het beschadigde deel
Vullen van het gat door DNA-polymerase
Sluiten van de streng met DNA-ligase
Onderverdelingen: Nucleotide Excision Repair (NER) en Base Excision Repair (BER).
Hoe werkt Nucleotide Excision Repair (NER) en Base Excision Repair (BER)?
Nucleotide Excision Repair (NER)
Herstelt grote, helix-vervormende schade: thymine-dimeren, bulky adducten.
Werkwijze: herkenning → helicase opent DNA → excisie van 24–32 nt → DNA-polymerase vult gat → DNA-ligase sluit.
Subtypes:
GG-NER: global genomic, overal in DNA
TC-NER: transcription-coupled, snelle reparatie op actieve genen
Base Excision Repair (BER)
Herstelt kleine, niet-helixvervormende schade: geoxideerde basen, gedeamineerde basen, alkylatie.
Werkwijze: DNA-glycosylase verwijdert base → AP-endonuclease knipt backbone → DNA-polymerase vult → DNA-ligase sluit.
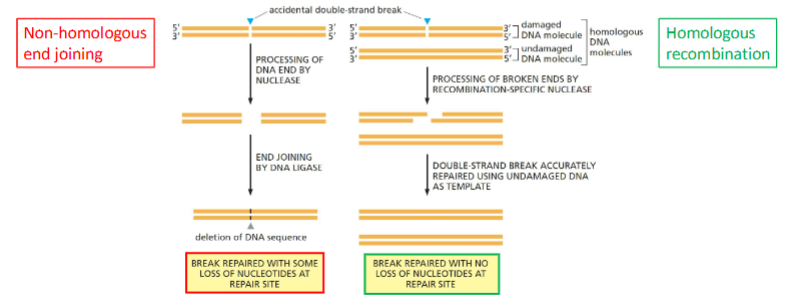
Welke twee typen Double-Strand Break Repair zijn er wat doen ze?
Herstelt breuken in beide strengen van het DNA.
Non-Homologous End Joining (NHEJ)
Snel, noodreparatie; breuken direct aan elkaar gelijmd
Risico op verlies van nucleotiden → mutaties mogelijk
Vooral in niet-delende cellen of G1-fase
Homologous Recombination (HR)
Foutloos herstel met zusterchromatide als sjabloon
Alleen tijdens S- en G2-fase
Herstel van de juiste sequentie met gespecialiseerde enzymen
Welke soorten mutaties kunnen tijdens DNA-replicatie optreden?
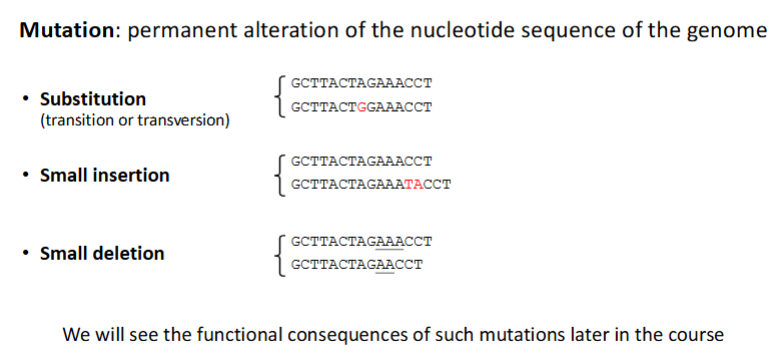
Puntmutaties – verandering van één nucleotide:
Substitutie: één base vervangen door een andere.
Silent: aminozuur blijft hetzelfde.
Missense: aminozuur verandert → eiwit kan veranderen.
Nonsense: stopcodon → eiwit voortijdig afgebroken.
Insertie: 1 of meer nucleotiden toegevoegd.
Deletie: 1 of meer nucleotiden verwijderd.
Frameshift-mutaties – insertie of deletie niet deelbaar door 3 nucleotiden.
Verandert het leesraam → vaak zwaar effect op eiwit.
Non-frameshift mutaties – insertie of deletie veelvoud van 3 nucleotiden.
Leesraam blijft intact → vaak milder effect.
Grote structurele mutaties – veranderingen in chromosoomdelen:
Duplicatie: extra genkopieën.
Deletie: verlies van genen.
Inversie: DNA-fragment omgekeerd.
Translocatie: DNA-fragment naar ander chromosoom.
Crossing-over (meiose): genetische uitwisseling.
Waardoor ontstaan DNA-mutaties?
Spontaan: fouten bij DNA-replicatie, depurinatie (verlies purine), deaminatie (C → U)
Externe factoren (mutagenen):
UV-straling → thymine-dimeren
Chemische stoffen → basemodificaties
Reactieve zuurstofmoleculen (ROS) → oxidatieve schade
Mechanische fouten: slippage van polymerase bij repeterende sequenties → kleine indels
Wat kunnen de effecten van DNA-mutaties zijn?
Neutraal: geen effect
Voordeel: evolutie/aanpassing (bijv. resistentie tegen ziekte)
Nadelig: ziekte of kanker (bv. cystic fibrosis, sikkelcelanemie)
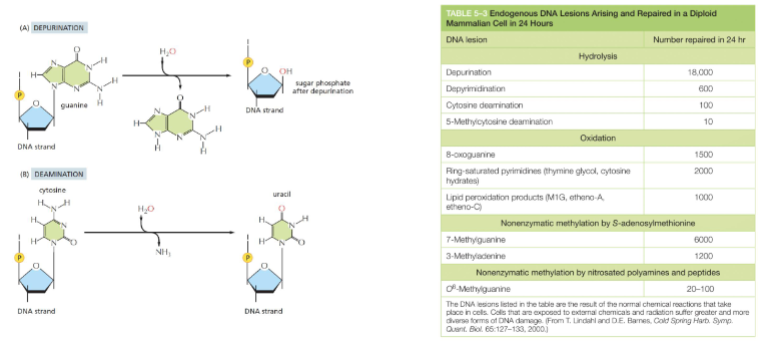
Wat gebeurt er bij depurinatie?
Wat gebeurt er:
Een purinebase (adenine (A) of guanine (G)) wordt spontaan verwijderd van de suiker in het DNA.
De N-glycosidische binding tussen de base en de suiker wordt verbroken.
Hierdoor blijft er een lege plek (een zogeheten apurinic site of AP-site) achter in de DNA-streng.
Gevolg:
Tijdens DNA-replicatie weet het polymerase niet welke base er moet worden ingebouwd tegenover die lege plek.
Dit kan leiden tot verkeerde base-inbouw → puntmutatie of deletie.
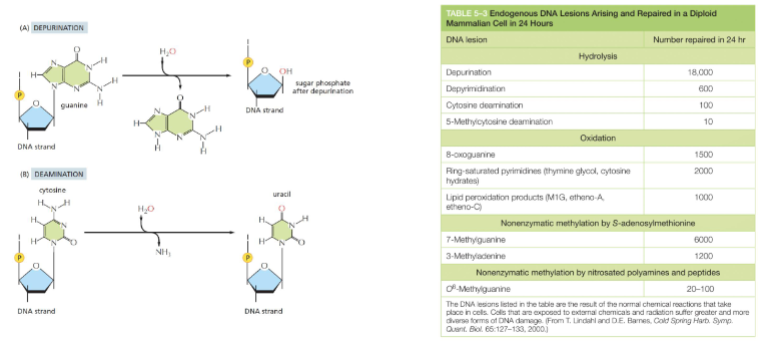
Wat gebeurt er bij deaminatie?
Wat gebeurt er:
Een base verliest een aminogroep (-NH₂).
Vooral cytosine (C) wordt hierdoor omgezet in uracil (U).
Omdat uracil normaal niet in DNA voorkomt, vormt dit een duidelijke afwijking.
Wat is het gevolg:
Tijdens replicatie herkent DNA-polymerase uracil als thymine (T) en koppelt er een adenine (A) tegenover.
De oorspronkelijke C–G base wordt dan vervangen door een T–A paar → puntmutatie (substitutie).
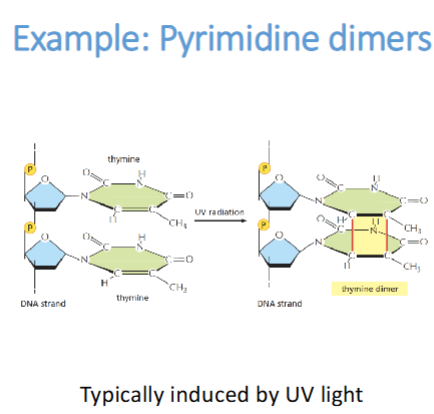
Wat gebeurt er bij thymine-dimeren
Wat gebeurt er:
UV-licht (zoals van zonlicht) veroorzaakt dat twee naburige thymine-basen (T-T) op dezelfde DNA-streng chemisch aan elkaar binden.
Dit vormt een covalente binding → een thymine-dimeer.
De structuur van het DNA vervormt en kan niet goed meer worden afgelezen.
Gevolg:
DNA-replicatie en transcriptie stokken op die plek.
Als het niet wordt