BC Proteine
1/27
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No study sessions yet.
28 Terms
Hydrophober Kollaps Modell
Frühe Zusammenlagerung hydrophober Reste
Schnelle Kompaktierung („molten globule“)
Hydrophober Effekt als Haupttriebkraft
Vorteil: erklärt schnelle Anfangsphasen der Faltung
Nachteil: erklärt nicht, wie spezifische Sekundärstrukturen entstehen
Was ist ein Westernblot und wieso werden 2 verschiedene Antikörper zur Detektion verwendet ?
es ist eine Methode mit der spezifische Proteine einer komplexen Probe nachgewiesen wird.
das verfahren kombiniert die Trennung von Proteinen nach ihrer Größe mittels gelelektrophorese unter Verwendung von Antikörper
Schritte nach SDS Page
Proteintransfer : Übertragung der Proteine aus dem Gel auf einen Membran
Blockierung: unspezifische bindungsstellen der Membran werden blockiert
Primärantikörper Inkubation: bindet spezifisch an das Zeilprotein
Waschen: entfernt ungebundenes Antikörper
Sekundärantikörper: bindet an den primärantikörper
Detektion: enzymatisch oder Flourszent
Warum 2?
primärantikörper erkennt spezifisches Zielprotein
Sekundärprotein: Sgnalverstärkung
Universell einsetzbar für viele primärantikörper
Vergleichen Sie energieidagramme einer unakatalysierten, einem katalysierten ( komplementär zum Übergangszustand und einer die komplementär zum Substrat ist
Unkatalysiert:
hohe aktivierungsnergie
Langsame Reaktion
Enzym katalysiert:
Stärkste Absenkung der AE
Übergangszustand wird stabilisiert
Schnellste Reaktion
Enzym komplementär
Übergangszustand cheered erreichbar
Stabilisiert das Substrat zu stark
Geringe Katalyse
Enzyme sind komplementär zum Übergangszustand nicht zum Substrat
Welche Aussage zu enzymatisch katalysierten Reaktionen ist nicht korrekt?
a. Enzyme verringern die Aktivierungsenergie einer Reaktion
b. Enzyme führen sterische Spannung in ein Substrat ein
c. Enzyme stellen eine Umgebung her, wodurch Reaktionen schneller ablaufen
d. Enzyme verschieben das Gleichgewicht zur Edukt-Seite
e. Mit Papain können Antikörper in drei Fragmente getrennt werden
D
Was sind Ribozyme und Abzyme und wie unterscheiden sich diese von anderen Enzymen? Nennen Sie
je zwei Beispiele.
Ribozyme
Enzyme aus RNA
Katalytisch aktive RNA-Moleküle
Beispiele:
Ribosom (Peptidyltransferase-Zentrum)
RNase P
Abzyme
Katalytisch aktive Antikörper
Erkennen den Übergangszustand
Beispiele:
Esterase-Abzyme
Protease-Abzyme
Unterschied zu klassischen Enzymen:
Nicht proteinbasiert (Ribozym)
Künstlich erzeugt (Abzym)
Nennen Sie vier Mechanismen der Enzymkatalyse mit je einem Beispiel.
Säure-Base-Katalyse
→ Histidin in ChymotrypsinKovalente Katalyse
→ Serin in SerinproteasenMetallionen-Katalyse
→ Zn²⁺ in CarboanhydraseNähe & Orientierung
→ Hexokinase bindet Glucose und ATP optimal
Welche Aussage ist nicht korrekt?
a. Metallionen können in Enzymen als Cofaktoren dienen.
b. Die Einschränkung der Mobilität/Flexibilität der Reaktanden und ihre korrekte Orientierung
durch Enzyme hat keinen Einfluss auf die Reaktionsrate.
c. Ein Histidin-Rest kann als allgemeine Base bei der Enzymkatalyse wirken.
d. Ein Histidin-Rest kann als allgemeine Säure bei der Enzymkatalyse dienen.
e. Das Substrat wird während der Katalyse im aktiven Zentrum des Enzyms gebunden.
B
Die nachfolgende Proteinsequenz wird mit den Proteasen Chymotrypsin und Trypsin behandelt.
Welche Peptidsequenzen sind nach dem Verdau zu erwarten? Erklären Sie, warum die Proteasen
unterschiedlich wirken.
Trypsin: spaltet nach Lys (K) und Arg (R)
Chymotrypsin: spaltet nach aromatischen AS
Phenylalanin (F)
Tyrosin (Y)
Tryptophan (W)
Erwartete Peptide
Trypsin: Schnitte hinter K/R → viele kurze Peptide
Chymotrypsin: Schnitte hinter F/Y/W → andere Fragmentlängen
Warum wirken sie unterschiedlich?
Unterschiedliche Substratbindungstaschen
Trypsin: negativ geladen → bindet K/R
Chymotrypsin: hydrophob → bindet aromatische Seitenketten
Bei Chymotrypsin handelt es sich um eine Serin-Protease, was bedeutet dies? Erklären Sie die katalytische
Triade
Serin-Protease bedeutet:
Serin ist das nukleophile Zentrum der Katalyse
Katalytische Triade
Ser195: Nukleophiler Angriff
His57: Protonenübertragung (Säure/Base)
Asp102: Stabilisiert Histidin
Funktion:
Aktiviert Serin
Ermöglicht Peptidspaltung
Stabilisiert Übergangszustand
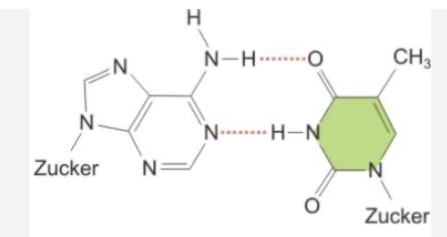
Welche Nukleoside sind das ?
Adenin und Thymin
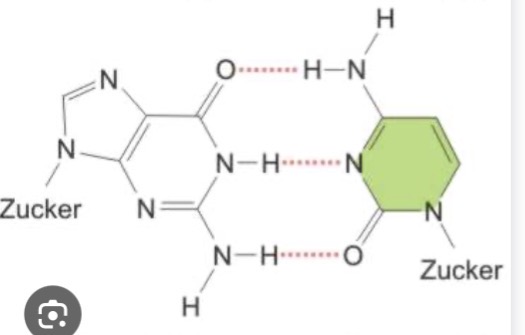
Welche nukleoside sind das ?
Guanin und cytosin
Nukleosom
grundlegende Struktureinheit des Chromatins in eukaryotischen Zellen, bestehend aus einem etwa 147 Basenpaare langen DNA-Abschnitt, der sich ungefähr 1,65-mal um einen Proteinkern aus acht Histonen (dem Histonoctamer) wickelt, ähnlich einer Perlenkette, was die DNA-Verpackung im Zellkern ermöglicht und reguliert.
Nukleus
Zellkern
Nucleolus
dichte Struktur innerhalb des Zellkerns eukaryotischer Zellen, die hauptsächlich für die Synthese und den Zusammenbau von Ribosomenvorstufen (rRNA und ribosomale Proteine) verantwortlich ist
Ligand
Moleküle, Ionen oder Atome, die sich an ein zentrales Atom (in der Chemie) oder ein Zielprotein (in der Biochemie, z. B. einen Rezeptor) anlagern, um Komplexe oder spezifische Bindungen zu bilden
Welche Aussage ist nicht korrekt?
Das Induced Fit-Modell beschreibt die konformationelle Änderung eines Proteins nach
LigandenbindungBei homotropen Interaktionen findet eine wechselseitige Beeinflussung zwischen gleichen
Liganden stattBei heterotropen Interaktionen findet eine wechselseitige Beeinflussung zwischen unterschiedlichen Liganden statt
Allosterische Proteine besitzen nach einer Ligandenbindung veränderliche Affinitäten zu einem weiteren Liganden
Allosterische Proteine weisen nie eine sigmoidale Sättigungskurve auf
5) es ist typisch
P ist ein monomeres Protein mit einer Bindungsstelle pro Molekül für den Liganden L.
Es bindet den Liganden L mit einer Assoziationskonstante Ka von 106 M-1. Skizzieren Sie die Bindungskurve für die Bindung des Liganden L an das Protein P in einem Diagramm, in dem sie den Sättigungsgrad & gegen steigende Konzentration des Liganden L auftragen. Die Bindungskurve soll die Affinität zwischen Protein P und Ligand L korrekt widerspiegeln.
Steiler Anstieg bei niedriger L
Halbsättigung bei KD 1/ K a = 10^-6 M
Hohe Affinität = frühe Sättigung
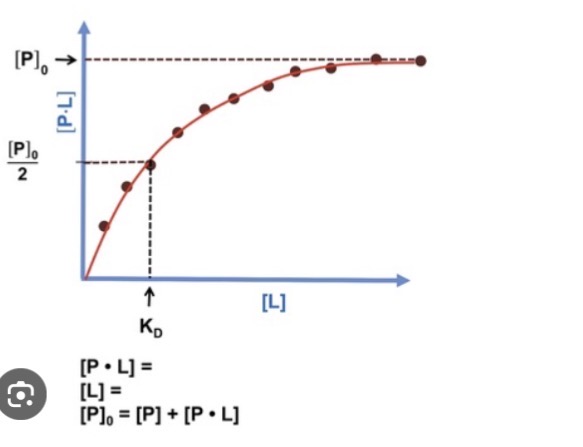
Erklären Sie den Begriff der Kooperativität und erläutern Sie, welche Parameter die Struktur des Hämoglobins und damit seine Affinität beeinflussen.
Kooperativität
Definition
Bindung eines Liganden beeinflusst die Affinität weiterer Bindungsstellen
Hämoglobin
Tetramer (α₂β₂)
Positive Kooperativität
S-förmige (sigmoidale) Bindungskurve
T- und R-Form
T-Form: niedrige Affinität
R-Form: hohe Affinität
Affinitätsmodulatoren
pH (Bohr-Effekt)
CO₂
2,3-BPG
O₂-Partialdruck
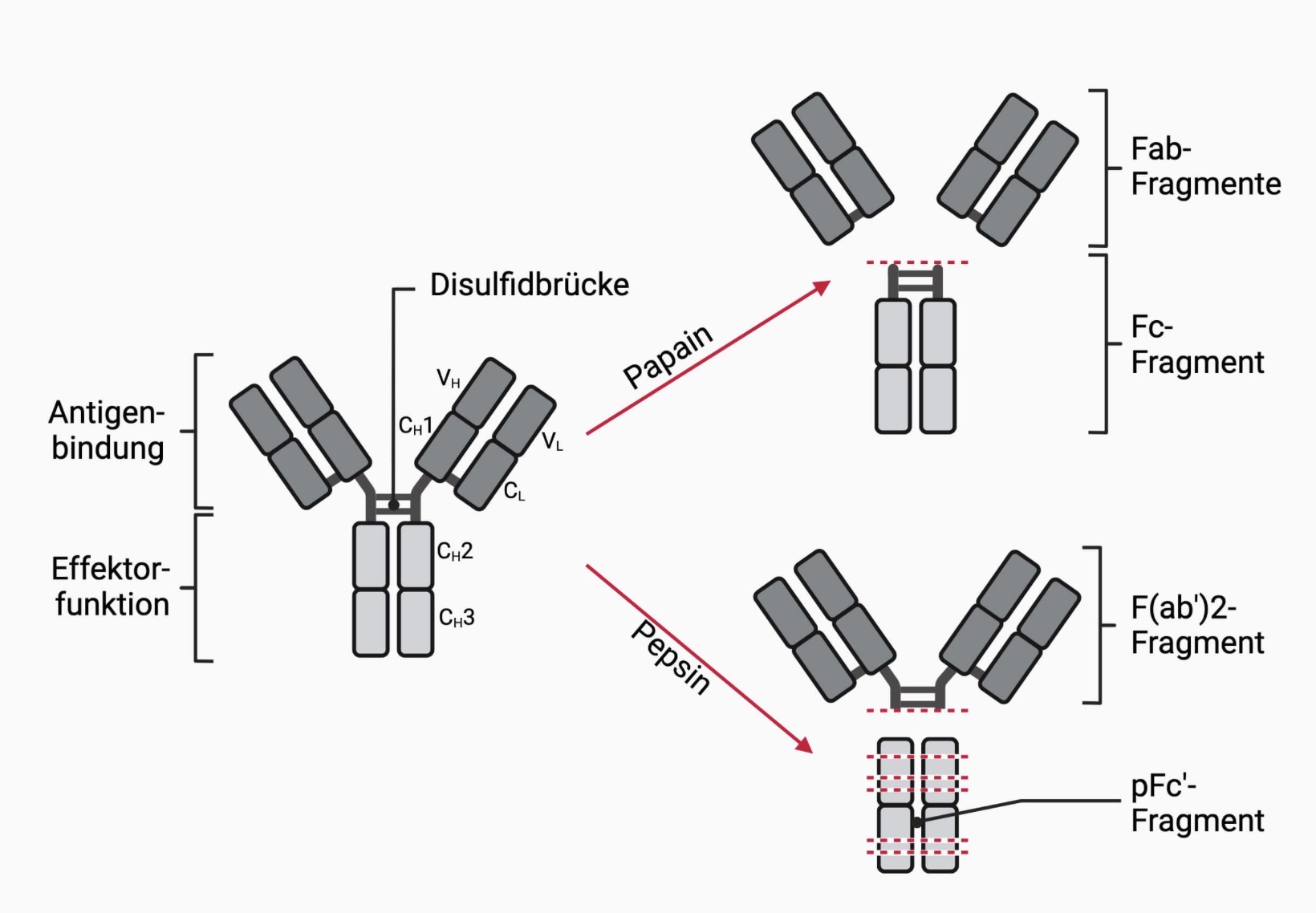
Skizzieren Sie Immunglobulin G. Ordnen Sie folgende Bestandteile zu: CH, CL, VH, VL, N-/C-Terminus, antigen-bindendes Fragment, kristallisierbares Fragment, Antigen-Bindestelle. Mit welchem Enzym lassen sich gezielt beide Fragmente trennen?
Ein Meilenstein in der Proteinforschung ist Affinsens Experiment zur Renaturierung von Proteinen.
Welche der folgenden Aussagen ist nicht korrekt?
Harnstoff und Mercaptoethanol zerstören nicht kovalente Wechselwirkungen.
Die denaturierte und reduzierte Form der Ribonuklease A zeigt keine Aktivität.
Die 3D-Struktur ist durch die Sequenz bestimmt und wird für die Proteinaktivität benötigt.
Katalytische Mengen von Mercaptoethanol sind von Vorteil, um die desaktivierte Ribonuklease A wieder in ihre native Form zu überführen.
Die native Form des Enzyms ist die Form der katalytischen Aktivität.
4) Mercaptiethanol muss vollständig entfernt werden, sonst werden disulfidbrücken nicht korrekt gebildet
Gro EL und Gro ES
Falsch gespaltetes Substratprotein bindet an die hydrophoben Bereiche in der inneren Oberfläche der offenen Gro EL zusammen mit ATP
GroES Klappe schließt und bildet ein Käfig
Im Käfig wird die Umgebung Hydrophil, was das Protein zum korrekten Falten bringt
Die hydrolyse von ATP führt zum Abbau des GRO ES EL
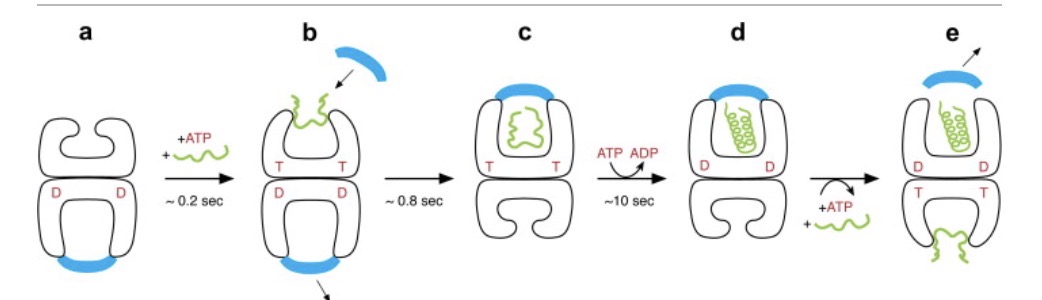
DNA K und DNA J System
DNA J erkennt hydrophobe Sequenzen
Hilft bei der Faltung von neuen Proteinen oder teilweise gefalteten
Essentiell fürs überleben unter Hitze, Dehydrierung
DNA K bindet an das Substrat und hydrosiert ATP
Was ist Renautierung ?
die Rückkehr von Biomolekülen (wie Proteinen oder DNA) in ihre ursprüngliche, funktionale dreidimensionale Struktur, nachdem sie durch Denaturierung (Hitze, Säure) ihre Form verloren haben
Framework Modell
Zuerst Bildung von α-Helices & β-Faltblättern
Danach Zusammenlagerung zur Tertiärstruktur
Vorteil: erklärt gezielte Bildung von sekundärstrukturen
Am besten arbeiten beide zusammen
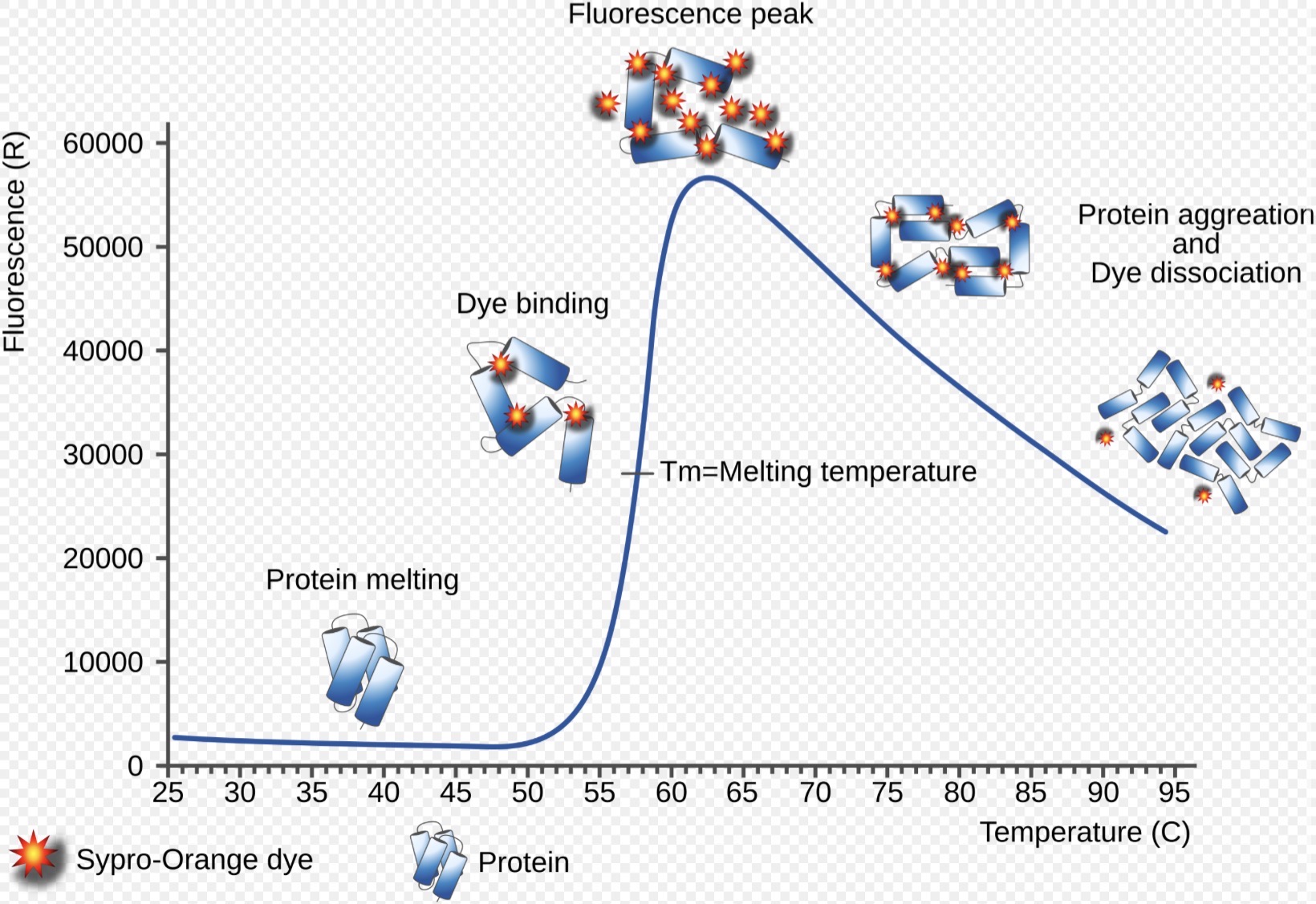
Eine Methode zur Messung der Schmelztemperatur eines Proteins besteht darin es mit einem Fluorophor zu inkubieren und die Temperatur schrittweise zu erhöhen. Der Farbstoff zeigt dabei nur in einer hydrophoben Umgebung ein Fluoreszenzsignal. Zeichnen sie ein Diagramm, in dem die Messwerte der Fluoreszenzintensitäten eines solchen Experiments aufgezeichnet wurden und kennzeichnen sie die Schmelztemperatur. Wie ist diese definiert?
Ein Flourophor bindet nur an hydrophobe Bereiche
Im nativen Protein sind sie meist verborgen → geringe Fluoreszenz
Beim erhitzen entfaltet sich das Protein → hydrophobe Bereiche werden exponiert → Fluoreszenz steigt
Schmelztemperatur: Tm= T bei der 50% des Proteins denaturiert sind
Entspricht dem Wendepunkt der Kurve
Was sind chaperone ?
Sie verhindern Aggregation fehlgefalteter Proteine
– neusynthese
– Stress ( Hitze)
– Renautierung