T8 | Variantes genéticas como um risco
1/9
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
10 Terms
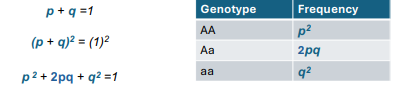
Princípio de Hardy-Weinberg
Este principio dita que uma variação genética numa população vai se manter constante de uma geração para outra, na ausência de fatores de disturbio.
Este pode ser pertubado por mutações, seleção natural, acasalamento não-aleatório, deriva genética e fluxo gênico (migração).
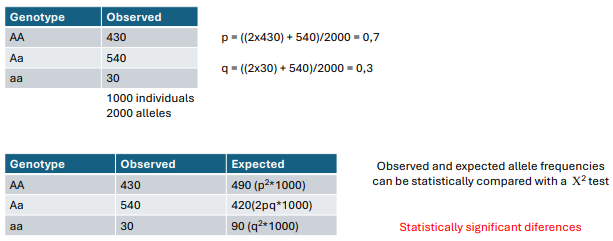
Como testar se uma população está no equilíbrio Hardy-Winberg
Importância do princípio
Podemos calcular a quantidade de carriers de uma determinada doença autossomal genética recessiva.

Estudos de associação
Candidate gene aproach - pequeno nº de genes escolhidos à priori baseado na relevância que tem para o mecanismo de ação de um fármaco.
Genome Wide Association Studies - Estudos que ajudam a identificar variações genéticas em todo o genoma e como essas variações podem estar relacionadas à predisposição a doenças, sem precisar de uma hipótese específica.
Para determinadas doenças, determinadas vias de sinalização são importantes para a causa das mesmas
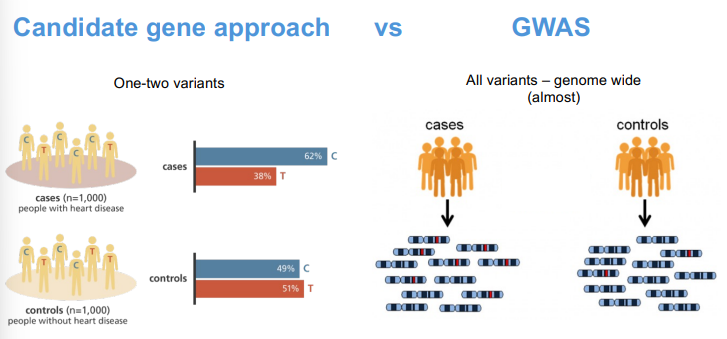
Candidate gene approach
Essa abordagem seleciona um pequeno número de genes que já são conhecidos ou suspeitos de estarem envolvidos no mecanismo de ação de uma doença ou resposta a um medicamento.
É baseada em conhecimento prévio sobre a função dos genes e sua relação com o fenótipo estudado.
Limitação: pode ignorar genes importantes que ainda não foram identificados como relevantes.
Tem apenas 1 ou 2 variantes
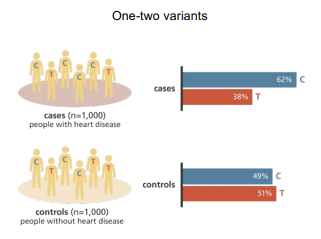
GWAS - Estudos de Associação Genômica Ampla
Abordagem sem hipótese prévia (hypothesis-free), ou seja, analisa todo o genoma sem focar em genes específicos.
Identifica variantes genéticas (polimorfismos) associadas a doenças e outras características.
Usa grandes conjuntos de dados para encontrar relações estatísticas entre variantes comuns no DNA e o risco de desenvolver uma doença.
Exemplo: GWAS pode ajudar a encontrar novas mutações associadas ao câncer, diabetes ou doenças neurodegenerativas.

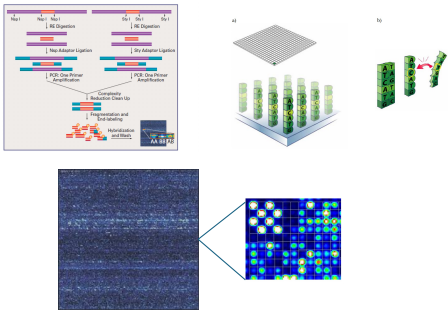
Genome-Wide Human SNPs Arrays
Tecnologia usada para analisar variações genéticas no genoma humano, especificamente os SNPs (Single Nucleotide Polymorphisms).
"arrays" são chips de laboratório que contêm uma grande quantidade de sondas (pequenas sequências de DNA) que se ligam a sequências genéticas específicas. Esses arrays permitem detectar e identificar múltiplos SNPs ao mesmo tempo.
É uma ferramenta usada para examinar uma grande quantidade de variações genéticas (SNPs) em todo o genoma humano de uma vez. Isso é útil para estudos de associação genética, que buscam entender como essas variações podem estar relacionadas a doenças ou outras características genéticas.
Next generation sequencing (NGS)
É uma tecnologia avançada de sequenciamento de DNA que permite determinar a sequência exata de nucleotídeos (as "letras" A, T, C, G) em uma molécula de DNA de forma rápida, precisa e a um custo muito mais baixo do que as tecnologias anteriores.
O NGS consegue sequenciar milhões de fragmentos de DNA ao mesmo tempo, de forma paralela, em vez de sequenciar um de cada vez como nas tecnologias mais antigas.
NGS na Illumina Platform
A plataforma Illumina é uma das tecnologias de sequenciamento de DNA mais populares e amplamente utilizadas no campo de Next Generation Sequencing (NGS).
1. Template Generation (Geração do Template)
O DNA a ser sequenciado é extraído da amostra.
O DNA é fragmentado em pedaços menores, criando as bibliotecas de DNA.
Esses fragmentos são então adaptados com sequências específicas (chamadas de adaptadores), necessárias para que o DNA se ligue à superfície do chip de sequenciamento.
2. Clonal Amplification (Amplificação Clonal)
Os fragmentos de DNA adaptados são carregados em uma superfície (como uma lâmina de fluxo).
Os fragmentos se anexam à superfície e, então, são amplificados por PCR (reação em cadeia da polimerase), criando clusters.
Cada cluster contém cópias idênticas do mesmo fragmento de DNA, o que permite detectar sequências com precisão.
3. Sequencing Reaction (Reação de Sequenciamento)
Durante o sequenciamento, os fragmentos de DNA amplificados são lidos uma base por vez.
As bases (A, T, C, G) são incorporadas sequencialmente em cada fragmento, e a reação gera sinais fluorescentes para cada base.
Esses sinais são captados por câmeras e processados para determinar a sequência de nucleotídeos de cada fragmento de DNA.