Chapitre 1 : The reference human genome
1/149
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
150 Terms
Anatomie basique du génome humain
Taille du génome & composition
❓ Qu’est-ce que la génétique ?
L’étude scientifique de la variation héréditaire chez les organismes vivants.
❓ Qu’est-ce que la génomique ?
L’étude de la structure et de la fonction des génomes
❓ Quels sont les 4 nucléotides de l’ADN ?
➡ Adénine (A), Cytosine (C), Thymine (T), Guanine (G).
❓ Quelles sont les 2 grandes informations codées dans le génome ?
a) Les gènes (protéines ou ARN).
b) La régulation des gènes.
❓ Le code génétique est-il identique chez tous les organismes vivants ?
➡ Presque, mais avec une exception : les mitochondries.
❓ Quelle est la composition en bases du génome humain ?
➡ Environ 40% GC et 60% AT.
❓ Comment varie le contenu GC chez les bactéries ?
➡ Entre 20% et 72%. Donc le contenu en GC peut varier fortement.
❓ Quel est le taux de méthylation des dinucléotides CpG chez les mammifères ?
➡ Environ 75%.
❓ Quelle mutation fréquente est liée aux CpG ?
➡ La désamination de C en T → CpG → TpG (10 à 50x plus fréquente que d’autres mutations, surtout durant la méiose/crossing-over).
❓ Que sont les isochores ?
➡ Anciens termes décrivant des régions supposées homogènes en GC.
WGS a prouvé que ça n’existait pas vraiment
❓ Quelle est la taille du génome humain ?
➡ Environ 3,2 Gb.
La taille du génome peut varier fortement entre les espèces (ex. E.coli quelques Mb)
❓ Qu’est-ce que le paradoxe de la valeur C (C-value paradox) ?
➡ La taille du génome (C-value est la somme d’ADN dans des cellules haploïdes en pg) ne corrèle pas avec la complexité de l’organisme.
→ Certains animaux ont des génomes énormes, composés majoritairement de séquences répétitives. Ils ont parfois un génome 40x plus grand.
Quelle taille fait le génome chez les mammifères ?
➡ De 1,63 pg à 8,40 pg (moyenne ≈ 3,20 pg).
Chromosomes
❓ Quelle proportion de l’ADN cellulaire est mitochondrial ?
➡ ≈ 1%.
❓ Comment visualiser les chromosomes condensés ?
➡ Par G-banding (trypsine + colorant spécifique → caryotype visible).
❓ Les mammifères ont-ils tous le même nombre de chromosomes ?
➡ Non, mais ils sont toujours diploïdes.
❓ Quelle découverte a montré la fusion de chromosomes chez l’humain ?
→ Le chromosome 2 humain provient de la fusion de deux chromosomes de grands singes.
Cette constatation a pu être réalisé grâce à une génomique comparative basée sur l’observation des bandes G en prophase tardive sur différentes espèces.
❓ Qu’est-ce que la syntenie ?
→ Une forte conservation de l’ordre et du contenu génique entre espèces.
→ C’est le cas entre les grands singes et l’humain
Quelle technique permet d’étudier la conservation évolutive des segments chromosomiques ?
→ Zoo-FISH (hybridation in situ fluorescente avec ADN humain marqué).
Sert à étudier la conservation évolutive des segments chromosomiques
Exemple d’utilisation du Zoo-FISH ?
→ Hybridation des chromosomes du cheval avec des sondes d’ADN du chromosome 4 humain (HSA4).
Qu’est-ce que les régions pseudo-autosomiques (PAR) ?
→ Régions communes aux chromosomes X et Y qui permettent la recombinaison.
Qu’a montré la syntenie du chromosome X chez les mammifères placentaire ?
→ Une forte conservation.
Comment s’est différencié le chromosome Y de l’X au cours de l’évolution ?
→ Acquisition du gène SRY (détermination mâle), restriction de recombinaison, perte de nombreux gènes non spécifiques du sexe.
Quels gènes le Y a-t-il conservés ?
→ Ceux essentiels à la fertilité mâle et à la détermination sexuelle.
Comment est déterminé le sexe chez les mammifères ?
→ XX = femelle, XY = mâle. Le gène SRY inhibe Foxl2, empêchant la différenciation ovarienne.
Comment est déterminé le sexe chez les oiseaux ?
→ ZW = femelle, ZZ = mâle. Une double dose de Dmrt1 → différenciation testiculaire ; Foxl2 → différenciation ovarienne.
Quelle est l’origine des chromosomes sexuels XY et ZW ?
→ Des autosomes qui ont acquis des gènes de détermination sexuelle (SRY issu d’un allèle de SOX3). Le chromosome porteur est devenu un proto-Y.
Exemple d’espèce ayant perdu son Y et son SRY ?
→ Le rat épineux d’Amami ; duplication spécifique de SOX9 a remplacé SRY.
Qu’est-ce qui contribue à la dégénérescence du chromosome Y ?
→ Suppression de la recombinaison (inversions), accumulation de mutations, perte de gènes non spécifiques.
Quelle est la particularité du chromosome Y actuel ?
→ Riche en hétérochromatine et contient plusieurs copies de certains gènes (ex. TSPY).
Coding and Non-coding Genes
❓ Combien de gènes possèdent les mammifères environ ?
→ ≈ 20 000 gènes.
❓ Comment explique-t-on ce nombre relativement faible chez l’humain ?
→ Grâce à l’épissage alternatif, qui permet de générer plusieurs transcripts à partir d’un même gène.
→ ≈ 390 000 transcripts pour ≈ 20 000 gènes.
❓ Quels sont les principaux types d’ARN non codants et leur rôle ?
→
rRNA : ribosomes
tRNA : traduction
snRNA (snRNP) : épissage
snoRNA : modification du rRNA
lncRNA : régulation transcriptionnelle (ex. XIST)
sncRNA : régulation traductionnelle (ex. miRNA)
❓ Exemple de fonction d’un lncRNA important ?
→ XIST condense et inactive un chromosome X dans un embryon XX via l’action sur le site XIC.
❓ Comment fonctionnent les microRNAs (miRNA) ?
→ Ils reconnaissent des séquences cibles d’ARNm :
Un miRNA peut cibler plusieurs ARNm.
Un ARNm peut être ciblé par plusieurs miRNAs.
Dans les plantes : homologie parfaite → dégradation ; partielle → inhibition traduction.
Dans les mammifères : siRNA = correspondance parfaite → dégradation ; miRNA = correspondance partielle → répression traduction.
❓ Différence de compensation de dosage des chromosomes sexuels entre mammifères et oiseaux ?
→ Chez les oiseaux, seuls les gènes sensibles au dosage sont réprimés par miRNAs pour éviter la surexpression.
❓ Répartition des gènes non codants ?
→ ≈ 35 000 lncRNAs, 5 000 sncRNAs, le reste misc noncoding genes.
❓ Qu’est-ce qu’un ortholog ?
→ Gènes identiques avec fonctions identiques dans différentes espèces.
❓ Qu’est-ce qu’un paralog ?
→ Gènes issus d’une duplication suivie d’une divergence.
Ex. Gènes de l’hémoglobine : forme fœtale (forte affinité O₂) vs adulte.
❓ Qu’est-ce qu’un pseudogène ?
→ Copie non fonctionnelle d’un gène.
❓ Quelles sont les deux grandes catégories de pseudogènes ?
→
Unprocessed (duplicated) : duplication → une copie fonctionnelle + une copie accumule des mutations
Processed : reverse transcription d’un ARNm → insertion dans le génome (pas d’introns, pas de promoteur, possible queue polyA). C’est médié par une machinerie de rétrotransposon
❓ Pourquoi en sait-on plus sur les gènes non codants humains que sur ceux d’autres mammifères ?
→ Le génome humain est plus étudié, d’autres espèces pourraient avoir plus de gènes non codants encore non identifiés.
Repetitives sequences
❓ Quelle proportion du génome code pour des protéines ?
→ Seulement 2 % ; le reste est constitué de séquences répétitives.
❓ Qu’est-ce que l’ADN satellite ?
→ Séquences répétées en tandem, importantes pour centromères et télomères.
❓ Quand et comment les satellites ont-ils été découverts ?
→ Dans les années 1960, via centrifugation en gradient.
❓ Quels sont les deux types de satellites selon leur taille ?
→
Minisatellites : 10-60 bases
Microsatellites : 1-6 bases (utilisés en forensique et diagnostic, ex. maladie de Huntington caractérisée par un expansion au niveau de cette région).
❓ Qu’est-ce que l’ADN intercalé (interspersed repetitive DNA) ?
→ Éléments génétiques mobiles présents dans le génome.
❓ Classification des éléments mobiles selon leur type de nucléique :
→
Classe 1 : RNA elements → LINEs, SINEs, éléments rétrovirus-like (ERV)
Classe 2 : DNA transposons fossils
❓ Ces séquences sont-elles toujours parasites ?
→ Initialement considérées comme des parasites génomiques, mais certaines ont été intégrées dans l’ADN de l’hôte(ex. syncytin-1 dérivé d’ERV, rôle dans la placentation).
❓ Comment les éléments transposables expriment-ils leurs gènes ?
→ Ils utilisent l’hôte et contiennent des séquences cis-régulatrices mimant les promoteurs de l’hôte.
❓ Quel est l’effet de leur insertion ?
→ Dépend du contexte local de l’ADN.
❓ LINEs et SINEs sont-ils régulés ?
→ Oui, ils sont souvent fortement méthylés pour limiter leur activité.
❓ Que montre la conservation des gènes orthologues entre espèces ?
→ Les gènes sont conservés, mais les régions flanquantes contiennent souvent différents éléments transposables ou répétitifs
❓ Qu’est-ce que les low-copy repeats (ou segmental duplications) ?
→ Grands segments d’ADN (1-200 kb) parfois dupliqués dans le génome.
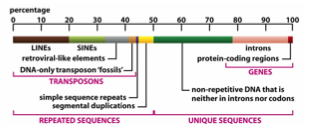
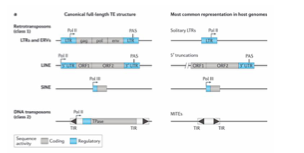
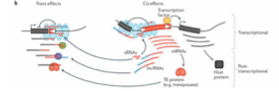
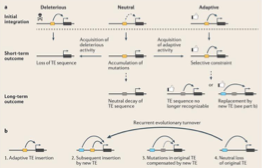
Gene regulation
❓ Comment le génome est-il organisé dans le noyau ?
→ En territoires chromosomiques puis en TADs (Topologically Associating Domains).
❓ Que séparent les TADs adjacents ?
→ Des séquences isolatrices (insulators) qui protègent les gènes des effets des régulateurs distants des TADs voisins.
❓ Quelles sont les deux états possibles des TADs ?
→ Actifs ou réprimés.
❓ Où se situent généralement les TADs réprimés ?
→ En périphérie du noyau, associés à la lamina nucléaire.
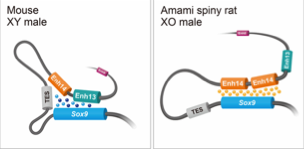
❓ Comment Sox9 est-il activé chez le mâle souris ?
→ SRY et NR5A1 se lient à des enhancers en amont, rapprochés du promoteur de Sox9 via chromatin looping, ce qui déclenche son expression.
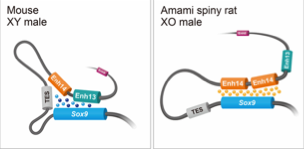
❓ Comment Sox9 est-il activé chez le rat épineux d’Amami, qui a perdu son chromosome Y ?
→ Un autre facteur de transcription se lie directement à Sox9 pour l’activer, remplaçant la fonction de SRY.
Epigenetics
❓ Que désigne le terme “épigénétique” ?
→ Tout ce qui est nécessaire au‑dessus de l’ADN pour permettre l’exécution correcte du programme génétique.
❓ Quels types de marques épigénétiques peuvent porter les cellules filles après mitose ?
→ Methylation de l’ADN et modifications post-traductionnelles des queues amino-terminales des histones.
❓ Quel rôle joue DNMT1 ?
→ Méthylation de maintenance et méthylation des cytosines dans les CpG, souvent pour silencer les gènes.
❓ Quels rôles jouent DNMT3a et DNMT3b ?
→ Ce sont des de novo DNA methylases, elles établissent de nouvelles patterns de méthylation.
❓ Quels complexes modifient les queues des histones ?
→ Polycomb Repressive Complex 1 et 2 (PRC1/2).
❓ Que déterminent les combinaisons de modifications des histones ?
→ Différents états de la chromatine (active ou réprimée).
❓ Quand les cellules germinales primordiales de la souris sont-elles déméthylées ?
→ Très tôt dans le développement.
❓ Quand la reméthylation se produit-elle ?
→ Pendant la gamétogenèse, à des moments différents selon le sexe.
❓ Après fécondation, que se passe-t-il pour les génomes parentaux ?
→
Génome paternel : déméthylation active rapide
Génome maternel : déméthylation passive pendant la réplication d’ADN
Méthylation est vraiment bas au stade blastocyte
❓ Quand se produit la méthylation de novo après fécondation ?
→ Après implantation, avec des niveaux différents :
Lignée embryonnaire : gain de méthylation
Lignées extraembryonnaires : restent relativement hypométhylées
❓ Les mécanismes épigénétiques peuvent-ils contribuer à des différences héréditaires ?
→ Oui, bien que débattu ; exemples établis chez plantes et souris.
❓ Exemple d’epiallele : activation d’un rétrotransposon IAP en amont du gène agouti
→ Plus la méthylation est faible, plus l’IAP est actif. La méthylation peut être partiellement transmise à la descendance. Plus la souris a un allèle méthylé, plus elle a tendance à transmettre l’épiallèle méthylé à sa progéniture.
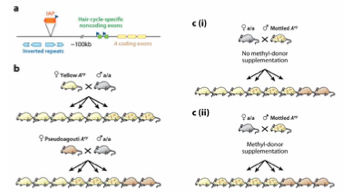
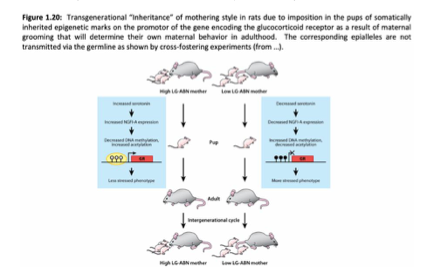
❓ Transmission épigénétique intergénérationnelle : toujours par la lignée germinale ?
→ Non, elle peut aussi se faire via le comportement.
❓ Qu’est-ce que l’imprinting parental ?
→ 150 gènes environ : un allèle est exprimé, l’autre est épigénétiquement réprimé.
❓ Quelle détermine l’allèle actif ?
→ L’origine parentale : paternal ou maternal. 50/50
❓ Où se trouvent la plupart des gènes imprintés ?
→ Dans des imprinted domains, souvent riches en lncRNAs non codants.
❓ Quelle est la base moléculaire de l’imprinting parental ?
→ Acquisition d’une marque épigénétique dans une des lignées germinales, souvent méthylation de l’ADN, plus fréquente dans le germline paternel.
❓ Quel est le rôle fonctionnel de l’imprinting parental chez les mammifères ?
→ Affecte surtout des gènes foetaux contrôlant le transfert de ressources de la mère vers la progéniture.
❓ Pourquoi l’imprinting parental aurait-il émergé ?
→ Chez les espèces avec un investissement maternel supérieur par rapport au paternel.
Microbiomes and microbiomes
❓ Que désigne le terme microbiota ?
→ Ensemble des bactéries, archées, protozoaires, virus et phages qui colonisent notre corps.
❓ Que désigne le terme microbiome ?
→ L’ensemble des gènes de ces micro-organismes.
❓ Quelle est la proportion estimée de cellules bactériennes par rapport aux cellules de l’hôte ?
→ Environ 10 fois plus de cellules bactériennes que de cellules humaines.
❓ Quelle est la proportion estimée de gènes microbiens par rapport aux gènes humains ?
→ Environ 150 fois plus de gènes microbiens.
❓ Quelle technique est utilisée pour déterminer la composition taxonomique globale ?
→ Le séquençage du gène ribosomal 16S.
❓ Quelle technique permet une résolution au niveau de l’espèce ?
→ Le séquençage shotgun suivi de l’assemblage des séquences.
❓ Qu’est-ce qu’une approche ciblée (targeted) ?
→ Utilisation d’une PCR universelle sur des régions conservées :
rRNA 16S chez les bactéries
rRNA 18S ou 28S chez les champignons et parasites
ITS (Internal Transcribed Spacer) chez les champignons et parasites
Possibilité aussi de RT-PCR ou multiplex PCR.
❓ Qu’est-ce qu’une approche non ciblée (untargeted) ?
→ Alignement du DNA et cDNA du prélèvement avec une bibliothèque de référence pour identifier les espèces présentes.
❓ Quels facteurs environnementaux peuvent affecter le microbiome intestinal ?
→ L’alimentation, le mode de vie, les conditions environnementales, etc.
❓ Le génome de l’hôte influence-t-il le microbiome ?
→ Oui, il existe des preuves que la génétique de l’hôte peut influencer sa composition.