Moleculaire Biologie
moleculaire biologie
= studie van het leven op moleculair niveau
mengdiscipline van genetica & biochemie
centrale dogma: DNA → RNA → eiwit
DNA is het “transforming principle”: Frederick Griffith (1928)
experimenten met streptococcus pneumoniae → voor een vaccin tegen longontsteking te ontwikkelen
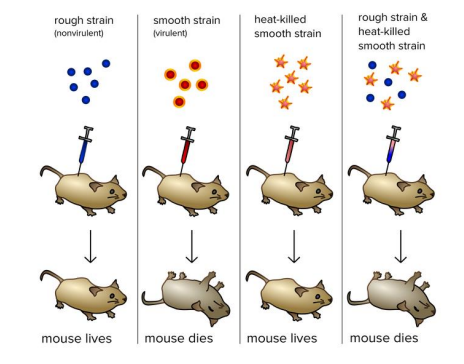
2 verwante bacteriestammen:
stam R (‘rough’) → niet-virulent, maken geïnfecteerde muizen niet ziek
stam S (‘smooth’) → met polysaccharidenmantel, beschermt tegen immunologische afweermechanismen van de gastheer
besmette muizen: ernstige longontsteking → dood
experiment: muizen injecteren met dode S bacteriën
muizen overleven de infectie
experiment: muizen injecteren met mengsel van dode S bacteriën & levende R bacteriën
muizen sterven
in het bloed: levende S bacteriën
→ conclusie: levende R bacteriën hebben een transformerende factor (‘transforming principle’) overgenomen van de dode S bacteriën
R bacteriën waren getransformeerd in S bacteriën
1/400
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
401 Terms
moleculaire biologie
= studie van het leven op moleculair niveau
mengdiscipline van genetica & biochemie
centrale dogma: DNA → RNA → eiwit
DNA is het “transforming principle”: Frederick Griffith (1928)
experimenten met streptococcus pneumoniae → voor een vaccin tegen longontsteking te ontwikkelen
2 verwante bacteriestammen:
stam R (‘rough’) → niet-virulent, maken geïnfecteerde muizen niet ziek
stam S (‘smooth’) → met polysaccharidenmantel, beschermt tegen immunologische afweermechanismen van de gastheer
besmette muizen: ernstige longontsteking → dood
experiment: muizen injecteren met dode S bacteriën
muizen overleven de infectie
experiment: muizen injecteren met mengsel van dode S bacteriën & levende R bacteriën
muizen sterven
in het bloed: levende S bacteriën
→ conclusie: levende R bacteriën hebben een transformerende factor (‘transforming principle’) overgenomen van de dode S bacteriën
R bacteriën waren getransformeerd in S bacteriën

serendipity
= door toevalligheden belangrijke ontdekkingen doen
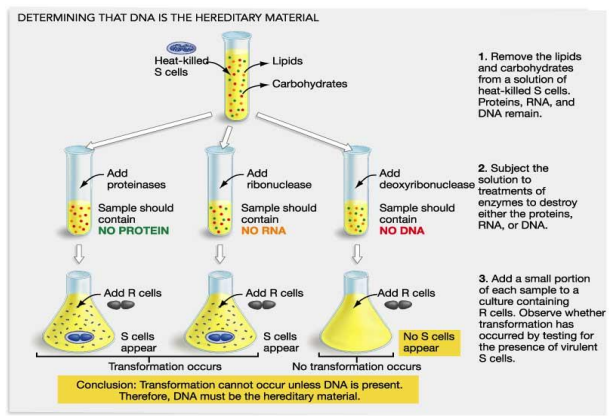
DNA is het “transforming principle”: Avery, McLeod & McCarthy (1944)
zoeken naar de biochemische identiteit van Griffith’s ‘transforming principle’
S bacteriën in vitro groeien & bacteriën doden door verhitting → dan gedode bacteriën behandelen met enzymen die specifiek bepaalde biomoleculen degraderen
proteasen => geen proteïnen meer
ribonucleasen => geen RNA meer
desoxyribonucleasen => geen DNA meer
→ enzymatisch behandelde dode S bacteriën combineren met levende R bacteriën => resultaat:
enkel levende S cellen in kweken met proteinase- & ribonuclease-behandelde S-cellen, niet in kweken met deoxyribonuclease-behandelde bacteriën
→ conclusie: DNA is (waarschijnlijk) de transformerende factor
(veel wetenschappers toch niet overtuigd van experiment → dachten nog steeds dat eiwitten de erfelijkheidsdragers waren)
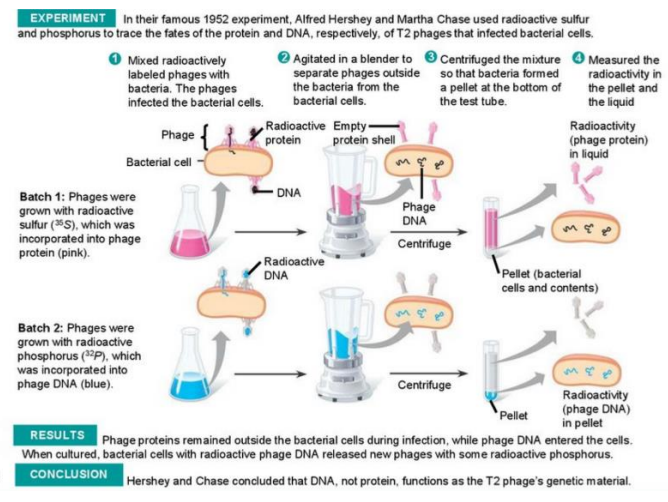
DNA is het “transforming principle”: Alfred Hershey & Martha Chase (1950)
experiment met bacteriofagen
bacteriofagen = virussen die bacteriën infecteren
simpele partikels → enkel eiwit & DNA
bacteriofagen infecteren bacteriën → dringen de bacterie niet binnen, maar injecteren een substantie in de bacteriën → substantie instrueert bacteriën om nieuwe faagpartikels aan te maken
substantie die de fagen injecteren bevat hun genetisch materiaal
eiwit of DNA-injectie? → experiment met radio-isotopen
fagen gegroeid met 35S (bij eiwitten) → enkel faageiwitten radioactief gekenmerkt
zwavel komt niet voor in DNA
DNA gekenmerkt met 32P → fosfor komt niet voor in eiwitten (tenzij na post-translationele modificatie, maar dat is voor faageiwitten niet van toepassing)
gemerkte fagen gebruiken om bacteriën te infecteren → daarna bacteriën & fagen in blender om van elkaar te scheiden
mengsel van bacteriën & fagen gecentrifugeerd
zwaardere partikels (bacteriën) werden gepelleteerd
lichtere fagen bleven in oplossing
→ waar bevindt de radio-activiteit zich?
35S (eiwit) signaal enkel in de fagen
32P (DNA) signaal volledig in de bacteriën
→ conclusie: DNA is de transformerende factor & de erfelijkheidsdrager
Een model voor de structuur van DNA: Erwin Chargaff (1949)
analyse van de basensamenstelling in DNA van verschillende soorten
vaststelling 1: A, T, C & G niet in gelijke hoeveelheden → hypothese van tetranucleotide verworpen
vaststelling 2: hoeveelheid van de verschillende basen verschillen tussen individuen van verschillende soorten, maar niet tussen individuen van eenzelfde soort
vaststelling 3: in alle onderzochte organismen is de hoeveelheid A steeds gelijk aan de hoeveelheid T, terwijl de hoeveelheid C altijd gelijk was aan de hoeveelheid G
= regel van Chargaff
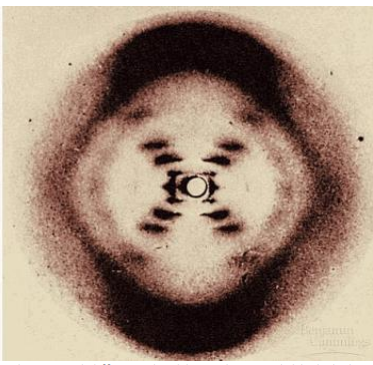
Een model voor de structuur van DNA: James Watson & Francis Crick (1953)
model voor de DNA structuur = dubbele helix
artikel geschreven in toonaangevende tijdschrift Nature
→ ontdekking gebaseerd op X-straal diffractie resultaten bekomen door Rosalind Franklin, die werkte in het laboratorium van Maurice Wilkins
Watson & Crick vergaarden zelf geen experimentele data, maar bouwden 3D modellen obv gekende moleculaire afstanden & bindingshoeken (methode ontwikkeld door Linus Pauling)
door data van Rosalind Franklin + bevindingen van Chargaff: correcte structuur van DNA gemodelleerd
Een model voor de structuur van DNA: Linus Pauling (1951)
toonde aan dat eiwitten een helicale secundaire structuur kunnen aannemen (α-helix)
→ was zelf ook op zoek naar de structuur van DNA
Het centrale dogma: Francis Crick (1957)
hoe wordt de info in het DNA gebruikt om eiwitten te maken? → beschrijving van informatiestroom van DNA naar eiwit: beschreven door Francis Crick als het ‘centrale dogma’ van de moleculaire biologie
DNA → RNA → eiwit
DNA codeert voor RNA, dat codeert voor eiwit
centrale dogma houdt nog steeds stand, maar wel enkele uitzonderingen
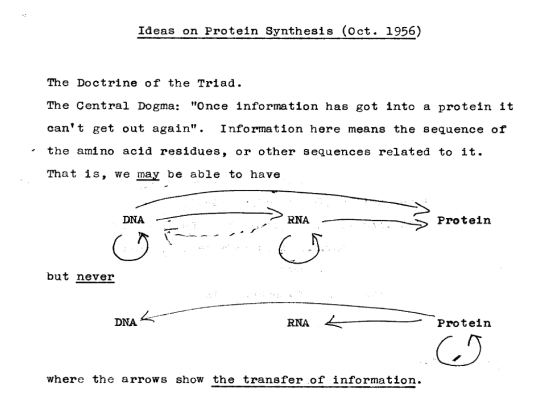
replicatie
= proces waarbij een exacte DNA kopie van de originele DNA matrijs wordt gemaakt
transcriptie
= proces waarbij DNA wordt gekopieerd naar een enkelstrengig RNA met dezelfde sequentie als 1 van de DNA strengen
→ dezelfde taal (nucleotiden) wordt gebruikt in DNA & RNA
translatie
= proces waarbij de RNA nucleotidesequentie wordt omgezet in eiwit
→ in een andere taal omgezet
Test jezelf H1

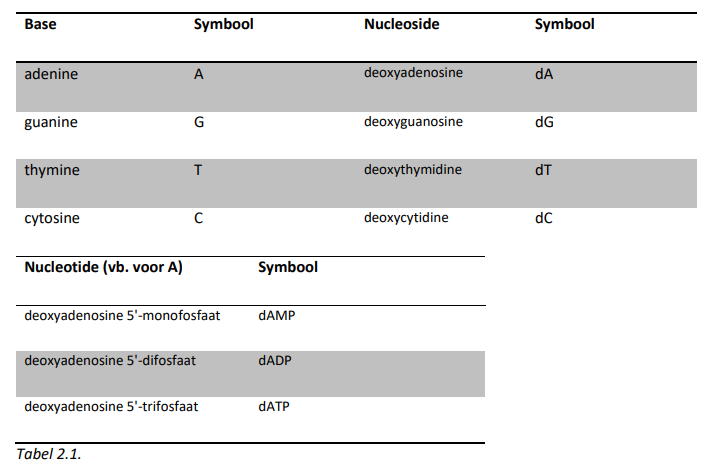
De primaire structuur van DNA: bouwstenen
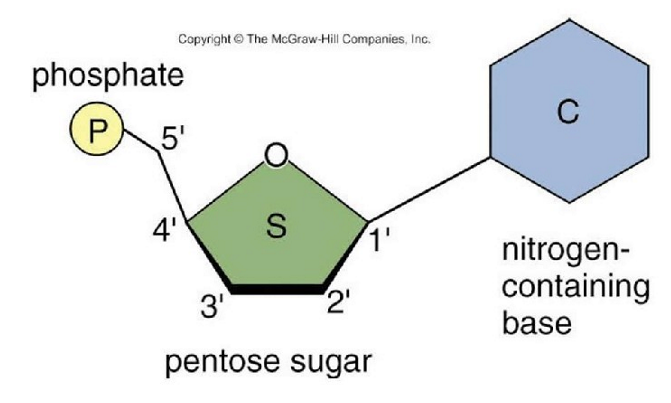
DNA = polymeer van nucleotide bouwstenen
nucleotide: suiker met 5 koolstoffen (pentose), fosfaatgroep & stikstofhoudende base
stikstofhoudende base: adenine (A), thymine (T), cytosine (G), guanine (G)
pentose in DNA → desoxyribose
β-D configuratie
nummering van koolstoffen: accenten → om onderscheid met koolstofnummering in de basen te maken
esterbinding met fosfaatgroep: op 5’-C
koppeling van base: op 1’-C
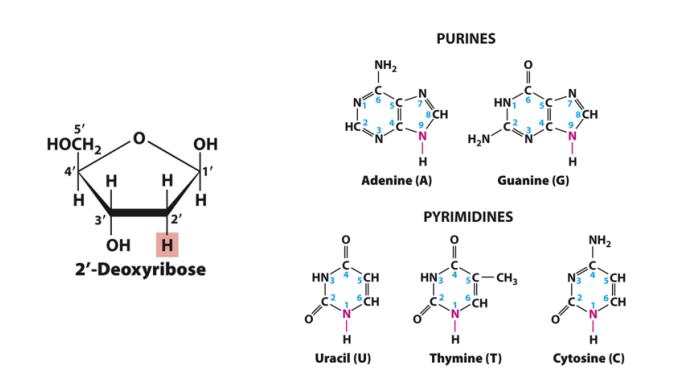
De primaire structuur van DNA: basen
stikstofhoudende moleculen met chemische eigenschappen van een base (molecule die in oplossing een H+ ion of proton kan accepteren)
niet geprotoneerd bij neutrale pH
purines
adenine & guanine
2 koolstof-stikstof ringen
met 9-N gekoppeld aan 1’-C op het pentose
pyrimidines
thymine & cytosine
enkele ringstructuur
met 1-N gekoppeld aan 1’-C op het pentose
De primaire structuur van DNA: fosfaatgroep
verleent het DNA zijn zure karakter
bij fysiologische pH: fosfaatgroep dissocieert → zorgt voor sterke negatieve lading van DNA
De primaire structuur van DNA: nomenclatuur
vorming DNA-molecule: 3 stappen
stikstofhoudende base gekoppeld aan 1’-C van het pentose → vorming nucleoside
fosfaatgroep covalent gebonden aan 5’-C van de ribose → vorming nucleotide
nucleotiden polymeriseren door een condensatiereactie tot een lange keten → hydroxylgroep (OH) op 3’-C van een suiker van 1 nucleotide zal reageren met fosfaatgroep op 5’-C van een suiker van een 2e nucleotide met vorming van een esterbinding
eliminatie van 1 molecule water & vrijstelling van 2 fosfaten (pyrofosfaat P2O74-)
= fosfodiësterbinding (suikers van 2 nucleotiden met mekaar verbonden)
na vorming van fosfodiësterbinding: 1 zuurstof van de fosfaatgroep nog steeds negatief geladen
fosfodiësterbinding is covalent & zeer stabiel
weinig spontane hydrolyse
fosfodiësterbinding wel doelwit van enzymatische hydrolyse
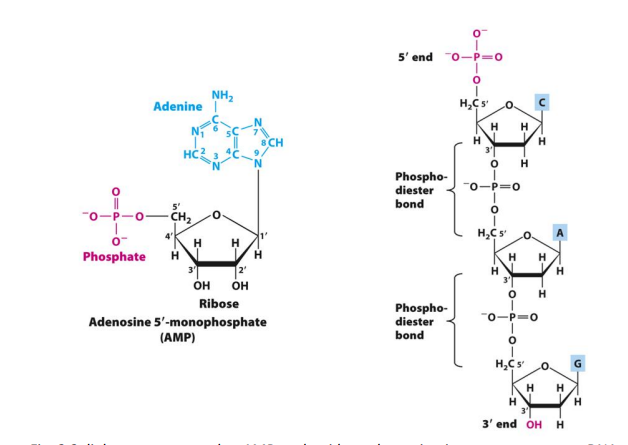
De primaire structuur van DNA: polariteit van de streng
5’ & 3’ uiteinden van een DNA streng verschillen → vertonen verschillende chemische eigenschappen
conventie: 5’ uiteinde links & 3’ uiteinde rechts
basenparen (bp) wordt gebruikt als maat voor de lengte van een dubbelstrengig DNA
De primaire structuur van DNA: oligonucleotiden
= korte enkelstrengige DNA moleculen
De secundaire structuur van DNA: dubbele helix → krachten
zwakke krachten voor secundaire structuur
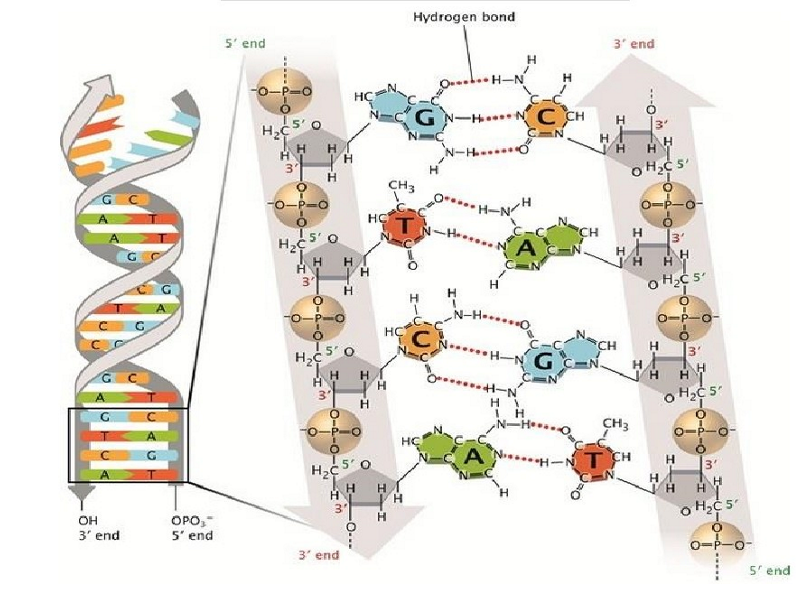
waterstofbruggen tussen stikstofhoudende basen van 2 complementaire DNA strengen
= zwakke bindingen waarbij waterstofatoom gedeeld wordt door 2 elektronegatieve atomen zoals zuurstof & stikstof
→ waterstofbinding op zich erg zwak, maar groot aantal in DNA dubbele helix geeft belangrijke bijdrage aan de stabiliteit van de secundaire structuur
basenparing tussen A & T of G & C: ‘Watson-Crick’ of complementaire basenparing
De secundaire structuur van DNA: base stacking
moleculaire processen van het leven: in waterige oplossing
stikstofhoudende basen zijn apolair → slecht oplosbaar in water
=> apolair karakter beperkt conformatie van DNA helix in oplossing
→ gepaarde, relatief vlakke, basen hebben neiging om zich op mekaar te stapelen = base stacking
gaan zo watermoleculen uit het binnenste van de dubbele helix verdrijven
De secundaire structuur van DNA: elektrostatische interacties (van der Waals)
hydrofobe basen dicht op mekaar gestapeld → worden gestabiliseerd door elektrostatische interacties (van der Waals) tussen atomen
zwakke interacties ontstaan door elektronen die constant in beweging zijn & die de atomen een kleine asymmetrie in lading bezorgen, fluctueert doorheen de tijd
basen naderen mekaar zeer dicht?: partiële lading op 1 base en tegengestelde lading op naburige base → aantrekking tussen beide basen
vrij zwak, maar levert wel belangrijke bijdrage aan de stabiliteit
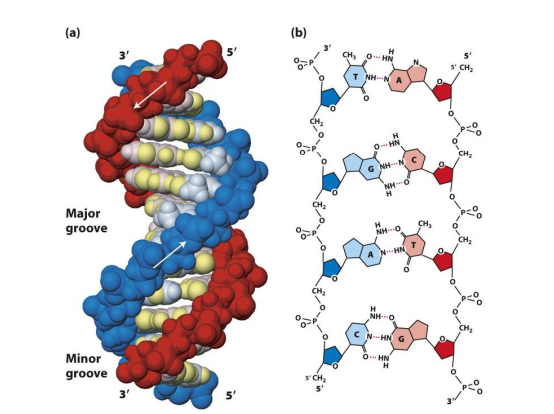
De secundaire structuur van DNA: grote & kleine groeves (‘major and minor grooves’)
2 glycosidische bindingen die een basenpaar verbinden met de desoxyribose suikers liggen niet perfect tegenover mekaar → suiker-fosfaat ruggengraten (‘backbones’) liggen niet op gelijke afstanden van mekaar → °grote & kleine groeves (‘major and minor grooves’)
grote groeve speelt een belangrijke rol in sequentiespecifieke interacties tussen DNA & eiwitten
sommige eiwitten ‘lezen’ chemische info die in groeve aanwezig is
buitenzijden van de purines & pyrimidines zijn in de grote groeve solventietoegankelijk → stikstof & zuurstofatomen van basen die grote groeves aflijnen kunnen waterstofbruggen aangaan met de zijketens van aminozuren van DNA-bindende eiwitten
in kleine groeve: minder chemische info voorhanden → gaat minder vaak specifieke eiwit-DNA interacties aangaan
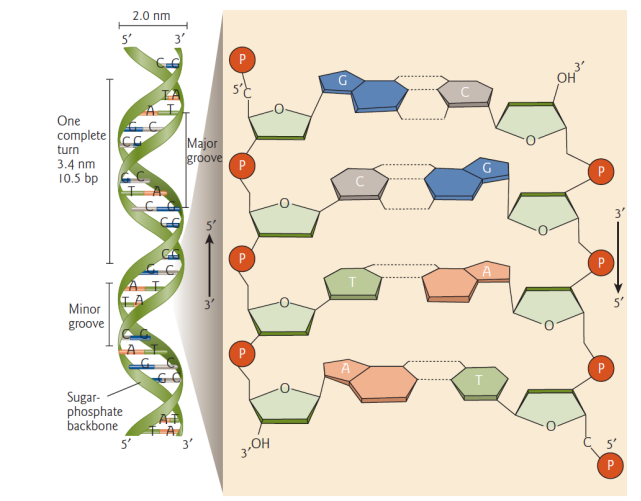
De secundaire structuur van DNA: kenmerken van de secundaire DNA structuur
DNA → dubbelstrengige helix, waarbij strengen door waterstofbruggen worden bijeengehouden
(regel van Chargaff): A gepaard met T, G met C
DNA helix → rechtshandig (draait in wijzerzin van boven naar beneden)
DNA helix → anti-parallel
5’ uiteinde van een streng is gepaard met 3’ uiteinde van de complementaire streng
DNA basenparen zijn verbonden via waterstofbruggen, maar ook de buitenranden van de stikstofhoudende basen kunnen betrokken worden in waterstofbrugvorming
zorgt voor DNA interacties met andere moleculen → essentieel voor replicatie & transcriptie van het DNA

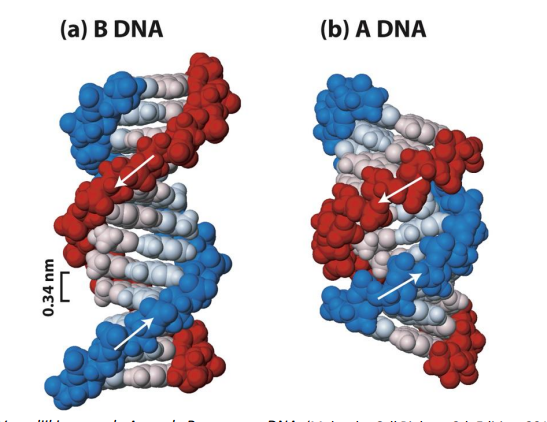
De secundaire structuur van DNA: alternatieve DNA conformaties
→ aangetoond via X-straal diffractie
geometrie & dimensies van DNA dubbele helix kunnen variëren
meest voorkomende conformatie (Watson & Crick structuur) = B-DNA
structuren met alternatieve helix configuratie = A-DNA
meer compact
komt niet voor in fysiologische, waterige milieu
komt wel voor in laboratoriumcondities waarbij organische solventen worden gebruikt & helix gedeshydrateerd wordt
Denaturatie & renaturatie: denaturatie
DNA strengscheiding: noodzakelijke vereiste bij essentiële processen zoals DNA replicatie & transcriptie naar RNA
experimenteel, ‘in vitro’ = denaturatie/ smelten
DNA verwarmen → waterstofbruggen tussen basenparen breken (helix desintegreert) & fosfodiësterbindingen blijven intact
ook andere methoden mogelijk om DNA te denatureren
bv. zoutconcentratie van DNA oplossing verlagen → denaturatie bevorderd want minder kationen aanwezig om negatieve lading van fosfaatgroepen te neutraliseren
ruggengraten van de 2 DNA strengen gaan mekaar afstoten, zodat DNA zelfs bij lage temperatuur kan denatureren
bv. hoge pH (bv. 0.5 M NaOH) of organische solventen kunnen denaturatie van DNA uitlokken
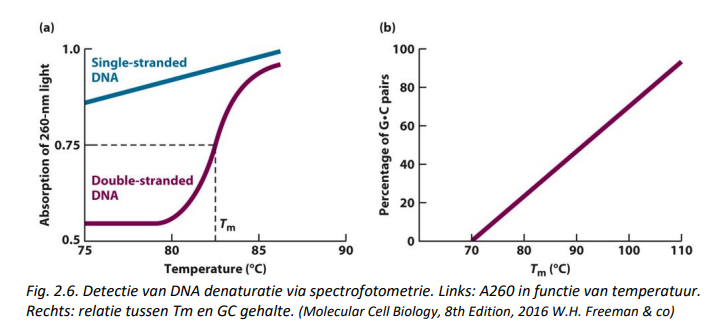
Denaturatie & renaturatie: smelten van DNA → spectrofotometrische meting
basen absorberen ultraviolet licht bij golflengte van 260 nm
absorptie bij enkelstrengig DNA 2x zo sterk als bij dubbelstrengig DNA
hyperchromiciteit = tijdens het smelten van DNA neemt zijn capaciteit om UV licht te absorberen toe
smelttemperatuur/ Tm = temperatuur waarbij de helft van de basenparen in een dubbelstrengige DNA molecule gedenatureerd zijn
Tm-waarde sterk bepaald door G-C gehalte
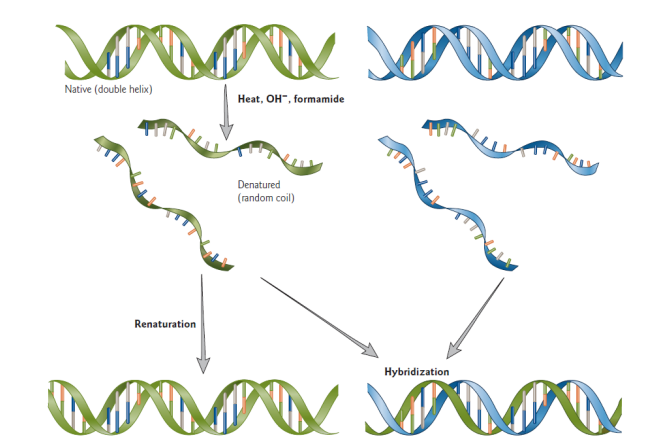
Denaturatie & renaturatie: renaturatie
verhitte, gedenatureerde, DNA oplossingen langzaam afkoelen → complementaire DNA strengen gaan mekaar terugvinden & een nieuwe dubbele helix vormen = renaturatie/ annealing
2 complementaire DNA strengen van verschillende oorsprong kunnen basenparen = hybridisatie
Denaturatie & renaturatie: renaturatiesnelheid wordt bepaald door:
DNA concentratie
ionenconcentratie
fosfaatgroepen hebben negatieve lading
weinig kationen aanwezig in oplossing? → fosfaatgroepen blijven negatief (worden niet geneutraliseerd), strengen gaan mekaar afstoten, renaturatie gaat trager
temperatuur
hogere temperatuur → moeilijkere renaturatie
complexiteit van hybridiserende DNAs
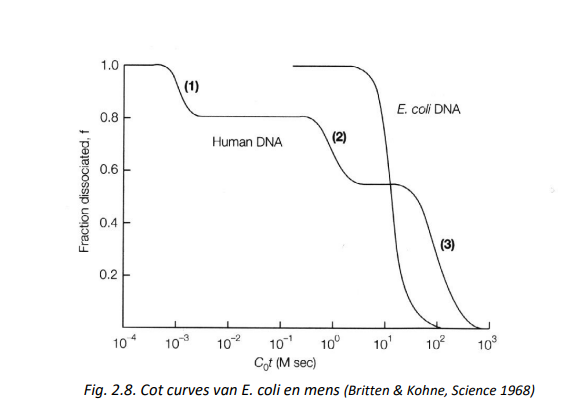
Denaturatie & renaturatie: Cot curve
om renaturatiesnelheid van DNA stalen met verschillende concentraties te vergelijken
Cot curve maken
genomisch DNA wordt in handelbare fragmenten verdeeld, daarna gedenatureerd (enkelstrengig gemaakt) door verhitting
reassociatie van de ssDNA strengen wordt in de tijd spectrofotometrisch gevolgd
Y-as: fractie DNA die nog gedenatureerd is (C/C0)
X-as: product van C0.t waarbij C = concentratie ssDNA op een gegeven tijdstip; C0 = initiële concentratie ssDNA, t = tijd waarop C wordt bepaald
product van C0.t noemt men gemakshalve ‘Cot’
op moment dat renaturatie halfweg is (t = t1/2) is C/C0 = ½
foto: vergelijking van Cot curves van E. coli & H. sapiens
Cot curve voor humaan DNA vertoont verschillende buigpunten
verklaring: humaan DNA is een verzameling van verschillende types DNA: hoog-repetitief, gemiddeld repetitief & uniek DNA
sequenties die meer dan 1 keer voorkomen in een DNA mengsel (repetitief DNA) hebben meer kans om hun complement terug te vinden dan een unieke DNA sequentie → lagere Cot waarde
E. coli: geen repeterende sequenties
De tertiaire structuur van DNA: supercoiling
DNA helix zal als geheel extra windingen in de ruimte vormen, door torsionele stress
veel in de natuur voorkomende DNA moleculen zijn circulair
twee cirkels van enkelstrengig DNA die om mekaar gedraaid zijn
erg vatbaar voor over- of onderwinding van het DNA (positieve vs. negatieve supercoiling)
bijna al het DNA in prokaryote & eukaryote cellen komt voor als supercoils
bv. DNA van eukaryoten → in complexen met architecturale eiwitten (nucleosomen) verpakt in de celkern
dubbele helix bijna 2x in een linkshandige spiraal om een nucleosoom gewonden = negatieve supercoiling van het DNA
structuur gestabiliseerd door zwakke interacties tussen DNA & DNA-bindende eiwitten
basisvereisten van het genetisch materiaal → functies
doorgeven van kenmerken aan het nageslacht
expressie & controle van erfelijke kenmerken
aanpassing aan het milieu
Test jezelf H2
Vele genoomsequenties zijn ontrafeld: genoom
= (gen + chromosoom) geheel aan genetische informatie van een organisme
in 1976: eerste genoomsequentie opgehelderd → van een bacteriofaag
sindsdien: ontwikkeling van DNA sequeneringsmethoden → kennis van duizenden Eubacteria & Archaea
volledige genoomsequenties van typische uni- & multicellulaire eukaryote modelorganismen beschikbaar: bakkersgist (Saccharomyces cerevisiae) - nematode Caenorhabditis elegans - fruitvlieg (Drosophila melanogaster) - plant Arabidopsis thalliana - muis mus musculus - mens (homo sapiens)

Vele genoomsequenties zijn ontrafeld: cellen bij de mens
elke cel bij de mens = diploïd equivalent van het volledige genoom
Klonen van een organisme vanuit een lichaamscel toont aan dat alle informatie nodig voor de ontwikkeling van een individu aanwezig is in alle cellen
uitzonderingen:
gameten (haploïd)
celtypes uit het bloed: erythrocyten (geen kern) + B- & T- lymfocyten (zijn bij opbouw van ‘immuun-repertoire’ DNA recombinaties opgetreden)
→ nagenoeg elk celtype bevat volledige genoom, maar slechts een deel van totale genetische informatie komt tot expressie
Vele genoomsequenties zijn ontrafeld: 3 grote rijken in de biologische wereld
eubacteria
archaea (& prokaryota)
eukaryota
→ indeling ondersteund door vergelijking van sequenties van ribosomale RNAs
ribosomale RNAs → essentieel onderdeel van ribosoom, fundamentele rol bij translatieproces
zeer geschikt voor verwantschapsanalyse, want blijven sterk geconserveerd tijdens de evolutie (ribosoom moet een optimale configuratie hebben zodat cel kan blijven voortbestaan)
Variaties in genoomorganisatie: opbouw van genomen
genomen opgebouwd uit DNA (veel virussen bevatten ook RNA genomen)
enkelstrengig of dubbelstrengig
circulair of lineair
→ alle prokaryote & meeste virale DNA genomen, plasmide, mitochondriale & chloroplast DNAs = circulair
→ sommige virale genomen: kunnen op verschillende wijzen voorkomen
sterke variatie van aantal chromosomen tussen soorten → genomen hebben een zeer dynamische structuur
Variaties in genoomorganisatie: polyploïdie
= volledige genoom tijdens de evolutie gedupliceerd
veel voorkomend bij planten
bv. genoom van zandraket (Arabidopsis thalliana) → 70% gedupliceerde genen
→ veel cultuurgewassen zijn polyploïd
tomaat = tetraploïd
tarwe & verschillende aardappelrassen = hexaploïd
Variaties in genoomorganisatie: genen
bij prokaryoten:
bijna 90% van DNA bestaat uit genen
genen = DNA sequenties die coderen voor eiwitten
± 500 - 8000 genen
bij mensen:
minder dan 2% genen
± 22.000 genen
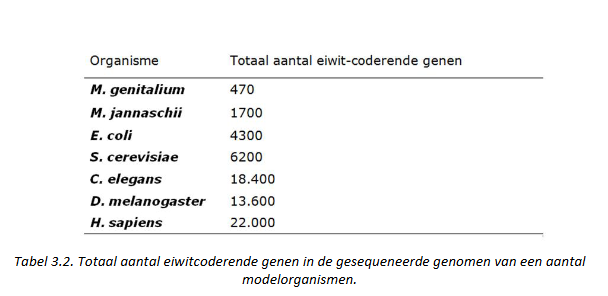
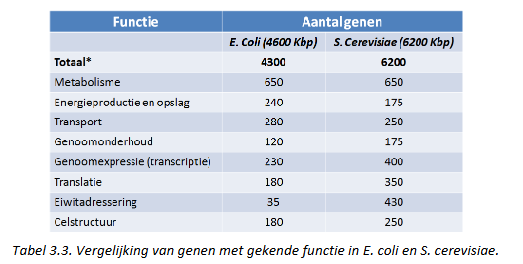
Variaties in genoomorganisatie: E. coli vs. S. cerevisiae
E. coli = bacterie → in darmflora van de mens
± 4.300 eiwit-coderende genen → meer dan 1/3 nog een totaal ongekende functie
S. cerevisiae (= bakkersgist) = eukaryoot
ééncellige, vrijlevende organismen
vrij compact genoom (in vgl met prokaryote genomen)
sterke uitbreiding van genen betrokken bij eiwitadressering (tov E. coli) → meer complexe opbouw van een eukaryote cel
functie van een groot deel van gecodeerde eiwitten ongekend
Variaties in genoomorganisatie: mycoplasma genitalium
= parasitair organisme → leeft in epitheelcellen van urogenitale & luchtweg kanalen
genoom is gereduceerd
slechts 470 gecodeerde eiwitten → sterke afhankelijkheid van gastheercel
kleinst gekende genoom
Variaties in genoomorganisatie: C. elegans & D. melanogaster
C. elegans (= worm) = multicellulair organisme
uit 959 lichaamscellen (& een variabel aantal gameten)
inzichten in regulatie van celcyclus & apoptose eerst in C. elegans bekomen
D. melanogaster (= fruitvlieg) = multicellulair organisme
studies met mutante fruitvliegen → op die manier genen identificeren die cruciale rol spelen
→ belangrijk voor studie van ontwikkelingsbiologie (want hebben gedetailleerde biologische & genetische kennis, relatief kleine genoom & snelle generatietijd)
Variaties in genoomorganisatie: modelorganismen
grote evolutieve afstand tussen modelorganismen & mens → MAAR: genoomcomplexiteit bij modelorganismen zijn niet zeer veel kleiner dan bij de mens
verschil: zeer sterke toename van eiwitten die een rol spelen bij regulatie van functies (bv. bij transcriptie & signaaloverdracht & communicatie tussen cellen)
verschil: opbouw van transcripten & eiwitten is meer complex
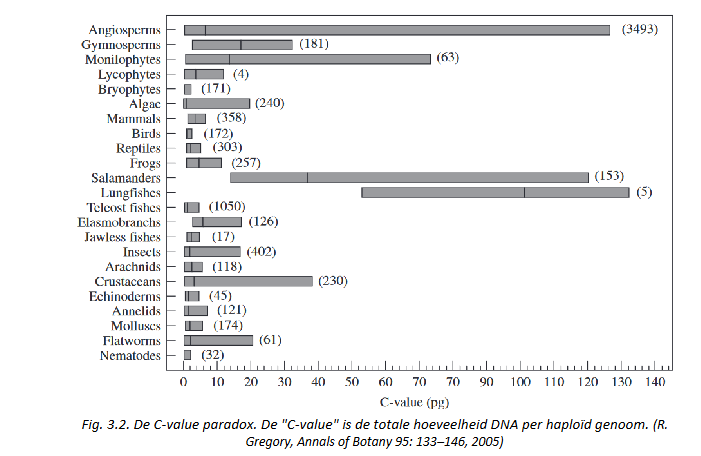
Eukaryote genomen bevatten veel repetitief DNA: C-value paradox
cellulaire DNA hoeveelheid correleert niet perfect met fylogenetische klassering
bv. menselijk genoom is 200x groter dan dat van gist, maar ook 200x kleiner dan dat van eencellige Amoeba dubia
C-value paradox = consequentie van aanwezigheid van repetitief, niet coderend DNA → hoeveelheid varieert sterk tussen organismen
Eukaryote genomen bevatten veel repetitief DNA: humaan genoom codeert minder dan 2% van DNA voor eiwitten
functie van 98% overblijvend DNA?
conservatieve definitie voor begrip gen = DNA sequentie die codeert voor een eiwit
MAAR: eukaryoot gen bevat ook stukken DNA die niet voor een eiwit coderen (intronen, UTRs, pseudogenen…)
→ nieuwe definitie van gen: DNA sequenties die coderen voor functionele RNAs
=> deze optellen bij eiwit-coderende gensequenties = ± 55% genen
Eukaryote genomen bevatten veel repetitief DNA: intergenische regio’s van het genoom
→ niet onder selectiedruk & mutaties in deze regio’s stapelen zich op & worden doorgegeven aan volgende generaties
intergenisch DNA: voor een groot deel uit gemiddeld- tot hoog-repetitief DNA (snel renaturerende DNA)
→ repetitieve sequenties worden opgedeeld in:
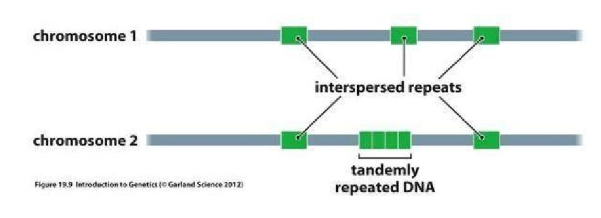
verspreid herhaalde sequenties (‘interspersed elements’)
tandem herhaalde sequenties (‘tandem repeats’)
uniek of low copy number DNA
‘spacer DNA’ tussen genen → daarin zitten regulatorische CIS-elementen
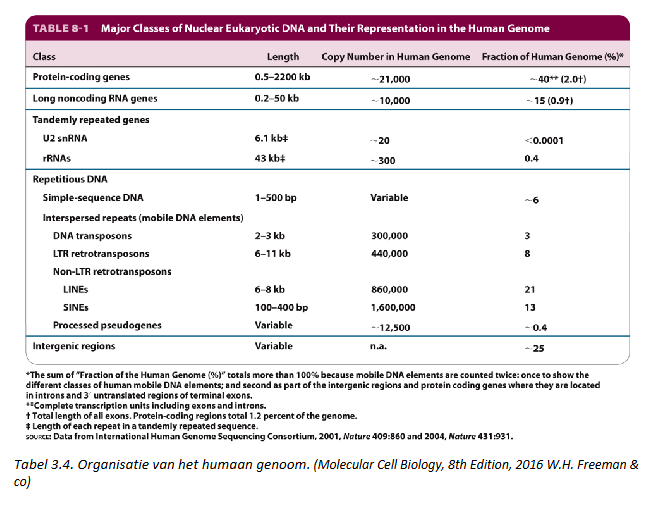
verspreid herhaalde sequenties (‘interspersed elements’)
verspreid over het genoom
gedegenereerde kopieën van transposeerbare elementen
die elementen (of kopieën) kunnen zich verplaatsen in het genoom
→ elementen kunnen zich vermeerderen & accumuleren in het genoom
transposities gebeuren zeer zelden, maar doorheen evolutie hebben transposeerbare elementen toch ± 40% van humaan genoom aangeleverd
correleert met gemiddeld repetitief DNA
verdere opdeling obv lengte:
LINEs (Long Interspersed Elements, 500 bp of meer)
SINEs (Short Interspersed Elements, <500 bp)
functie → niet gekend
mogelijk ‘selfish’ of ‘parasitair DNA’ → kan zichzelf dupliceren & op willekeurige wijze integreren in het genoom
dragen bij tot dynamiek van het genoom
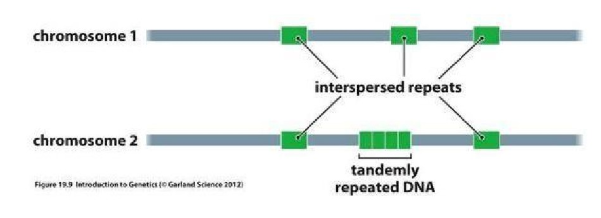
tandem herhaalde sequentie (‘tandem repeats’)
hoog-repetitief DNA
te onderscheiden van de rest van het genomisch DNA obv densiteit
heeft een gemiddeld lager GC gehalte = satelliet DNA
meestal in heterochromatine → thv centromeren & telomeren
1000en bp lang & onderverdeeld in 3 subcategorieën obv hun lengte:
satellieten
minisatellieten (of VNTRS = variable number tandem repeats)
15 tot 50 bp lange sequentiemotieven → totale lengte van tandem herhalingen: tussen 50 bp & 20 kb
microsatellieten (of STRs = ‘short tandem repeats’)
sequentiemotieven van 2 tot 6 bp → totale lengte tussen 50 & 500 bp
meest voorkomende microsatellieten: dinucleotide herhalingen (bv. (CA)n, komt 3 × 10^6 maal voor in het humaan genoom
genetische variatie tussen individuen in satelliet DNA: meestal gebaseerd op variatie in het aantal herhalingen, maar ook verschillen in sequentie kunnen optreden
binnen klasse van tandem repeats → voornamelijk VNTRs gekend omwille van gebruik bij forensische analyses
Extrachromosomaal DNA & horizontale gentransfer: horizontale of laterale gentransfer
= genomisch DNA van 1 organisme kan aan een ander doorgegeven worden
speciaal geval: horizontale gentransfer tussen endosymbiont & gastheer
heeft aanleiding gegeven tot organel-genomen die in eukaryote cellen teruggevonden worden & hun oorsprong als onafhankelijke vrijlevende cellen weerspiegelt
die extranucleaire genomen: relicten van prokaryote genomen van endosymbiontische bacterieën die partnerschap met eukaryote cel aangingen
→ bv. mitochondriën & chloroplasten (uit bacteriële endosymbionten → bezitten eigen genetische informatie)
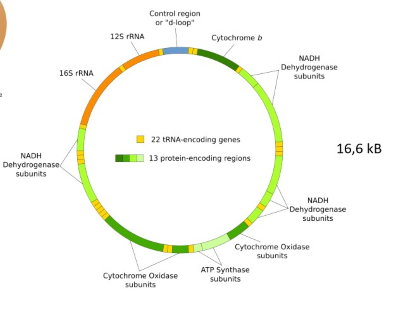
Extrachromosomaal DNA & horizontale gentransfer: mitochondriaal DNA/ mtDNA
bij de mens: 16.569 bp (0,0005% van totale genoom)
codeert voor beperkte set van mitochondriale functies
codeert voor essentiële enzymen betrokken bij ATP productie & aantal functionele RNAs (rRNA & tRNA)
overerving: niet-Mendeliaans, maar bijna uitsluitend via eicel (maternale overerving)
1 cel: meerdere duizenden exemplaren (meerdere mitochondria per cel, meerdere mtDNAs per mitochondrion)
heteroplasmie
mutaties in mitochondriaal DNA → leiden tot heterogeen genoom: zowel normale als mutante mtDNAs kunnen aanwezig zijn
circulair (zoals meeste bacteriële genomen)
bevat geen niet-coderend DNA
DNA sequentie van humaan mtDNA: sterk verwant aan genoom van endoparasiet Rickettsia prowazekii
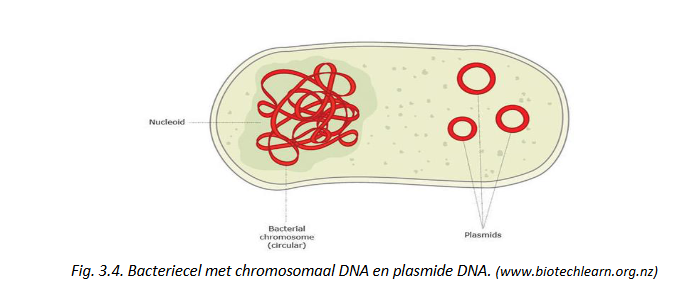
Extrachromosomaal DNA & horizontale gentransfer: extrachromosomaal DNA in prokaryoten
bv. 4.700 kb E. coli genoom
1 circulaire dubbelstrengige DNA molecule → 1 chromosoom
gecondenseerd tot nucleoïd
meeste prokaryoten bevatten ook plasmide DNA
circulair DNA met lengte tussen 2 & 100 kb
repliceren onafhankelijk van het chromosomaal DNA
bij celdeling: tenminste 1 kopie van plasmide doorgegeven aan dochtercellen
= soort parasitaire/ symbiontische relatie met gastheerbacterie
dragen resistentiegenen tegen antibiotica
zijn de ‘werkpaarden’ van de recombinante DNA technologie
Extrachromosomaal DNA & horizontale gentransfer: horizontale gentransfer bij prokaryoten
essentieel mechanisme van bacteriële evolutie
E. coli stam met gendefect waarbij hij aminozuur leucine niet kan synthetiseren zal afsterven in medium zonder dit aminozuur
stam met een gendefect voor methionine zal afsterven in medium zonder dit aminozuur
→ beide stammen apart laten groeien onder selectieve condities: ze sterven af
MAAR: culturen samenbrengen gedurende enkele uren (vooraleer dubbele selectiedruk toe te passen): ze groeien
=> verklaring: uitwisseling van genen die coderen voor de niet-gemuteerde enzymen
uitwisseling op verschillende manieren:
conjugatie
transformatie
transductie
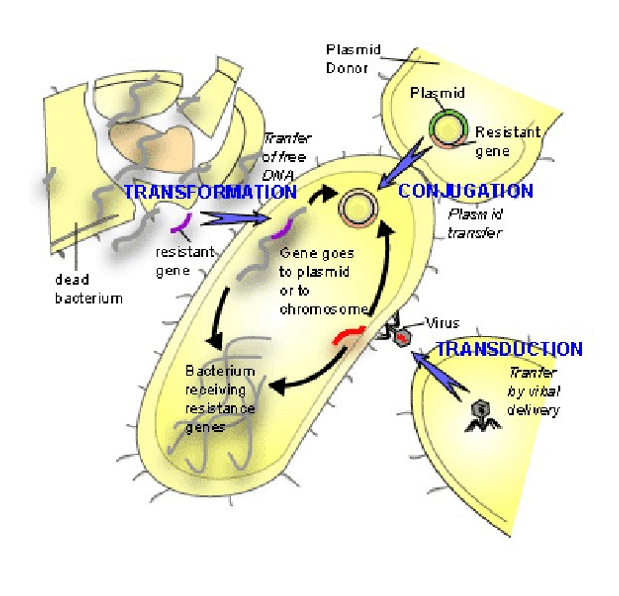
conjugatie
= bacterieel paren
kenmerk gecodeerd op plasmide (niet alle bacteriën hebben dit): F(ertiliteit)-plasmide
conjugatie uitsluitend tussen ‘mannelijke’ bacterie die plasmide bevat (F+ stam) → kan F-pilus vormen die contact maakt met ‘vrouwelijke’ F-bacterie
bij replicatie van plasmide: nieuwe replicon wordt doorheen de cytoplasmatische brug overgedragen op F- cel
F- cel krijgt daardoor zelf F+ fenotype
mechanisme laat weinig genetische variatie toe: enkel beperkte genenset op F-factor wordt overgedragen
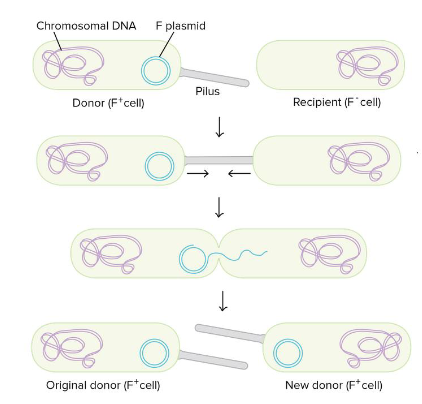
conjugatie: transfer van resistentiegenen via F-plasmide
plasmide kan (met zekere frequentie) door homologe recombinatie integreren in E. coli chromosoom
vorming van Hfr-cel (High frequency of recombination)
conjugatie nog steeds mogelijk
F-factor kan zich door excisie weer uit chromosoom verwijderen
→ proces gebeurt niet altijd correct
stukken chromosomen worden in F-factor geïntegreerd => F-plasmiden ontstaan met geïntegreerde genen afkomstig van verschillende bacteriën
bij voldoende selectie: °plasmiden die resistentiegenen (R-genen) dragen tegen meerdere antibiotica
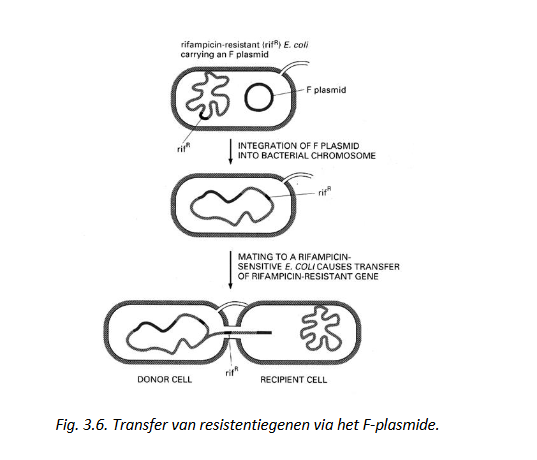
transformatie
vreemd DNA wordt rechtstreeks opgenomen uit omringend milieu
bv. DNA afkomstig van gelyseerde bacteriën (zie experiment van Griffith)
nadeel:
vrij DNA wordt snel afgebroken
zeer belangrijke routine praktijk bij recombinant DNA onderzoek
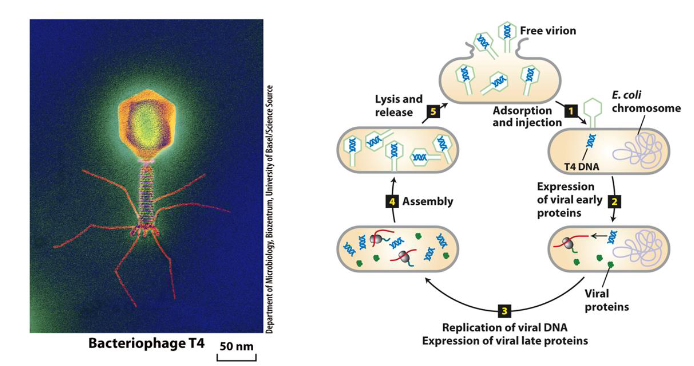
transductie
levenscyclus van een bacteriofaag
na binding op celmembraan: virus injecteert dubbelstrengig DNA genoom in gastcel
dan: transcriptie → aanmaak van enkele viraal-gecodeerde eiwitten, bv. kapseleiwitten
dan: replicatie → vermeerdering van genoom
gebruik makend van biochemische machinerie van gastheercel
na assemblage van viruspartikels: gastheercel barst open & nieuwe viruspartikels komen vrij
= lytische groeicyclus
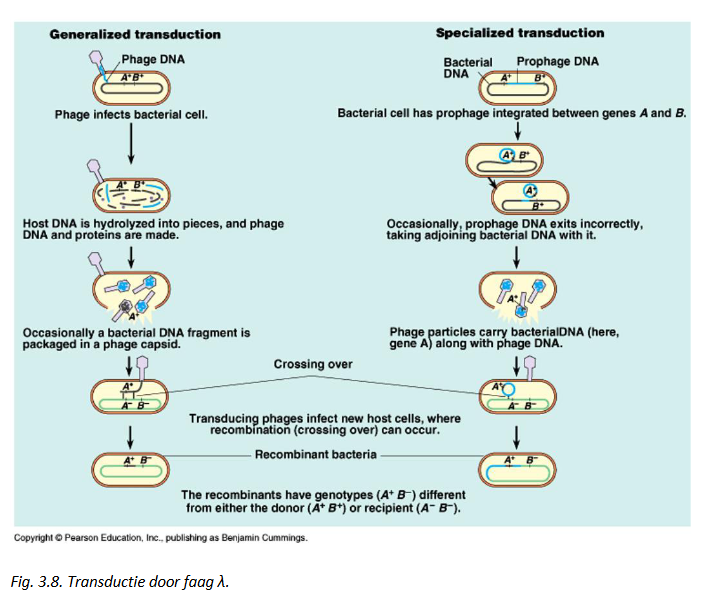
transductie → bacteriofaag λ
vaak gebruikt in moleculair biologisch onderzoek
uniek bij faag λ → 2 alternatieve ‘leefwijzen’:
klassieke lytische groeicyclus
honderden nieuwe viruspartikels per geïnfecteerde cel geproduceerd
faaginfectie zorgt ervoor dat bacterieel genoom in kleine fragmenten wordt verknipt
→ occasioneel kan bacterieel genomisch DNA fragment in viruspartikel worden verpakt & worden overgedragen naar andere bacteriecel = transductie
lysogene leefwijze
λ genoom integreert in E. coli chromosoom → vorming van pro-faag
λ genoom wordt stiekem mee vermenigvuldigd met DNA van waardcel
dan: milieuwijziging (bv. UV-licht): start programma op waarbij λ genoom weer uitsplitst → dan opstarten van lytische groeicyclus
→ integratie in bacterieel genoom: obv herkenning van specifieke chromosoomsequentie door integrase
→ (analoog aan excisie van F-factor uit chromosoom) geïntegreerde λ genoom kan foutief worden uitgeknipt → gastheer DNA wordt in viruspartikel mee getransporteerd naar andere cel = transductie
→ op analoge wijze kunnen eukaryote virussen gastheer DNA opnemen
samenvatting: horizontale gen-transfer bij prokaryoten
Genenclusters: solitaire genen & genduplicatie
± de helft van de humane genen zijn solitaire genen
solitaire genen: 1 kopij per genoom
genduplicatie: meerdere kopijen per genoom
zitten in multigen superfamilie, ontstaan door duplicatie uit ‘oergen’
homologe genen (paraloog/ ortholoog)
Genenclusters: bv. hemoglobine cluster
bij de vertebraten: tetramere eiwitten opgebouwd uit 2 α-type en 2 β-type globine polypeptiden
meerdere genen (& pseudogenen) voor α- en β-globines komen voor bij alle vertebraten
→ samen met genen voor myoglobine, voor subeenheden van intervertebrate globines & voor plant leghemoglobines = multigen superfamilie
= al die genen zijn door duplicatie ontstaan uit een ‘oer-gen’ = ancestrale gen
α- en β-globine genen komen gegroepeerd voor als genenclusters in ‘dichtbevolkte’ delen van genoom → weerspiegeling van evolutieve genexpansie
genenclusters: direct gevolg van mechanisme van genduplicatie, waarbij gedupliceerde genen naast elkaar komen te liggen
β-globine genduplicaties: van vrij recente oorsprong waardoor genen bij de mens nog samen op chromosoom 11 voorkomen & daardoor nog gemakkelijk als genfamilie kunnen herkend worden
α-globine genencluster: op chromosoom 16
functioneel & structureel nog duidelijk verwant aan β-globines, maar toch evolutief chromosomale scheiding opgetreden tussen genfamilies
→ chromosomen zijn evolutief fragiel & kunnen door translocaties, inversies, fusies & duplicaties variëren tussen soorten
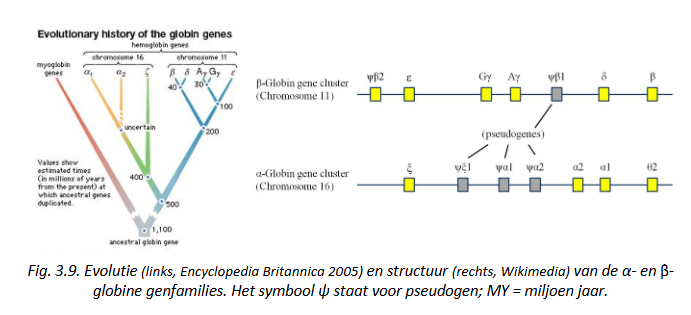
Genenclusters: genfragmenten & pseudogenen
→ als gevolg van genduplicatie
‘evolutief wrakhout’
pseudogenen = niet-functionele kopijen die nog duidelijke verwantschap vertonen aan een normaal gen
door mutaties na genduplicatie komen pseudogenen niet meer functioneel tot expressie
dragen bij tot ‘gene-pool’ (genenpoel) van soort (ook al zijn ze vanuit perspectief van individu waardeloos) → kunnen belangrijke rol spelen bij evolutie van soorten
Genenclusters: homologe genen/ paraloog/ ortholoog
homologe genen = genen die ontstaan door genduplicatie
paraloog → verwantschap tussen homologe genen binnen een species
ortholoog → verwantschap over species grenzen heen
Genenclusters: syntenie
= aantal chromosomen tussen soorten is verschillend, maar genenkoppeling blijft dikwijls behouden
tijdens evolutie van soorten: groepen van genen kunnen door chromosomale translocatie verplaatst worden binnen het genoom
vergelijking van genoomsequenties: verwantschap traceren
bv. tussen mens & muis: 183 syntenische genoomsegmenten (soms nog zeer groot) → bv. volledige humane chromosomen 17 & 20 verwant aan segmenten op chromosomen 13 & 2 bij de muis
Genenclusters: drijvende kracht achter toename van aantal genen + voordelen genduplicatie
= nood aan voldoende eiwit/ RNA productie
bv. bij mitose: zeer snel grote hoeveelheden histonen of ribosomale RNAs aanmaken door elke dochtercel om DNA condensatie & eiwitsynthese toe te laten
bv. duplicatie van interferon-genen: selectief voordeel bij antivirale afweer
→ dergelijke genenclusters kunnen zeer uitgebreid zijn: uit tientallen tot meer dan duizend genen & pseudogenen
bv. voor olfactorische genfamilie die codeert voor serpentine receptoren betrokken bij waarnemen van geuren
na genduplicaties: functionele verschillen in gecodeerde eiwitten door accumulatie van allerlei mutaties
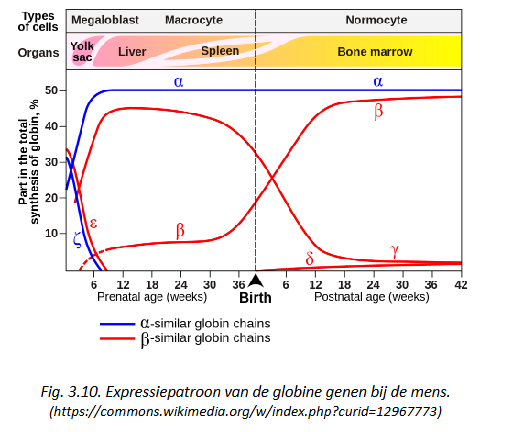
bv. bij globines: verschillende O2-affiniteit & complementaire, gespecialiseerde functies
embryonaal hemoglobine is ƺ2ε2 → vanaf tiental weken zwangerschap: expressie van de α- en γ-globines aangeschakeld met vorming van α2γ2 hemoglobine, blijft aanwezig gedurende merendeel van foetale leven
kort na de geboorte: γ-globines vervangen
door β-globine → vorming van het adulte α2β2 hemoglobine
δ-globine gen slechts zeer zwak tot expressie
→ het interessante hierbij: foetaal globine is efficient in O2 opname, zodat ter hoogte van de placenta O2 kan onttrokken worden aan het bloed van de moeder
Het menselijk genoom project
meeste fenotypische kenmerken (incl. veel ziektebeelden) → terug te brengen tot uitdrukking van genetisch materiaal
→ beschikbaarheid van volledige DNA sequentie van menselijk genoom is een schat aan informatie
internationaal gecoördineerd onderzoeksprogramma: opgestart in ‘90
in 2001: eerste ‘klad’ versie van volledige genoom van de mens gepubliceerd
2 onderzoeken op hetzelfde moment:
Human Genome Sequencing Consortium → in Nature gepubliceerd
bedrijf Celera Genomics → in Science gepubliceerd
vanaf dan: niet meer reductionistische benadering, maar wel nieuwe methoden die holistische analyse toelaten
simultaan-analysen van grote populaties DNA of RNA moleculen, van eiwitten of van metabolieten: mogelijk door verregaande automatisering, robotica & miniaturisatie, gekoppeld aan efficiënte dataverwerking
door functioneel onderzoek: tal van ongekende genetische elementen beschikbaar geworden, ook structuren van genen & van gecodeerde eiwitten, incl. de controlesystemen die expressie ervan bepalen
→ biedt mogelijkheden om nieuwe fundamentele inzichten te verwerven in replicatie- & transcriptieprocessen & complexe biologische systemen
functional genomics: precieze functies van alle genen bepalen → in de toekomst
vergelijking van sequenties binnen menselijk genoom met deze van andere organismen: bieden inzicht in functie van een bepaald ‘nieuw’ gen
comparative genomics: sequenties met elkaar vergelijken
gedetailleerd inzicht in evolutiemechanismen & in genoomdynamiek
Polymorfisme binnen het menselijk genoom
tussen 2 personen: ± 3 × 10^6 genetische verschillen
2 willekeurige individuen zijn voor ± 99,9% genetisch identiek → inclusief rassenverschillen
variaties kunnen leiden tot verschillen in eiwitten & in expressiepatronen ervan → liggen aan de basis van fenotypische verschillen tussen individuën (incl. ziektebeelden of verschillend gedrag op medicaties)
binnen volledige populatie: 20% van alle menselijke eiwitten zijn in meer of mindere mate polymorf
= in meer dan 1 vorm voorkomend
voor veel complexe kenmerken (bv. intelligentie) spelen ook omgevingsfactoren een grote rol
Polymorfisme binnen het menselijk genoom: vergelijkende analyse van genoomsequenties van 1.092 mensen uit 14 verschillende populaties in 2012 (www.1000genomes.org)
polymorfismen dikwijls beperkt tot 1 nucleotide → SNP’s (= snips, single nucleotide polymorphisms)
individuele nucleotiden binnen genoom waarvan 2 of meer variaties bestaan → komen binnen populatie voor aan een frequentie > 1%
± 3 miljoen SNP’s herkend in sequenering van het menselijk genoom = ± 1 per 1.000 bp
zeer nuttig voor medische genetica, kunnen helpen bij identificatie van ± 4000 ziektebeelden met gekende genetische achtergrond
waarschijnlijk slechts een kleine proportie van SNP’s functioneel relevant → MAAR: leidt toch tot structurele diversiteit van duizenden eiwitten
mogelijkheid tot koppeling tussen SNP’s & andere mutaties die wel betrokken zijn bij genetische ziekten
farmaco-genomics
snel groeiend onderzoeksgebied
om na te gaan hoe de relatie is tussen genetische verschillen & effectiviteit en toxiciteit van geneesmiddelen
genotypering kan snel gebeuren & meeste genetische info is bekend → farmaceutische industrie streeft naar: geneesmiddelen op maat
geneesmiddelen werken vaak in op eiwitten: receptoren & liganden, enzymen enz. → efficientie waarin interactie beïnvloed wordt, hangt af van precieze aminozuursamenstelling van de eiwitten
genetische variatie = verschillen in eiwitten = verschillende binding van geneesmiddelen
grotere variaties (deleties, inserties) = Copy Number Variants
VNTRs = variable number tandem repeats → tandemherhalingen waar sequentie in een populatie gelijk is, maar het aantal keer dat ze voorkomt is verschillend
meest bestudeerde polymorfe gen-familie: HLA systeem (Humaan Leucocytair Antigen systeem) = histocompatibiliteitscomplex
membraangebonden glycoproteïnen aanwezig op alle cellen
per locus: tot 75 verschillende allelen
typering van HLA-type: belangrijk voor transplantaties
HLA-matching = aantal verschillen zo klein mogelijk houden, om afstoting te onderdrukken
DNA fingerprinting → gebuikt polymorfismen in VNTRs
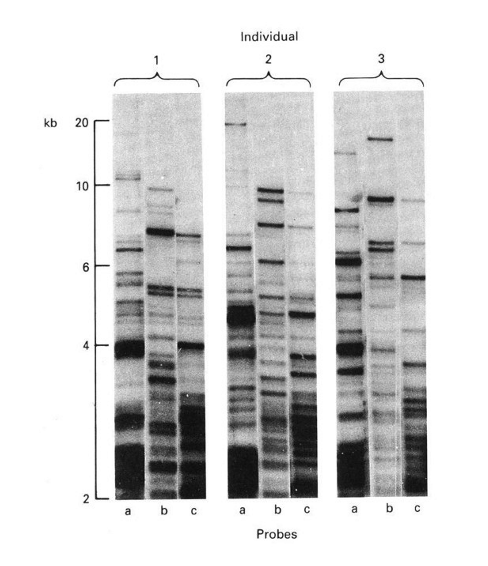
Polymorfisme binnen het menselijk genoom: DNA fingerprinting
gebruikt bij forensisch onderzoek
gebruik makend van polymorfismen in VNTRs
genomisch DNA isoleren van verschillende individuen waarvan men het genoom wenst te vergelijken (bv. uit bloed)
DNA wordt via enzymatische behandeling in kleine stukjes verknipt & op grootte gescheiden via agarose gelelektroforese
DNA denatureren (en probe ook) → probe gaat hybridiseren op plaats waar die het complement herkent
DNA fragmenten van gel worden via Southern blotting overgebracht op nylon membraan
membraan wordt geïncubeerd met ‘radioactief’ gemerkte probe die interageert met een bepaalde VNTR sequentie
probe zo maken dat die complementair is aan het stuk dat je wilt onderzoeken
detectie van probe → via autoradiografie (of andere niet-radioactieve methodes)
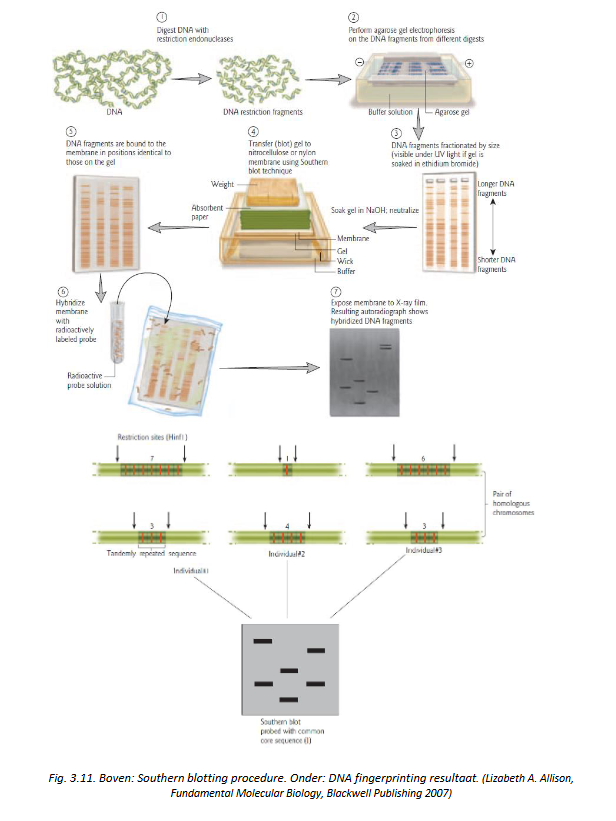
Southern blotting
stukje DNA dat je wilt onderzoeken → DNA enzymatisch kapot knippen in kleine fragmenten
fragmenten scheiden op een gel → poreuze matrix waardoor moleculen kunnen migreren
grote moleculen: migreren traag
kleine moleculen: migreren snel
→ mengsel van DNA scheiden obv hun grootte
gel in basische oplossing steken (buffer) → DNA gaat denatureren
op de gel: nitrocellulose membraan leggen, daarboven papieren doekjes
buffer gaat doorheen gel naar boven, op de doekjes → gaat het DNA meenemen & dit transfereren op het membraan
gebeurt zo omdat dat makkelijker is om mee te werken (gewoon gel is papperig en niet handig) → DNA naar werkbaar formaat overbrengen
probe toevoegen op de filter
probe radioactief om te kunnen detecteren (vandaag meer fluorescente probe)
probe bindt op het complement
dan film erop leggen → energie die in de probe zit gaat de film belichten
=> enkel op die plaats zal je een bandje zien
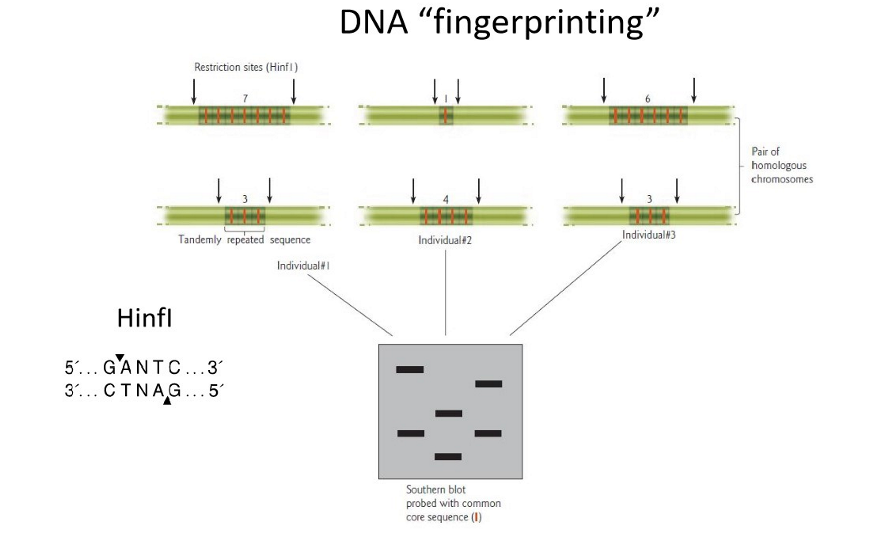
voorbeeld DNA fingerprinting
3 individuen
kijken naar VNTRs → naar bepaalde locus waar VNTR zit
elk individu: 2 allelen → daarin ook variatie in aantal herhalingen
enzyme gebruiken dat sequentiespecifiek knipt → restrictie-enzym Hinf1
sequentie: GANTC
verschillende grootte van fragmenten
DNA fragmenten scheiden op een gel
gedetecteerd met de Southern blotting
Genoomsequenties van hominiden (grote apen)
bij de hominiden: chimpansee & bonobo meest verwant aan de mens
afsplitsing van gemeenschappelijke voorouder ± 6 miljoen jaar geleden
alle grote mensapen: 1 extra paar chromosomen in vgl met de mens
tijdens evolutie naar de mens: fusie van 2 chromosomen thv telomeren (chromosoomuiteinden) → vorming van grote chromosoom 2
bij grote apen (chimpansees, bonobo’s, gorilla’s…) → chromosomen 2A & 2B
genoomsequenering van bonobo in 2012: 99,6% DNA sequentie identiteit met chimpansee, 98,7% met de mens
± 35 miljoen SNP’s, 5 miljoen kleine ‘indels’ (inserties/ deleties) & meerdere lokale chromosoom-rearrangementen
daartussen: genetische kenmerken die unieke plaats van de mens in evolutie bepalen
genetische variatie bij de mens: veel kleiner dan bij de chimpansee
menselijke populatie → heeft ‘bottleneck’ situatie meegemaakt
populatie tijdens de ‘Out of Africa’ migratie naar Europa gereduceerd tot minder dan 10.000 individuen
genoom van Neanderthaler bepaald in 2010: ± 99,8% identiek aan ons genoom
migreerde Noordwaarts vanuit Afrika tussen 400.000 & 800.000 jaar geleden, vestigde zich in grote delen van Europa, Midden-Oosten & Centraal Azië
± 30.000 jaar geleden uitgestorven
tussen 1 & 4% Neanderthal-karakteristieke sequenties terug te vinden bij alle mensenrassen (behalve van sub-Sahara populaties) => beperkte kruising tussen Homo sapiens & Homo neanderthalensis na migratie van Homo sapiens vanuit Afrika, ongeveer 60.000 jaar terug
in 2008 in Denisova grot in Siberië: resten opgegraven van een meisje
genoomsequentie in 2012 gepubliceerd: zowel verschillend van Homo neanderthalensis als van homo sapiens
wel tussen 4 & 6% van genetisch materiaal van homo denisova terug bij Melanesiërs => ook ‘interbreeding’
dit DNA niet teruggevonden in Chinese populatie (nochtans veel dichter bij Denisova mens geleefd)
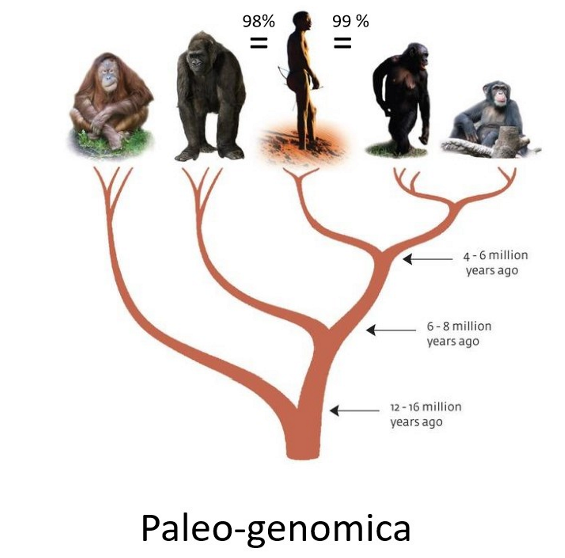
Condensatie van het genoom:
prokaryoot (bv. E. coli): moet 1 mm DNA passen in een cel met lengte van ± 2 µm
bij de mens: 2 m DNA verpakken in een cel van ± 10 µm
DNA neemt groot volume in door onderlinge afstoting van negatieve ladingen
→ hoe kunnen lange DNA moleculen verpakt worden in cellen?
diversiteit in aantal chromosomen van verschillende eukaryote organismen → MAAR: hetzelfde mechanisme om genoom te verpakken
DNA molecule om histoneiwitten gewonden = vorming van nucleosomen
nucleosomen worden in verschillende stappen gecondenseerd → uiteindelijke vorming chromosoom
Condensatie van het genoom: DNA condensatie
19e eeuw: Friedrich Miescher toonde aan dat DNA voorkomt in associatie met eiwitten (nucleïne: uit 1/3 DNA & 2/3 eiwit)
in 1970: aangetoond dat celkernen kleine basische eiwitten bevatten → interageren met DNA
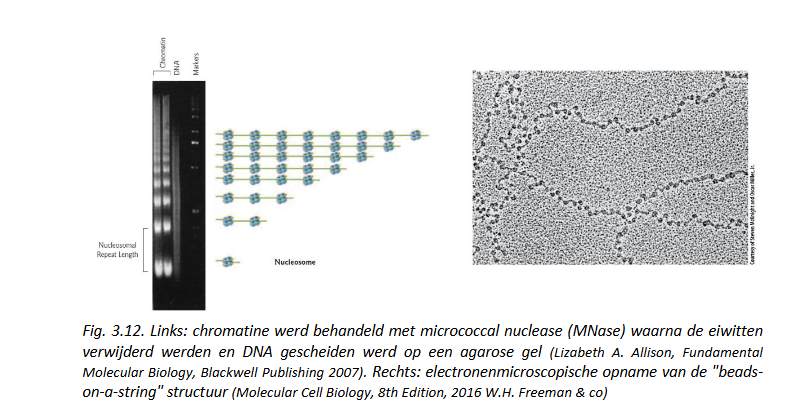
→ eerste inzichten van DNA condensatie: toevallige ontdekkingen (= serendipity)
bv. dmv nuclease experimenten (uitgevoerd met een ander doel): ontdekt dat DNA in chromatine degradeert met vorming van aantal discrete fragmenten die 180 bp in grootte verschillen => regelmatig verknippingspatroon ontstaat doordat DNA om architecturale eiwitten gewonden is die niet vatbaar zijn voor verknipping door micrococcal nuclease (MNase)
resultaten van MNase experimenten: in overeenstemming met electronenmicroscopische opnamen van chromatine uit 1975 → streng met daarop partikels met uniforme grootte in repeterend patroon = parelsnoer/ beads-on-a-string structuur
beads: complex van DNA & histonen
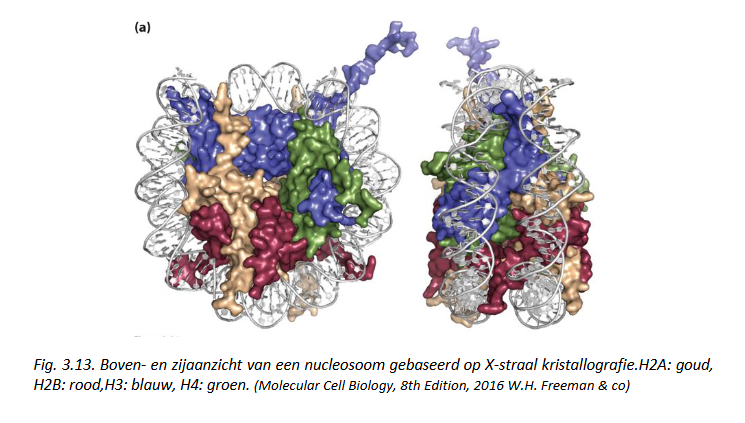
Condensatie van het genoom: histonen
kleine eiwitten met molecuulgewicht tussen 10 & 20 kDa
rijk aan arginines & lysines (positief geladen AZ) → basisch karakter
2 soorten:
kern histonen/ core histones (H2A, H2B, H3 & H4)
behoren tot traagst evoluerende eiwitten
H4 van de koe verschilt bv. maar in 2 AZ van erwt histon H4
vormen octameer met 2 moleculen H2A, H2B, H3 & H4 → vormen spoel waarrond DNA gewonden wordt
negatief geladen DNA maakt 2 windingen van samen 146 bp rond positief geladen histon octameer
kern histon octameer + linker DNA (niet om histonen gewonden) = nucleosoom → bevat 180 bp DNA
hebben geladen N-termini (aanrijking van lysine residuen) = histonstaarten
doelwit van post-translationele modificaties (PTMs)
linker histonen (bv. H1)
Condensatie van het genoom: rol van histonen
→ tweeledig & paradoxaal
condensatie van DNA in een stabiele & compacte structuur
vervullen belangrijke controlefuncties → toegankelijkheid van welbepaalde DNA segmenten voor transcriptionele machinerie
Condensatie van het genoom: histon acetylasen
histon-modificerende enzymen
brengen acetylgroepen aan op positief geladen lysine zijgroepen van histoneiwitten → beïnvloeden zo de interactie tussen de histonen & DNA en andere eiwitten → bepalen de graad van chromatine condensatie
acetylaties: gecontroleerd proces, belangrijke rol bij transcriptie-regulatie
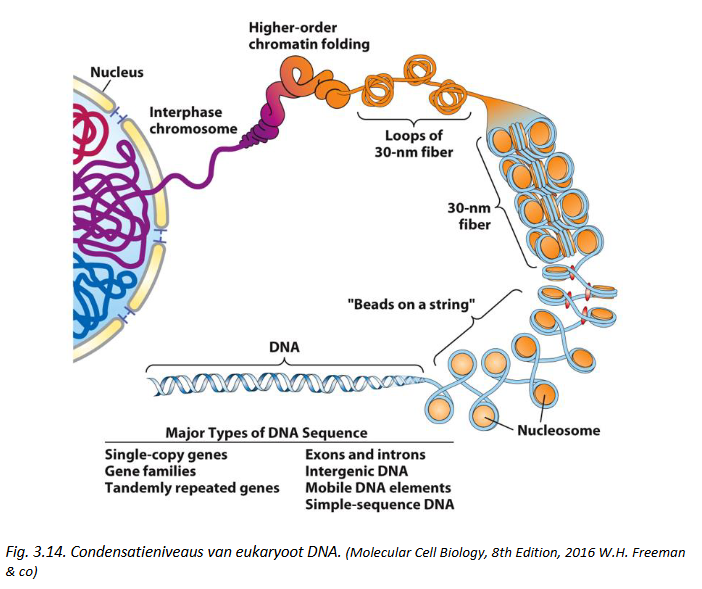
Condensatie van het genoom: solenoïde - DNA lussen - chromosomen
beads-on-a-string structuur → enkel zichtbaar bij niet-fysiologische lage zout concentratie (< 10 mM)
chromatine in situ: nucleosomen zijn georganiseerd in een compacte solenoïde vezel met diameter van ± 30 nm
H1 linker histon stabiliseert de solenoïde structuur (zit niet in nucleosoom)
solenoïde vezel verder gecondenseerd door bevestiging aan een ‘scaffold’/ eiwitskelet → °lussen van 50-100 kB DNA
DNA lussen verder opgewonden in gespiraliseerde structuur van 600 nm
dan ingepakt in chromosomen met diameter van ± 1400 nm per arm
→ in totaal: 10.000-voudige condensatiefactor
Condensatie van het genoom: celcyclus
elk chromosoom: ± 100 Mb DNA
DNA eenheden kunnen worden gekleurd met kleurstoffen en zichtbaar gemaakt via lichtmicroscopie
voorkomen van DNA: afhankelijk van celcyclus
tijdens mitose (metafase): individuele chromosomen zichtbaar
metafase = korte periode van de M-fase in de celcyclus
DNA zeer sterk gecondenseerd → geen of nauwelijks transcriptie
karyotypering → beschrijven van morfologie van chromosomen-set: aantal, voorkomen van translocaties, deleties…
tijdens interfase: DNA onder vorm van chromatine-draden, in verschillende graden van condensering
heterochromatine: zeer sterk gecondenseerd
meest markante vorm: Barr-lichaampje = volledig gecondenseerd X-chromosoom, komt voor in alle vrouwelijke cellen
transcriptioneel niet actief
euchromatine: zeer zwak gecondenseerd
electronenmicroscopisch: zichtbaar als parelsnoer-structuur
meestal transcriptioneel actief
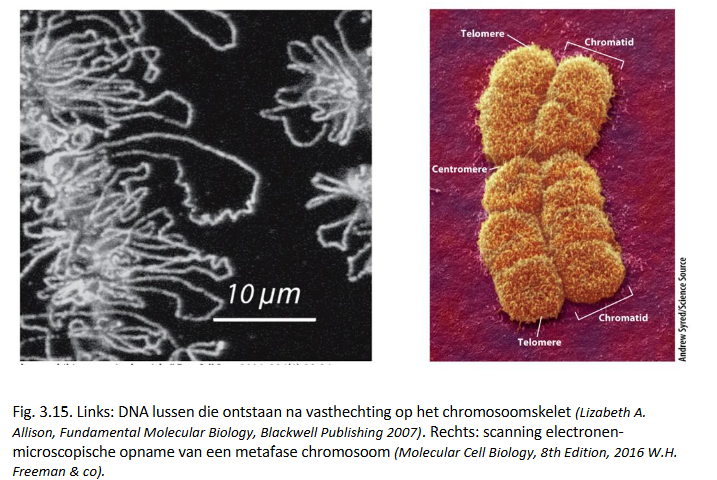
samenvatting: stapsgewijze condensatie van het eukaryoot genoom
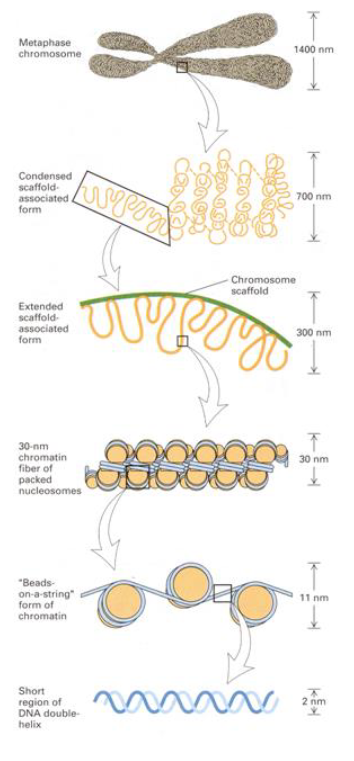
Test jezelf H3

DNA replicatie is semi-conservatief: 3 mogelijke modellen voor DNA replicatie
semi-conservatieve replicatie
elke nieuwe DNA molecule wordt gemaakt uit 1 originele/ parentale streng & 1 nieuwe/ dochterstreng
conservatieve replicatie
nieuwe helix altijd uit 2 dochter strengen
parentale helix zou geconserveerd blijven na replicatie
dispersieve replicatie
sommige delen van originele helix blijven bewaard, andere niet
→ juiste model in 1958 aangetoond door Matthew Meselson & Franklin Stahl
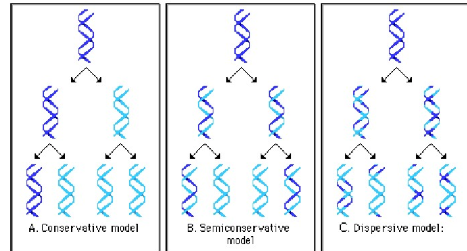
DNA replicatie is semi-conservatief: experiment van Matthew Meselson & Franklin Stahl
experiment gebruik makend van isotopen & densiteitsgradiënt centrifugatie
E. coli laten groeien in medium met 15N = zware isotoop van stikstof, 1 neutron meer dan 14N
15N is stabiel & niet-radioactief
bacteriën werden in verschillende generaties in ‘zware’ medium gegroeid → E. coli DNA werd denser omdat stikstofhoudende basen het 15N hadden ingebouwd
→ na verschillende generaties in zware medium: bacteriën overgebracht naar medium met 14N
dan densiteit van bacterieel DNA op einde van 15N cultuur (generatie 0) vergelijken met opeenvolgende generaties bij groei in 14N
om densiteit van DNA te bepalen: densiteitsgradiënt centrifugatie gebruiken
DNA samenbrengen met CsCl (= zout van zware metaal cesium)
in een tube in oplossing brengen & gedurende uren op hoog toerental afdraaien in een ultracentrifuge
→ uiteindelijk gradiënt vormen met hoge concentratie CsCl op bodem van tube & lage concentratie aan de top
→ DNA zal in gradiënt migreren naar positie waar densiteit gelijk is aan deze van gradiënt
DNA detecteren door tubes te belichten met UV (260 nm)
na 1 replicatiecyclus in 14N medium: DNA heeft intermediaire densiteit (14N + 15N)
na 2e replicatiecyclus: DNA met intermediaire densiteit + DNA met lichte (14N) densiteit
in verdere generaties: intermediaire band steeds meer uitgedund
→ conclusie: DNA replicatie is semi-conservatief
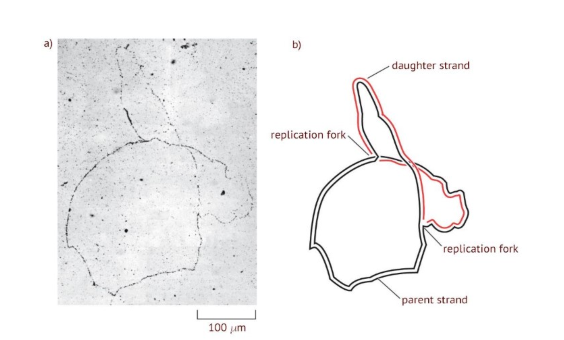
DNA replicatie gebeurt meestal bidirectioneel: 1963 John Cairns
toonde semiconservatieve DNA replicatie visueel aan via autoradiografie van 3H-thymidine gemerkt E. coli DNA gecombineerd met elektronenmicroscopie
extra ontdekking: bacterieel DNA vertoont 3 lussen → door 2 zogenaamde replicatievorken
lussen: ontstaan wanneer circulaire DNA zicht opent bij replicatie
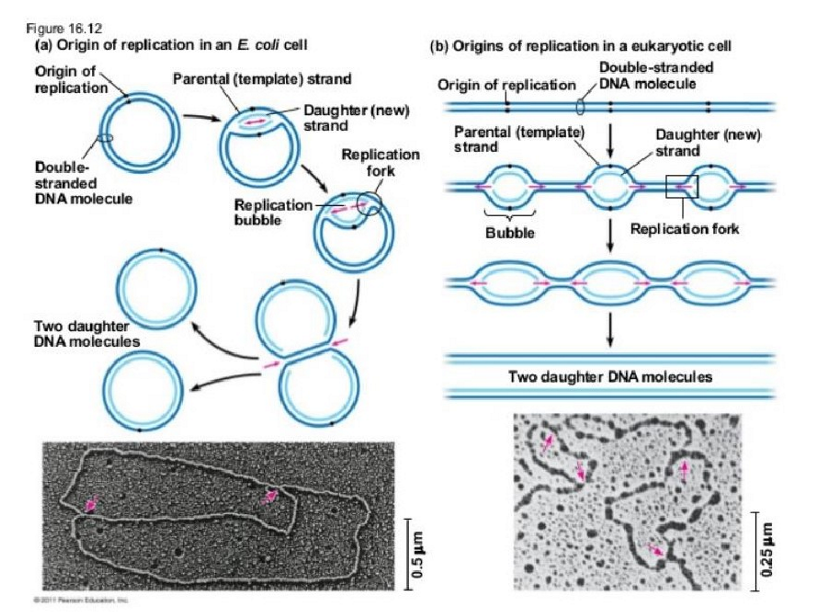
DNA replicatie gebeurt meestal bidirectioneel: origin of replication (ori)
= welbepaalde plaats op het chromosoom, waar de replicatie start
replicatie gebeurt bidirectioneel
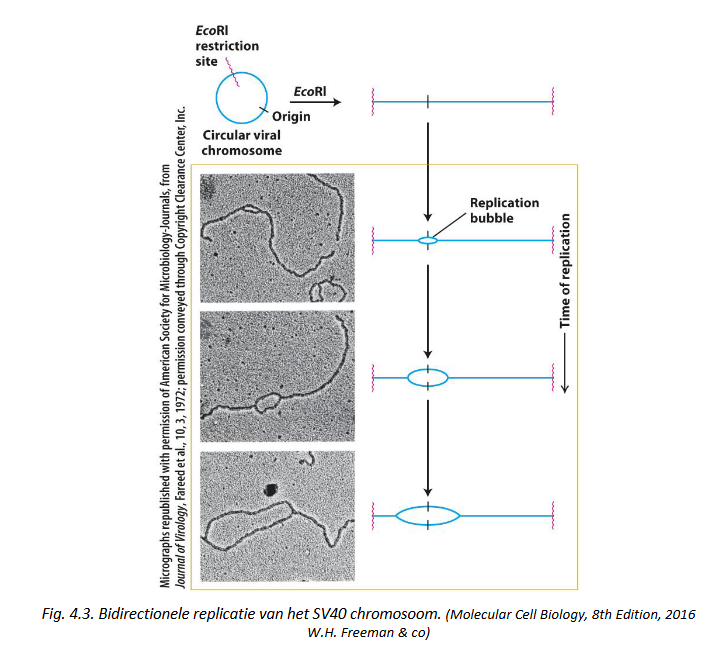
DNA replicatie gebeurt meestal bidirectioneel: experiment voor aantonen bidirectionaliteit
circulair viraal chromosoom gelineariseerd → door verknipping met EcoRI enzyme
EcoRI enzyme = endonuclease → verknipt DNA sequentiespeciek
gebruikte DNA bevatte slechts 1 knipplaats voor EcoRI enzyme per molecule
gaf mogelijkheid om afstand van replicatiebellen tot EcoRI knipplaats te bepalen & dit in identieke DNA moleculen in verschillende stadia van replicatiecyclus
DNA gevisualiseerd via elektronenmicroscopie
→ observatie: afstand van midden van replicatiebel tot uiteinden van gelineariseerde DNA strengen is gelijk, ongeacht replicatiestadium waarin DNA molecule zich bevindt
→ conclusie: replicatie start van in het midden van replicatiebel (origin of replication) & van daaruit in 2 richtingen (bidirectioneel)
DNA replicatie gebeurt meestal bidirectioneel: uitzonderingen
bidirectionele replicatie is de standaard bij zowel pro- als eukaryoten (enkele uitzonderingen op de regel)
E. coli chromosomen hebben slechts 1 ori, menselijke chromosomen hebben meerdere ori’s & vertonen ook meerdere replicatiebellen
DNA replicatie gebeurt meestal bidirectioneel: replicon
= DNA segment waarvan replicatie gestuurd wordt vanuit 1 ori
duur van replicatiecyclus verschilt tussen E. coli & mens
E. coli genoom: grootte van 4,6 ×106 bp → bidirectioneel gerepliceerd in ± 40 min vanuit 1 ori (=> snelheid van ± 1000 bp per seconde per vork)
menselijk genoom: grootte van 3 × 109 bp → ± 8 uur
± 10.000-100.000 groeivorken, maar meeste werken gedurende relatief korte tijd
± 100 bp per seconde per vork gesynthetiseerd → tragere synthesesnelheid is wellicht gevolg van complexere chromatinestructuur bij eukaryoten
DNA replicatie gebeurt meestal bidirectioneel: prokaryote & eukaryote ori’s
verschillende gelijkenissen:
unieke sequenties, met meerdere gerepeteerde subdomeinen
repeats zijn herkenningsplaatsen voor eiwitten
aantal van sequenties zijn goed gekend (N staat voor om het even welk van de 4 nucleotiden)
ori’s bijna altijd geflankeerd door A/T rijke gebieden
A/T-rijke gebieden: makkelijker open te maken, want hebben maar 2 H-bruggen onderling

DNA polymerasen katalyseren DNA synthese van 5’ naar 3’: DNA polymerasen
= enzymen die nucleotiden polymeriseren in een groeiende DNA keten
basiskenmerk: kunnen DNA synthese niet de novo beginnen → hebben een primer nodig
primer: kort stukje RNA dat op DNA matrijs (‘template’) wordt gesynthetiseerd
DNA polymerasen herkennen & binden de vrije 3’ hydroxylgroep (3’-OH) op einde van primer
eens geprimed: DNA polymerasen kunnen zeer snel & met hoge getrouwheid (fidelity) een bestaande DNA keten verlengen
soorten polymerasen:
bacteriële polymerasen → 500 nucleotide per seconde toevoegen
zoogdier DNA polymerasen → snelheid van 50 nucleotide per seconde
basiskenmerk: nucleotiden enkel 1 per 1 toevoegen & enkel in 5’ → 3’ richting
bij elke ingebouwde nucleotide: pyrofosfaatgroep afgesplitst
door werking van een pyrofosfatase → reactie wordt irreversibel
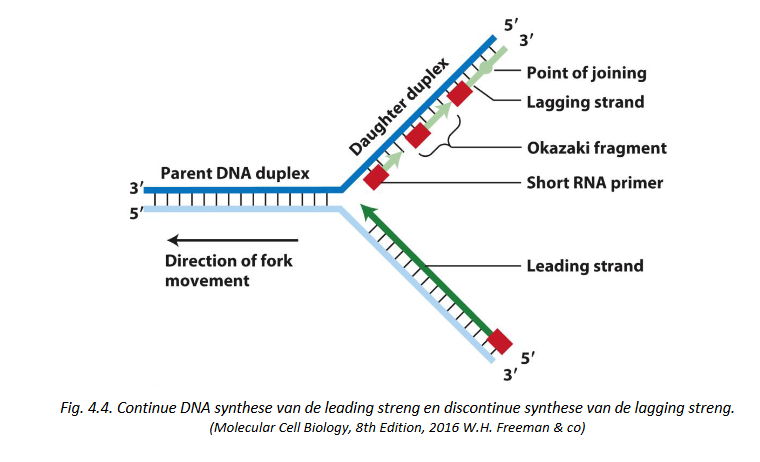
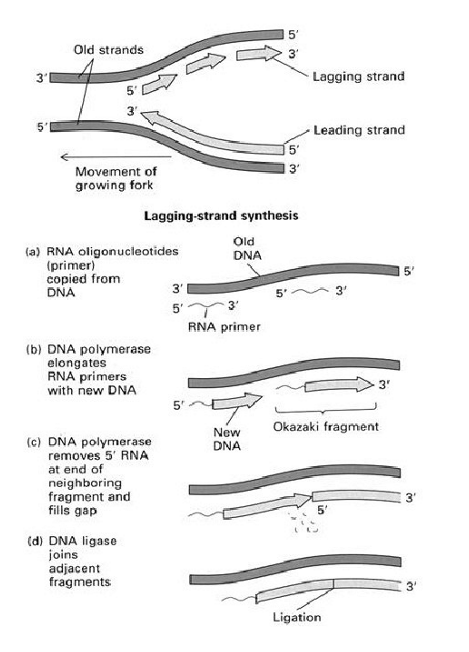
DNA polymerasen katalyseren DNA synthese van 5’ naar 3’: semi-discontinue replicatie
DNA helix verloopt antiparallel => beide strengen kunnen niet in dezelfde richting worden gerepliceerd → DNA repliceren via semi-discontinue replicatie
beide strengen worden van 5’ → 3’ aangemaakt, maar via verschillende mechanismen:
1 streng continu aangemaakt
1 streng in korte fragmenten gesynthetiseerd (discontinu)
DNA polymerasen katalyseren DNA synthese van 5’ naar 3’: ‘leading’ template & ‘lagging’ template
replicatie in ori:
continue 5’→3’ replicatie op ‘leading’ template streng
op ‘lagging’ template streng: replicatie niet continu door de directionaliteit van DNA polymerase
lagging streng wordt in korte fragmenten gekopieerd = Okazaki fragmenten
Okazaki in bacteriën langer dan in eukaryoten
→ hoewel synthese van lagging streng een aantal extra bewerkingen vergt tov deze van de leading streng worden beide strengen gelijktijdig & met dezelfde snelheid verlengd door 2 DNA polymerasen , 1 voor elke streng
overzicht DNA replicatie
multi-eiwit moleculaire machines verzorgen DNA replicatie: inzichten over mechanismen van DNA replicatie
uit studie van E. coli
eukaryote replicatie: voornamelijk bestudeerd door gebruik te maken van simian virus 40 (SV40) → repliceert in eukaryote cellen
SV40: klein genoom van ± 5 kb & gebruikt eigen multifunctionele eiwit (virale T antigen) gecombineerd met gastheereiwitten voor replicatie
fundamentele kenmerken van replicatie zijn geconserveerd van E. coli tot de mens → verschillen zitten in de details
DNA replicatie → samenwerking van een heleboel eiwitten
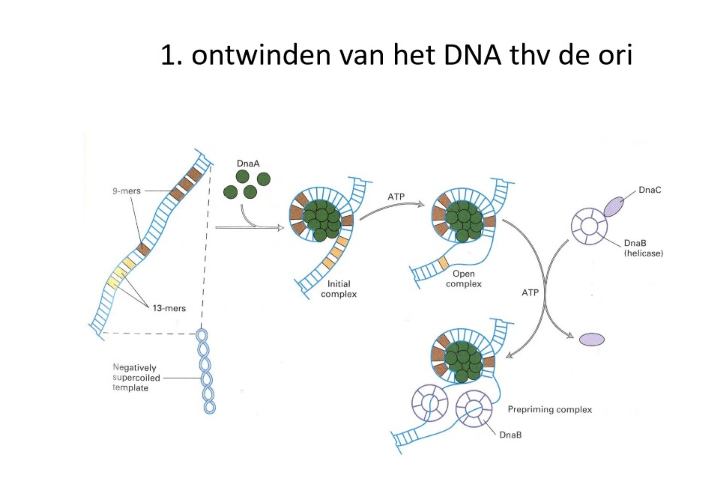
multi-eiwit moleculaire machines verzorgen DNA replicatie: DnaA eiwitten
initiëren replicatie in E. coli
verbruik van ATP → ontwinding van lokale A/T-rijke DNA structuur
vorming van open complex
multi-eiwit moleculaire machines verzorgen DNA replicatie: helicase (DnaB)
ontwindt duplex DNA
DnaB bindt als ringvormige klem op beide DNA strengen in het ‘open complex’
ATP nodig
°pre-priming complex
helicase schuift over DNA streng in 5’→3’ richting & veroorzaakt lokale strengscheiding met verbruik van ATP
als gevolg van klemstructuur zal het slechts loskomen op 3’ uiteinde van DNA streng
multi-eiwit moleculaire machines verzorgen DNA replicatie: single-strand binding
= eiwitten die de ontwonden structuur stabiliseren
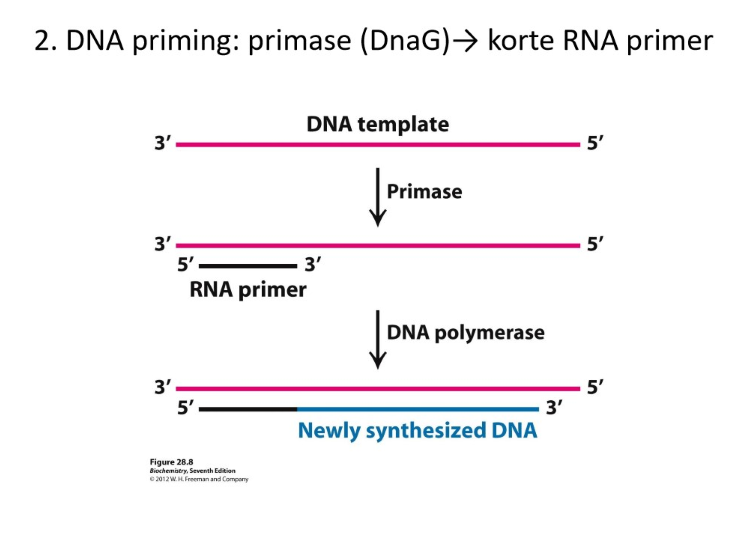
multi-eiwit moleculaire machines verzorgen DNA replicatie: primase (DnaG)
katalyseert synthese van korte RNA primers
= RNA-polymerase
werkt niet thv specifieke sequenties
vorming van mobiele primosoom
gesynthetiseerde RNA primers zijn kort
multi-eiwit moleculaire machines verzorgen DNA replicatie: polymerase I
verwijdert de RNA primers & vervangt deze door DNA
speelt ook een belangrijke rol bij DNA reparatie
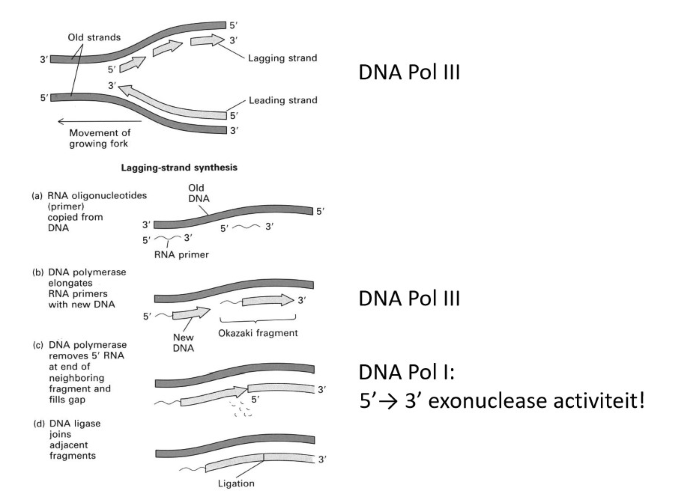
multi-eiwit moleculaire machines verzorgen DNA replicatie: polymerase III
verantwoordelijk voor de bulk van DNA synthese
2 gekoppelde DNA Pol III enzymes synthetiseren tegelijkertijd een leading & lagging streng
koppeling gebeurt door dimeer van τ-subeenheid
leading strengsynthese gebeurt continu, onmiddellijk na vooruitschuiven van helicase op lagging streng
RNA primer synthese
synthese van Okazaki fragment op lagging streng
RNA polymerase III dissocieert
dan synthese overgenomen door Pol I
tenslotte: Okazaki fragmenten worden door fosfodiësterbinding met mekaar verbonden → door DNA ligase
replisoom = dynamische multi-eiwit machine die replicatie uitvoert
initiatie van DNA replicatie in prokaryoten vs eukaryoten