technique de génétique
1/6
There's no tags or description
Looks like no tags are added yet.
Name | Mastery | Learn | Test | Matching | Spaced | Call with Kai |
|---|
No analytics yet
Send a link to your students to track their progress
7 Terms
puce à ADN
Extraction et marquage de l’ARN :
L’ARN (souvent ARNm) est extrait de cellules ou tissus à étudier.
Cet ARN est rétrotranscrit en ADNcomplémentaire et marqué avec un colorant fluorescent (souvent rouge ou vert selon les conditions).
Hybridation :
L’ADNcomplémentaire marqué est déposé sur la puce.
Il s’hybride avec les sondes complémentaires sur la puce.
Couleur 1 (ex : Cy5) pour un échantillon (ex : cellule cancéreuse)
🟢 Couleur 2 (ex : Cy3) pour un échantillon témoin (ex : cellule saine)
Lavage et lecture :
La puce est lavée pour éliminer les liaisons non spécifiques.
Un scanner détecte les signaux fluorescents à chaque spot, révélant quels gènes sont exprimés et à quel niveau.
Permet (par exemple) d’identifier si certains gènes sont plus exprimées chez individu sain ou malade
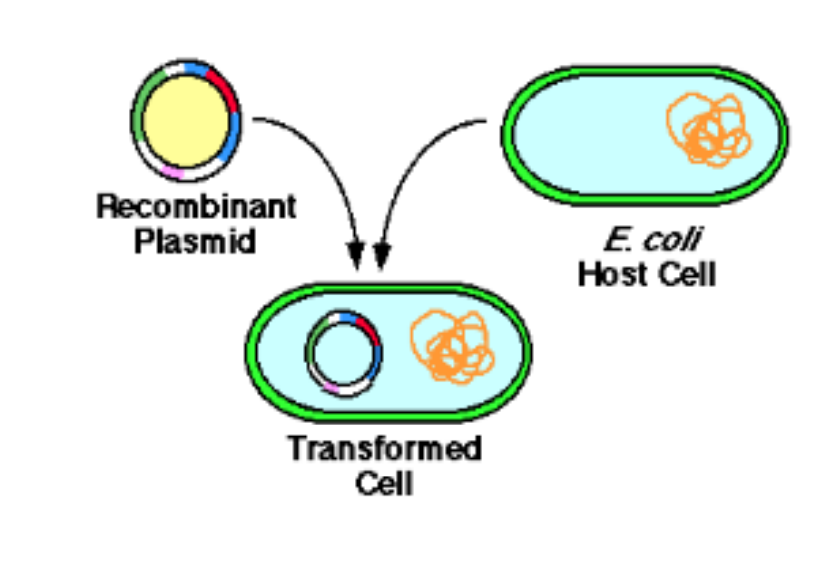
transgénèse
Tp bactérie fluorescente
dentification et isolement du gène d’intérêt.
Insertion du gène dans un vecteur (ex : plasmide, virus).
Transfert du transgène dans l’organisme cible (ex : par micro-injection, électroporation, agrobacterium…).
Intégration stable dans le génome.
Expression du gène et transmission à la descendance (si germinal).
intérêt :
Thérapie génique : corriger un gène défectueux chez un patient (encore en développement).
Agriculture : créer des plantes OGM résistantes aux insectes, maladies, sécheresse.
💊 Santé :
Produire des protéines thérapeutiques (ex : insuline humaine par bactéries transgéniques).
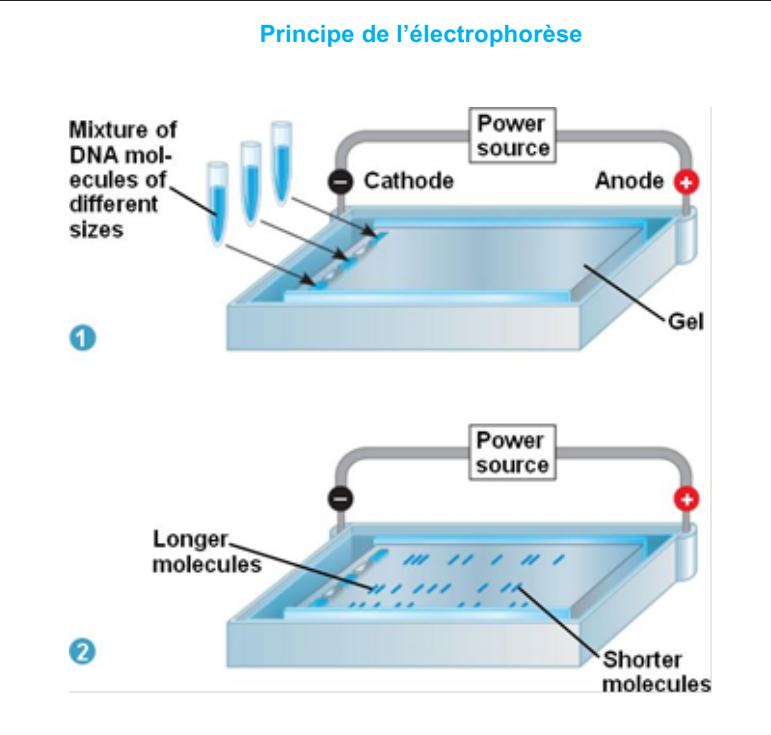
électrophorèse
Les molécules sont placées dans un gel (agarose ou polyacrylamide).
Un champ électrique est appliqué :
🔌 les molécules chargées négativement (comme l’ADN) migrent vers l’électrode positive. (chargé positif comme aa migre vers négatif)La vitesse de migration dépend de la taille :
👉 petites molécules migrent plus vite et plus loin dans le gel.
Vérifier la taille d’un fragment d’ADN.
Analyser des profils génétiques (empreintes ADN).
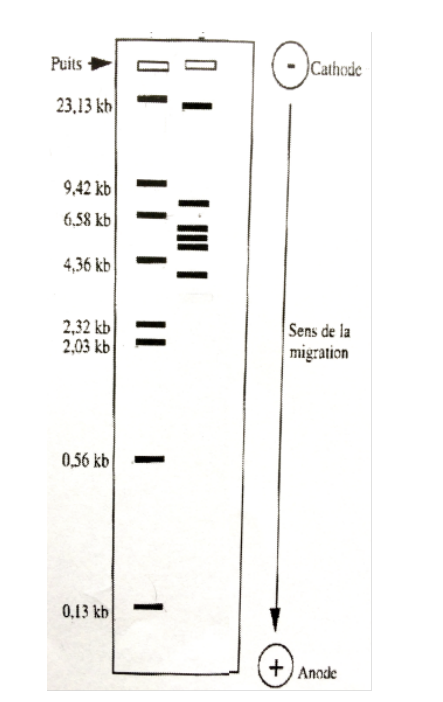
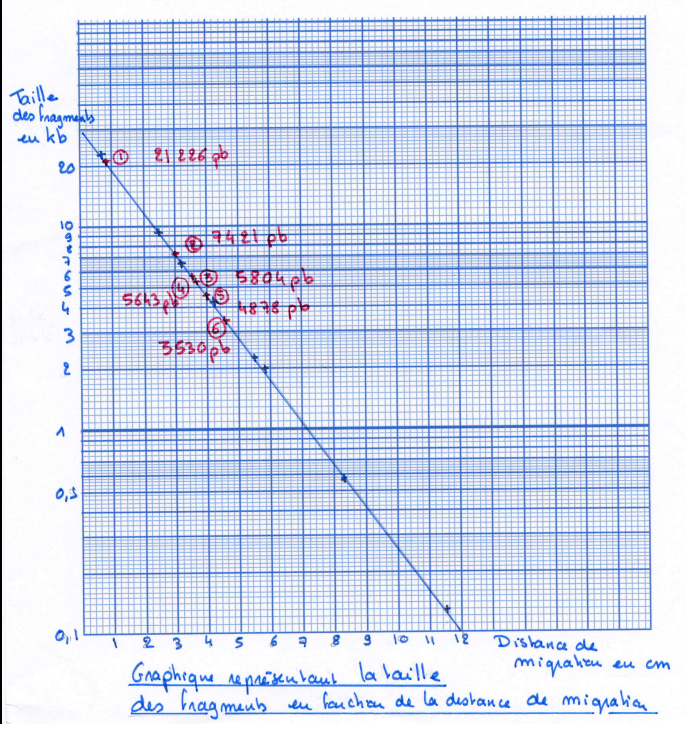
Exercice reporter résultat sur papier semi-log
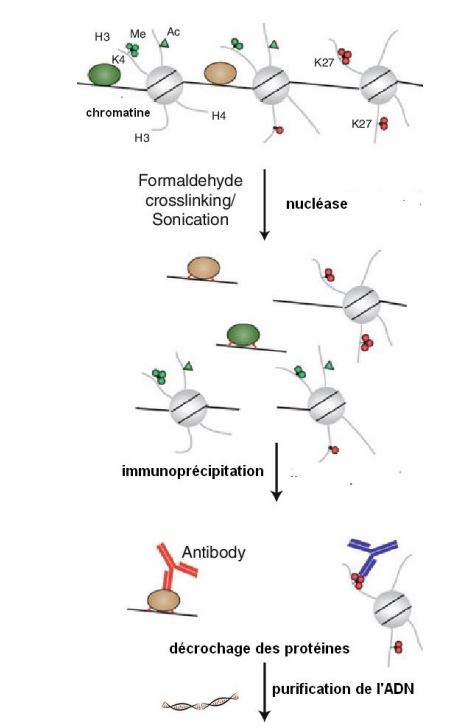
Technique de ChIP : immunoprécipitation de chromatine
Étapes principales (résumées)
Fixation (crosslinking) :
Les protéines sont liées à l’ADN dans les cellules vivantes.
Un agent chimique (ex : formaldéhyde) fixe les interactions ADN-protéine.
Fragmentation de la chromatine :
L’ADN est découpé en petits fragments (par sonication ou enzymes).
Immunoprécipitation :
On utilise un anticorps spécifique pour isoler la protéine d’intérêt et l’ADN qui lui est lié.
Purification et désolidarisation :
On inhibe la fixation (reversion du crosslink) pour libérer l’ADN.
Analyse de l’ADN :
L’ADN récupéré est analysé par PCR, qPCR, ChIP-chip ou ChIP-seq pour identifier les régions génomiques associées à la protéine.
Applications
Localiser les sites de liaison des facteurs de transcription.
Étudier les modifications épigénétiques (ex : histones acétylées).
Comprendre la régulation de l’expression génique.
Ici étude d’arabidopsis ( effet de la vernalisation )
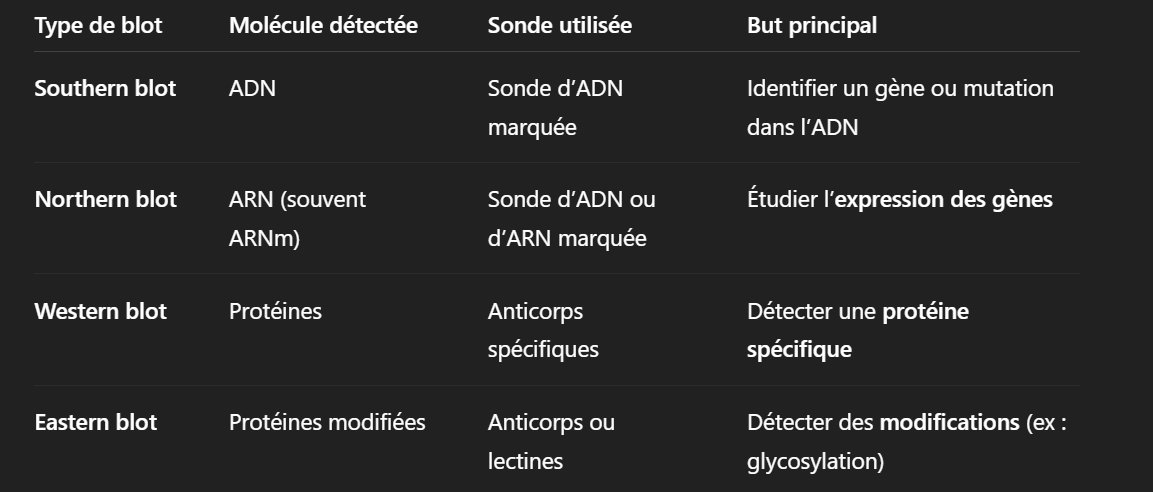
donner les 4 types de blot existant
Rq Le Eastern blot est une technique dérivée du Western blot, utilisée pour détecter des modifications post-traductionnelles des protéines, comme :
✅ l'acétylation, etc
Principe général d’un blot
Extraction de la molécule (ADN, ARN ou protéine)
Séparation par électrophorèse
Transfert sur membrane (nitrocellulose ou PVDF)
Hybridation ou incubation avec une sonde spécifique (radioactive, fluorescente ou enzymatique)
Détection du signal (par autoradiographie, chimiluminescence, fluorescence...)
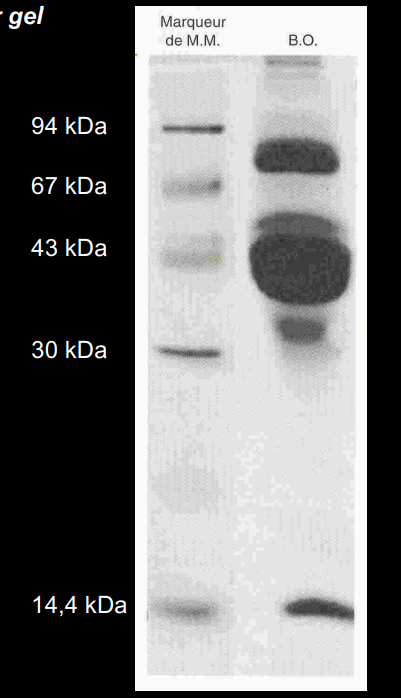
SDS - Page principe
Dégradation des structures protéiques :
On traite les protéines avec :
SDS (détergent anionique) :
👉 Déroule les protéines (dénaturation)
👉 Confère une charge négative uniforme (proportionnelle à la taille)DTT ou β-mercaptoéthanol :
👉 Rompent les ponts disulfure
Migration dans un gel de polyacrylamide :
Un champ électrique est appliqué.
Les protéines migrent vers le pôle positif (anode).
Les petites protéines migrent plus vite à travers les mailles du gel.
Coloration et visualisation :
Après migration, on colore les protéines (ex : bleu de Coomassie).
On obtient des bandes, chacune correspondant à une protéine de masse spécifique.
📏 Résultat
La distance de migration est inversement proportionnelle à la masse.
Un marqueur de poids moléculaire permet d’estimer la taille des protéines inconnues.