la biologie de l'ARN
page 38/51
questions: à la slide 34, je ne comprends pas trop si la partie du bas explique les cas où c’est avantageux de mettre des ARNm dans les p-bodies?
chez les eucaryotes les étapes par lesquelles l’ADN passe pour pouvoir avoir un bon fonctionnement cellulaire:
ADN → ARN (peut faire des protéines, des associations protéines et ARNnon codant, que ARN non codant).
une mutation dans un ARN peut influencer nombreux gènes, exemple de maladie: alzheimer, cancer, fragile X syndrome
Xnr1: exonucléase 5’→3’ . Facteur de signalisation appartenant à la famille des TGF-β — transforming growth factor beta
exosome: exonucléase 3’ → 5’
exemple de l’alzheimer
normalement
il y a une protéine transmembranaire, la β-secretase clive un bout de protéine d’un coté de la membrane et la γ-secetase clive l’autre coté de la protéine. Les secretase (en tout cas la β) sont codées par le gène BACE1.
problématique
BACE 1 possède normalement un ARNm sense et peut être un anti sens mais pas toujours exprimé? en tout cas normalement si on a un ARN double brins ça le dégrade sauf dans ce cas c’est une exception! ARNm BACE1 antisens, stabilise le ARNm sens et augmente la production des AβPP (partie de la protéine clivée). Dans l’alzheimer tout est déréglé et ça forme une accumulation de AβPP et des plaques de β-amyloide.
rôle de HOTAIR dans le cancer
HOTAIR: Hox Transcript Antisense RNA → active la formation d’hétérochromatine et rend silencieux des gènes
les cluster HOX est un regroupement de gènes important pour le développement des segments du corps, tous les petits gènes sont traduits dans le même sens et font des protéines.
Au milieu de ce cluster il y a HOTAIR, un gène qui forme un long brin d’ARN noncodant et qui se transcrit dans le sens contraire des petits gènes. L’ARN d’HOTAIR s’associe en 5’ à une histone démethylase (LSD1) et en 3’ à un polycomb repressive complex (PRC2→ méthyle les histones). Il active la formation d’hétérochromatine et rend des gènes silencieux au niveau épigénétique.
Le soucis c’est que cette répression épigénétique a lieu sur des gènes suppresseur de tumeur! HOTAIR est donc un lncRNA (long non-coding RNA) oncogénique. Le niveau d’expression de HOTAIR est indicatif pour certains types de cancer. Un haut taux de HOTAIR provoque une néoplasie c’est à dire un développement anormal et incontrôlé des cellules.
en gros: HOTAIR → gène empêche tumeur → foramtion de tumeur
épissage alternatif
on peut à partir d’un même pré-ARNm former différentes protéines grâce à l’épissage alternatif. C’est un processus très utilisé par l’être humaine, par exemple on peut faire 7 version de l’ α-tropomyosine selon si elle se trouve dans les muscles lisses, les muscles striés, les cerveau… Alors un seul gène produit une famille de protéine
ARN Dscam chez la drosophile
Dscam est un gène important chez les drosophile pour le guidage des axones vers leur cible, le gène a 24 exons et on pourrait alors faire théoriquement 38’016 protéines différentes
façon de splicer un pré-ARNm
normal
on enlève tous les introns et on laisse tous les exons
exon skipped
on enlève tous les introns et on laisse quelques exons mais pas tous
exon extended
on laisse tous les exons et on enlève les introns mais pas proprement, on laisse un bout d’intron quelque part
intron retained
on laisse tous les exons et un intron entier
alternative exons
on exprime pas tous les exons et on fait différente recombinaison
mirtron
en fait lorsque l’on splice un intron, celui ci peut former un double brins et être exporté hors de la cellule grâce à une exportine, c’est un transport actic qui nécessit de l’ATP. Ensuite le double brins est coupé par DICER et présenté au complexe RISC, celui ci va réprimer la traduction des ARNm correspondant
le miRNA est codé par une région intronique d’un pre-mRNA, il n’a pas besoin de microprocesseur (enzyme comme Drosha ou DGCR8) pour sa maturation
snoRNA
c’est une classe d’ARN non codant, important dans la maturation des ARN ribosomiques car il guident les modification chimique telles que la méthylation et la pseudouridylation (guide l’enzyme qui pseudouridyle)
pseudourydilation: U→ Ψ et il est lu comme C
les snoRNA sont produit comme les mirtron avec des séquences introniques et ils s’associent ensuite à des protéines
maladies humaines liées à l’épissage
dystrophie myotonique de Steinert
c’est une maladie qui touche 1/20’000 personne, elle provoque de la faiblesse musculaire et des problèmes cardiaques, les gens meurent généralment entre 50-60 ans. Elle est autosomal dominante mais d’une pénétrance partielle, c’est à dire que toutes les personnes qui héritent du gène muté ne développeront pas nécessairement la maladie, ou ne la développeront pas avec la même gravité.
c’est le gène DMPK qui est touché— il code pour une protéine kinase— et en fait ça c’est un cas rare ou c’est l’ARN DMPK qui est toxique et nos la protéine DMPK
cause:
il y a une mutation dans la région 3’UTR, chez les individus sains, le nombre de répétitions CTG dans l’ADN est limité. Chez les personnes atteintes de DM1, ce nombre est considérablement augmenté. Les répétitions de triplet CUG dans l’ARNm provoque une structure anormale de l’ARNm et perturbe la régulation de l’épissage. Les ARNm DMPK s’accumulent dans le noyau et forment des aggrégats. Ces structures anormales séquestrent des protéines essentielles, telles que MBNL1 (muscleblind-like 1), qui jouent un rôle crucial dans la régulation de l'épissage de l'ARN. La séquestration de MBNL1 entraîne des anomalies d'épissage de nombreux ARNm, ce qui contribue à la diversité des symptômes observés dans la DM1. On appelle ça une spliceopathie.
dystrophie musculaire de Duchenne
Il y a une perte de tissus musculaire dans certaines région, notemment le pelvis et les cuisses, les tissus musculaires sont remplacés par des tissus graisseux. L’éspérance de vie est de 30 ans dû à une insuffisance respiratoire
cause:
Elle est liéé au chromosome X (et recessive) mais elle touche plus fortement les hommes ~1/16’500 homme. Elle touche le gène DMD qui est le plus long gène humain (il y a 86 exons et 99% d’i ntrons avec 7 promoteurs différents).
La plus part des mutations sont des “loss of function”, dans la plus grande part des cas elle arrive sur des exons
mutation ponctuelle (faux sens ou non sens)
délétion ou insertion — avec ou sans changement de cadre de lecture
Mais il y a quelques mutations “introns flanquant” (mutation dans l’intron mais proche de l’exon et provoque modification des sites d’épissage 5’ ou 3’), des mutations au niveau des promoteurs qui provoque une trop faible transcription ou des mutations “deep intronique” qui sont profondement au milieu de l’intron et elles sont difficilement répérables car elles n'affectent pas directement les sites d'épissage connus.
→ exemple: dans le gène, dans un intron il y a un G transformé en T, normalement on s’en fiche le souci c’est que dans l’ARNm le T devient A et ce A est reconnu comme site d’épissage, alors on épisse pas assez de l’intron et un partie reste. C’est un peu comme le “exon extended” mais pas fait exprès. De plus dans le bout d’intron qu’on a laisser (=pseudoexon) il y a un codon stop très tôt, la protéine est alors tronquée.
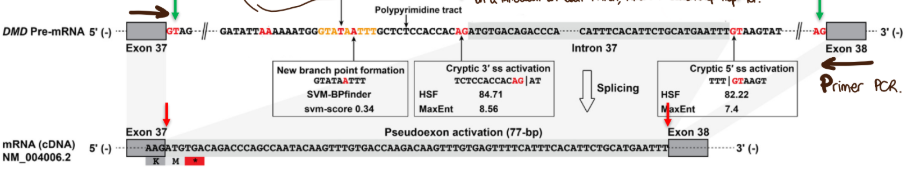
conséquences:
Les mutations du gène DMD entraînent une production insuffisante ou absente de dystrophine → protéine qui connecte le squelette d’actine à la membrane basale de la fibre musculaire.
On peut faire un diagnostique prénatal avec des scan ultrason de la structure musculaire ou un test ADN.
détermination du sexe chez la drosophile
rappel: afin de déterminer le sexe, il faut regarder le nombre de jeu de chromosome et le ratio de X présent [#X/#de jeu de chromosome]. Si le ratio est plus grand ou égal à 1, femelle, mâle sinon
l’ARNm doublesex femelle est formé par cascade de réaction de splicing alternatif tandis que l’ARNm doublesex mâle est fait par défaut.
sex lethal preARNm
il y a trois exon, le mâle les prend les trois, la femelle prend le premier et le dernier. En fait il y a un codon stop dans l’exon du milieu, si on le prend la protéine ne fonctionne pas (d’où le fait que le mâle n’ait pas de protéine fonctionnelle). La protéine Sxl (sex lethal) est fonctionnelle chez la femelle et qu’est ce qu’elle fait? elle autocataslyse sa production en inhibant la “prise” de l’exon avec le codon stop et elle inhibe aussi l’exon du mileu dans l’ARN transformer
transformer preARNm
c’est le même principe, trois exons avec celui qui milieu qui a un codon stop, le mâle prend tout donc protéine pas fonctionnelle et la femelle prend juste l’exon 1 et 3 car la protéine Sxl produite à la première étape encourage cette réaction. La femelle a donc une protéine Tra celle ci va encourager un certain épissage du pré ARNm double sex dont nous allons parler maintenant
double sex preARNm
il y a trois exon, mais cette fois celui du milieu a un signal d’épissage faible c’est à dire qu’il n’est par défaut pas pris dans l’ARNm mature. Le mâle prend donc le premier et le dernier exon formant une ceraine protéine Dsx.
La femelle possède une protéine Tra fonctionnelle, elle va former un complexe avec une autre protéine Tra2 et rendre l’exon du milieu mieux visible et encourager à le prendre dans l’épissage. L’exon du milieu est un peu spécial, il a un site de polyadénisation qui permet de lui rajouter sa queue poly A et termine l’épissage, la femelle ne peut donc pas prendre le dernier exon.
Au final, les mâles et les femelles ont les deux des protéines double sex différentes mais foncitonnelle.
la cascade pour réaliser ces deux protéines a des avantages: amplification des protéines et offre une régulation plus fine
Régulation de l’activité d’un mRNA
il y a trois niveaux auxquels l’ARNm peut être régulé:
niveau de traduction,
sa stabilité
sa localisation
steady state level of an mRNA: quantité stationnaire d’un mARN donné à un moment donné. Il dépend du taux de transcription du ARN et du taux de dégradation du mARN (indiqué par la demi vie). Le steady state level d’un ARNm influence son activité.
voies de dégradation d’un ARNm
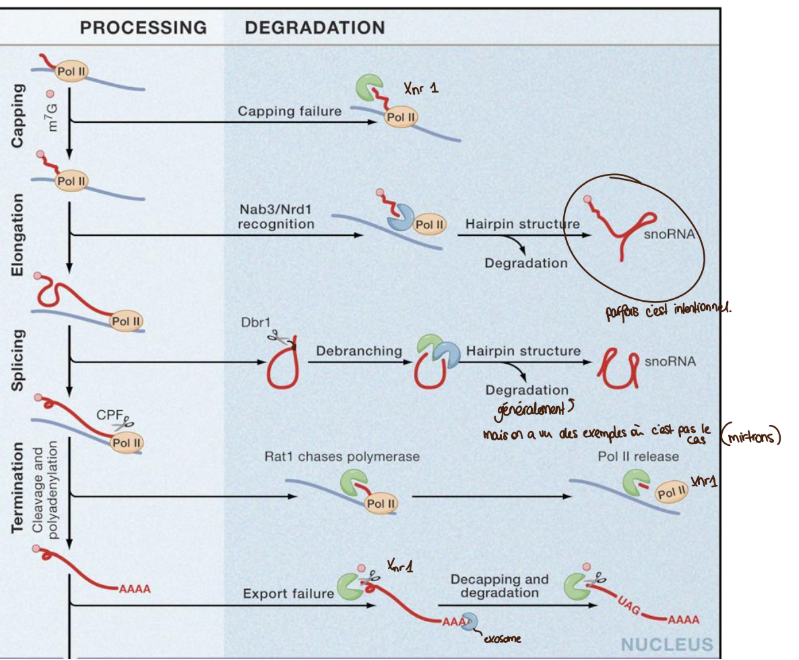
dans le noyau
dans le cytosol
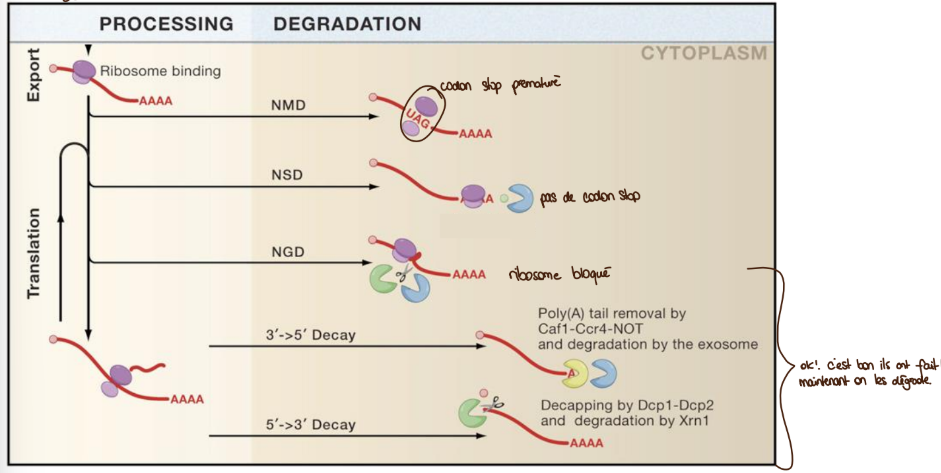
problèmes qui peuvent survenir dans le ARNm pour qu’il soit dégradé dans le cytosol: codon stop prématuré, pas de codon stop, ribosome bloqué, juste ils ont vécu assez longtemps maintenant ils doivent être dégradés.
déadénylation
on enlève un bout de la queue poly A, celle ci stabilisait initalement l’ARNm et donc en son abscence il va être dégradé. Il y a un décaping puis une exonucléase 5’→ 3’ va dégrader l’ARNm depuis la coiffe donc
parfois on envlève toute la queue poly A et une exonucléase 3’→ 5’ la dégrade depuis la fin
decaping
ça arrive généralement si un codon stop est apparu trop tôt, on enlève la coiffe et une exonucléase 5’→3’dégrade l’ARNm
endonucléase
une endonucléase fait des trous partout dans l’ARNm et il aime pas, il va être dégradé
dégradation sélective d’un ARNm — élément riche en AU
chez le lapin, l’ARNm de la β-globine est stable cas il possède une région 3' riche en G et C tandis que le ARm pour le facteur de croissance GM-CSF a une demi vie plus courte et lui il possède une région 3’ riche en A et U. C’est ces régions riche en A/U et G/C qu’on peut satabiliser l’ARNm et environs 10% des ARNm de mammifères contiennet ces domaines d’insatbilité, on les appelle ARE.
contrôle de la dégradation de l’ARNm tubuline par son polypetide
il y a un ARNm pour la tubuline, sa traduction commence et le polypéptide en formation sort du ribosome. Les tubines déjà formées reconnaissent cette protéines en croissance et elles recrutent une fonction RNase du ribosome. C’est à dire que le ribosome va découper l’ARNm et il ne sera plus utilisable.
colchicine: déassemblage des microtubules. Cause une baisse de la synthèse de tubuline.
vinblastine: déassemblage des microtubules et précipitation des tubulines. Cause une augmentation de la synthèse de la protéine tubuline.
Staufen-mediated decay (SMD)
Parfois les ARNm se mettent sous forme de loop pour rendre la traduction en boucle plus efficace mais dans certains cas les ARNm doivent être traduit qu’une seule fois puis être dégradé.
Ces ARNm sont spéciaux, ils ont un codon start et stop comme normal mais en région 3’UTR, ils ont un SBS — Staufen-binding site, ça fait une bobby pin. La protéine STAU se lie au site SBS. Une autre protéine UPF1 reconnait quand STAU se lie et si UPF1 est phosphorylé alors elle a une activité d’hélivace. Elle va inhiber la traduction du ARNm et favoriser sa dégradation.
Nonsense-mediated decay (NMD)
c’est un contrôle de qualité, on dégrade l’ARNm si le codon stop apparait trop tôt
normalement
Les complexes de jonction exon-exon (EJC) sont déposés lors de l'épissage et sont retirés par les ribosomes lors de la traduction. Le codon stop se trouve à la fin de la région codante, quand le ribosome atteint la fin de l’ARNm, tous les EJC sont retirés.
quand le codon stop apparait trop tôt
le codon stop apparait trop tôt, il apparait avant le dernier EJC, les protéines Upf (Up-frameshift) se lient au ribosome arrêté et interagissent avec les EJC en aval du codon stop prématuré. Cette interaction déclenche une cascade d'événements qui conduit à la dégradation de l'ARNm: déadénylation, décapping et dégradation de l’ARNm par des exonucléases
Nonstop mediated decay
c’est aussi un contrôle de qualtié, on dégrade l’ARNm s’il lui manque son codon stop.
mécanisme: le ribosome traduit tout l’ARNm y compris la queue poly A comme il n’a pas pu s’arrêter. ça produit beaucoup de lysine, en effet le codon AAA correspond à la lysine. Un complexe reconnait cette suite de lysine et intervient comme stop au ribosome, il conduit aussi au dessassemblage, à la dégradation de l’ARNm et de la protéine
No-go-mediated decay
c’est quand le ribosome est bloqué sur l’ARNm, par exemple s’il manque un ARNt. Le même complexe qui reconnait les suites de lysine s’introduit comme facteur d’arrêt de transcription. Le processus ressemble beaucoup au nonstop mediated decay à la différence que cette protéine n’est pas dégradée, seulement l’ARNm
méthylation de l’ARNm
C’est un processus réversible car il y a un “writer” m6A methyltransferase et un “eraser” m6A demethylase. La méthylation se fait toujours sur un A, celui ci est dans un consensus. Il est ensuite lu par un “reader”, une protéine qui se lit à m6A car elle reconnait la méthylation. La méthylation est souvent proche du codon stop et on en trouve pas seulement dans les ARNm mais aussi dans les miRNA, rRNA, tRNA, long noncoding RNAs (”Xist”). Mais très peut du total des adénosine est méthylé.
La m6A joue un rôle crucial dans la régulation de l'expression des gènes, et des dysfonctionnements de ce processus ont été associés à diverses maladies, typiquement dans l’artériosclérose ou l’hypertrophie cardiaque.
Si la méthylation a lieu dans la séquence codante de l’ARNm, les sites méthylés ralentissent le ribosome dans sa lecture et ça conduit à la dégradation de l’ARNm — cf le point précédent: no-go-mediated decay. Afin de les dégarder on les envoie dans les P-bodies.
Les P-bodies sont les principaux sites de stockage et de dégradation des ARNm dans le cytoplasme. Ils agissent comme des "zones de triage" pour les ARNm qui ne sont pas activement traduits par les ribosomes: soit les ARNm sont dégradés soit ils sont stockés temportairement puis relâchés pour la traduction. Plus un ARNm est méthylé, plus il est associé aux P-bodies. On peut étudier ça à l’aide d’une immunopreciptiation avec un anti-m6A
L’avantage des P-bodies et du stockage des ARNm
régulation fine et rapide de l’expression génique.
Par exemple permet à la cellule de répondre rapidement aux changements environnementaux ou aux besoins cellulaires en cas de stress. Les ARNm sont stockés en cas de mauvaises conditions et quand l’environnement redevient favorable ils sont relachés.
prévention de la traduction aberrante
quand des ARNm contiennent des erreurs (cf les points précedents) ils sont envoyés au P-bodies pour être dégradés et ils ne sont pas traduits entre deux.
→ ce sont plutôt les gènes ponctuels qui sont méthylé et stockés dans les P-bodies tandis que les house-keeping gens ne sont pas ou peu méthylé.
localisation de l’ARNm
afin d’étudier la localisation des ARNm on utilise l’hybridation in situ sur des cellules qui ont été fixées (c’est à dire qu’elles ont été stoppées dans leur métabolisme)
localisation d’un ARNm dans une cellule
on fait une hybridation in situ → dans son milieu naturel
marquage indirect
une sonde qui contient un antigène (digoxigenin) dont on a les anticorps
marquage biotine/streptavidin
la sonde est formée par de l’ADN ou ARN qui contient une cytosine+biotine en son extrémité 3’. La biotine est détectable par la streptavidin. Celle si est souvent liée à une enzyme qui produit une réaction colorée
marquage direct
des nucléotides liés à des molécules fluorescentes sont incorporé dans les sondes et on peut donc “exciter” les sondes
exportation des ARNm du noyau
les ARNm doivent passer par des pores nucléaires, c’est un transport actif et séléctif. Ils ont besoin de protéines d’exportation notement l’exportine-t et la caryophérine.
Les pre-miRNAs sont exportés par association au transporteur XPO5 (exportin 5) qui leur facilite le passage par le pore nucléaire.
Les pre-miARN peuvent provenir d’un exon transcrit ou d’un intron epissé, c’est alors un “mirtron”: le gène pour le miRNA se situe dans un intron. Il n’est pas maturé par Drosha, mais produit par épissage et clivage du site de branchement.
Exportation nucléaire des ARNm chez les métazoaires
La protéine UAP56 se lie à l'ARNm en fonction de critères spécifiques liés à sa maturation, tels que la structure de l'ARNm, l'épissage, la transcription et la formation de l'extrémité 3'.
Cette liaison est une étape cruciale pour l'exportation de l'ARNm.
Le complexe recrute ensuite la proétine ALY qui joue un rôle de liaison entre UAP56 et TAP
ALY recrute à son tour TAP-NXT qui est essentiel pour l’exportation de l’ARNm, il intéragit avec les nucléoporines par son domaine central
Contrôle de l'exportation des ARNm par des protéines à domaine SR
Certaines protéines SR (par exemple : 9G8, SF2/ASF) font la navette entre le noyau et le cytoplasme. Elles peuvent controler l’exportation de l’ARNm. Ces navettes dépendent de la phosphorylation de certains acides aminés sérine. Les protéines SR peuvent contrôler l'exportation de différents types d'ARNm, qu'ils soient épissés ou non.
Il est suggerer
qu'il existe une relation entre l'épissage et l'exportation de l'ARNm, mais le mécanisme exact n'est pas clair
la protéine Aly fonctionne pour l’épissage — en interagissant avec le pré-ARNm pendant l’assemblage au spliceosome— et pour l’exportation du ARNm.
Aly se lie fortement à l’ARNm épissé et favorise son exportation
importation de différents types d'ARN dans le noyau
les sous unités U1, U2, U4, U5 snRNAs sont des petits ARN nucléaires impliqués dans l'épissage de l'ARN. Ils sont transcrit dans le noyau, exportés vers le cytoplasme pour subir une maturation puis réimportés dans le noyau pour participer à l’épissage de l’ARN.
Le 5S rRNA nucléaire est importé dans les mitochondries, il est impliqué dans la traduction des protéines mitochondriales et de nombreux ARNt sont importés dans la mitochondrie