7 Ácidos nucleicos (TANS)
7.1 Estructura del ADN y replicación
7.1.1 Nucleosomas: conjuntos de ADN y proteínas
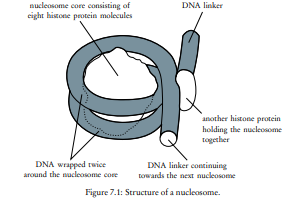
Los nucleosomas son las unidades en las que se organiza la cromatina en el núcleo de células eucariotas. Un nucleosoma consiste en una estructura globular con un núcleo de 8 proteínas histonas, alrededor del cual se enrolla el ADN. La histona H1 conecta el ADN con el núcleo proteico, y cada "conjunto" de ADN está enlazado con el siguiente mediante histonas conectoras. Las colas N-terminales de las histonas se extienden hacia afuera y pueden unirse a nucleosomas vecinos, acercándolos y permitiendo el superenrollamiento durante la condensación de los cromosomas en la mitosis. La modificación de las histonas (como la metilación o acetilación) afecta el nivel de condensación y regula procesos como la transcripción al hacer que el ADN sea accesible o impedir el acceso de enzimas.7.1.2 Replicación del ADN
El ADN consiste en dos hebras antiparalelas, cada una formada por una columna vertebral de azúcar-fosfato y una de las cuatro bases nitrogenadas unidas mediante enlaces de hidrógeno complementarios:
Adenina (purina) se une a Timina (pirimidina) con dos enlaces de hidrógeno.
Guanina (purina) se une a Citosina (pirimidina) con tres enlaces de hidrógeno.
Este apareamiento específico reduce el riesgo de mutaciones, ya que cada base tiene alta afinidad por su pareja complementaria.
Proceso de replicación en procariotas:
La replicación ocurre en dos hebras parentales:
Hebra líder (5’-3’): replicación continua.
Hebra rezagada (3’-5’): replicación discontinua mediante fragmentos de Okazaki.
Pasos en la replicación:
Helicasa desenrolla y separa las hebras parentales, y girasa estabiliza el ADN en la horquilla de replicación.
Proteínas de unión a cadena sencilla estabilizan las hebras y evitan su re-enrollamiento.
En la hebra rezagada:
Primasa añade un cebador de ARN en el sitio cercano a la horquilla de replicación.
A medida que el ADN se desenrolla más, se añaden más cebadores.
ADN polimerasa III añade nucleótidos en dirección 5'-3'.
ADN polimerasa I reemplaza los cebadores de ARN por nucleótidos de ADN.
Ligasa une los fragmentos de Okazaki.
En la hebra líder:
Primasa coloca un cebador de ARN en el extremo 3'.
ADN polimerasa III añade nucleótidos en dirección 5'-3'.
ADN polimerasa I reemplaza el cebador de ARN.
7.1.2 Replicación del ADN
El ADN consiste en dos hebras antiparalelas, cada una formada por una columna vertebral de azúcar-fosfato y una de las cuatro bases nitrogenadas unidas mediante enlaces de hidrógeno complementarios:
Adenina (purina) se une a Timina (pirimidina) con dos enlaces de hidrógeno.
Guanina (purina) se une a Citosina (pirimidina) con tres enlaces de hidrógeno.
Este apareamiento específico reduce el riesgo de mutaciones, ya que cada base tiene alta afinidad por su pareja complementaria.
Proceso de replicación en procariotas:
La replicación ocurre en dos hebras parentales:
Hebra líder (5’-3’): replicación continua.
Hebra rezagada (3’-5’): replicación discontinua mediante fragmentos de Okazaki.
Pasos en la replicación:
Helicasa desenrolla y separa las hebras parentales, y girasa estabiliza el ADN en la horquilla de replicación.
Proteínas de unión a cadena sencilla estabilizan las hebras y evitan su re-enrollamiento.
En la hebra rezagada:
Primasa añade un cebador de ARN en el sitio cercano a la horquilla de replicación.
A medida que el ADN se desenrolla más, se añaden más cebadores.
ADN polimerasa III añade nucleótidos en dirección 5'-3'.
ADN polimerasa I reemplaza los cebadores de ARN por nucleótidos de ADN.
Ligasa une los fragmentos de Okazaki.
En la hebra líder:
Primasa coloca un cebador de ARN en el extremo 3'.
ADN polimerasa III añade nucleótidos en dirección 5'-3'.
ADN polimerasa I reemplaza el cebador de ARN.
7.1.3 ADN no codificante
No todo el ADN del núcleo es transcrito y traducido en proteínas. Las regiones codificantes se llaman "exones" y las no codificantes "intrones". Algunas regiones de ADN no codificante tienen funciones importantes:
Regulación de la expresión génica: sitios donde las proteínas pueden unirse para promover o reprimir la transcripción de genes.
Telómeros: secuencias en los extremos de los cromosomas que se acortan en cada replicación, protegiendo los genes importantes.
Genes de ARNt y ARNr: estos no codifican proteínas, pero son esenciales para la traducción.
7.2 Transcripción y expresión génica
7.2.1 Transcripción
La transcripción es la síntesis dirigida por el ADN de ARNm mediante la ARN polimerasa. Al igual que en la replicación del ADN, el proceso de transcripción ocurre en la dirección 5′–3′. La ARN polimerasa solo puede añadir nucleótidos de ARN libres al extremo 3′ de la molécula de ARNm en crecimiento. A continuación se describe el proceso de transcripción:
Iniciación: La ARN polimerasa se une a la región promotora del gen (esta región contiene una secuencia específica de bases y señala a la enzima que inicie la transcripción).
Elongación: La ARN polimerasa desenrolla y transcribe simultáneamente la región codificante en dirección 5′–3′.
Terminación: La ARN polimerasa se separa de la cadena de ARNm después de llegar a la secuencia de terminación, y el ARNm se desprende de la plantilla de ADN para ser modificado y luego traducido fuera del núcleo.
La cadena de ADN que se transcribe se llama la "cadena antisentido" (el ARNm contendrá una secuencia idéntica a la "cadena sentido", que codifica los genes de interés, con uracilo en lugar de timina).
7.2.2 Modificaciones postranscripcionales del ARNm
En organismos eucariotas, el ARNm se modifica antes de salir del núcleo para ser traducido. La modificación principal consiste en la eliminación de intrones mediante el corte y empalme del ARN, que "elimina" las secuencias no codificantes y une los exones restantes. Muchos genes tienen varios exones que pueden empalmarse de diferentes maneras para crear una amplia gama de proteínas distintas.
7.2.3 Expresión génica: regulación de la transcripción
El proceso de transcripción puede regularse tanto por proteínas como por factores ambientales. Las proteínas que se unen a la región promotora, conocidas como factores de transcripción (FT), son un ejemplo de esto. Los FT alteran la capacidad de la ARN polimerasa para unirse al promotor, afectando así la transcripción de genes específicos. Existen FT normales, activadores que aumentan la transcripción, y represores que disminuyen la tasa de transcripción. Los factores ambientales también pueden afectar la expresión génica de manera no hereditaria. El estudio de la epigenética se centra en estos factores y en cómo alteran la transcripción, las modificaciones del ARNm, la traducción, etc. Por ejemplo, la dieta, la edad y las enfermedades pueden influir en los cambios en la expresión génica sin que necesariamente impliquen alteraciones directas en la secuencia del ADN. Algunas modificaciones epigenéticas incluyen:
Metilación del ADN: puede fijar genes en una posición "inactiva", impidiendo la transcripción.
Modificación de histonas: la acetilación y metilación pueden afectar el nivel de condensación de la cromatina.
MicroARNs: pueden unirse al ARNm, provocando la degradación del complejo completo, lo que impide que el gen sea traducido.
Como se mencionó anteriormente, el enrollamiento y desenrollamiento del ADN por las interacciones con los nucleosomas pueden regular el proceso de traducción. Las regiones de ADN altamente condensado generalmente no se transcriben, ya que las enzimas no pueden acceder fácilmente al ADN (las regiones que permanecen altamente condensadas todo el tiempo se conocen como heterocromatina y no se transcriben normalmente). Por otro lado, la cromatina descondensada es fácilmente accesible para las enzimas de transcripción y los factores de transcripción, y se transcribe con facilidad.
7.3 Traducción
7.3.1 Traducción
La traducción es el proceso mediante el cual los transcritos de ARNm se convierten en cadenas polipeptídicas que luego pueden modificarse para crear proteínas funcionales.
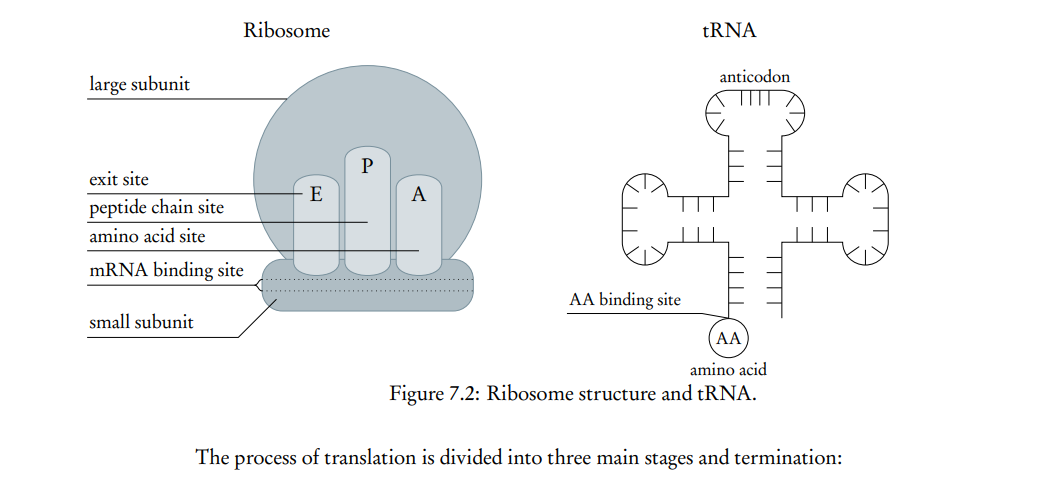
El proceso de traducción se divide en tres etapas principales y la terminación:
Iniciación: La molécula de ARNt con el anticodón de inicio UAC se une a la subunidad pequeña del ribosoma (sitio de la cadena peptídica en el diagrama). La subunidad pequeña luego se une al extremo 5′ de la cadena de ARNm y se desliza a lo largo de ella en dirección 5′ a 3′ hasta encontrar el codón de inicio en el ARNm (AUG). Las subunidades grande y pequeña del ribosoma se unen. Una molécula de ARNt con el anticodón complementario al siguiente codón del ARNm en la cadena se une al sitio del aminoácido del ribosoma. Comienza el proceso de elongación.
Elongación: El ARNt entrante se aparea con su codón complementario del ARNm en el sitio del aminoácido del ribosoma. Los aminoácidos de los ARNt en los sitios peptídico y de aminoácidos forman un enlace peptídico, y la cadena polipeptídica en crecimiento permanece unida al ARNt en el sitio de aminoácidos. La formación del enlace peptídico es catalizada por el ribosoma.
Translocación: La subunidad grande del ribosoma avanza y la subunidad pequeña se desliza detrás de ella, moviendo todo el ribosoma tres nucleótidos a lo largo de la cadena de ARNm. El ARNt en el sitio peptídico se desplaza al sitio de salida (donde el ARNt se libera y puede unirse a un nuevo aminoácido). El ARNt que lleva la cadena polipeptídica en crecimiento se desplaza al sitio peptídico. El siguiente codón a traducir se encuentra en el sitio de aminoácidos, donde su codón complementario del ARNt puede unirse. El proceso se repite.
Terminación: Cuando uno de los tres codones de paro (UAG, UAA, UGA) alcanza el sitio de aminoácidos del ribosoma, la subunidad grande avanza sobre la subunidad pequeña, separándose de ella y provocando que la cadena polipeptídica se libere del último ARNt. La polipeptídica puede entonces plegarse y ser modificada para crear una proteína funcional.
Es importante señalar que la traducción de una única cadena de ARNm puede ocurrir en varios ribosomas simultáneamente (muchos se pueden unir), formando un polisoma. Los polisomas se pueden visualizar utilizando software de visualización molecular.
7.3.2 Ribosomas libres vs. ribosomas unidos
En organismos procariotas, los ribosomas libres pueden comenzar el proceso de traducción inmediatamente después de la transcripción, ya que no hay membrana nuclear. En eucariotas, sin embargo, el ARNm debe salir primero del núcleo a través de los poros nucleares y luego puede ser traducido en ribosomas libres en el citoplasma o en ribosomas unidos al retículo endoplásmico rugoso.