régulation
slide 19/34
régulation de l’expression des gènes
il existe plusieurs type des gènes: ceux exprimer dans chaque cellules, utile au fonctionnement de base, on les appelle les house keeping genes, ensuite il y a d’autre gène spécifiques aux cellules mais aussi au temps/stade de développement. Ces derniers ont différent niveau de régulation (plutôt chez les eucaryotes): ADN, ARN et protéines
au niveau de l’ADN de l’expression des gènes, on distingue le contrôle négatif avec un répresseur et celui positif avec un activateur
exemple de régulation génétique ADN
négatif
par répression
ajout d’arginine dans le milieu et la bactérie réprime les enzymes de synthèse de l’arginine.
Mécanisme: l’arginine se lie au répresseur et l’active, celui ci peut maintenant se lier à l’opérateur sur le gène qui code ces enzymes de synthèse et empêche leur production. Sans l’arginine, le represseur est présent mais inactif car il n’a pas la bonne configuration
par induction
abscence de lactose dans le milieu va réprimer la production de galactosidase
Mécanisme: comme dans le cas de l’arginine, le répresseur est toujours présent, la différence ici c’est que le répresseur est actif lorsqu’il n’y a pas de lactose et lors d’ajout de lactose la molécule va transformer la conformation du répresseur et l’empêcher de se lier à l’opérateur du gène. Le gène sera donc transcrit en présence de lactose et formera des galactosidases qui pourront dégrader le lactose en galactose et glucose
positif
par induction
ajout de maltose dans le milieu, la bactérie induit l’opéron codant pour la dégradation du maltose, ici il n’y a pas de répresseur
Mécanisme: la présence de maltose encourage la transcription du gène car la molécule se lie à l’opéron et récrute une polymérase, le site de liaison de l’activateur n’est pas toujours à proximité du gène qu’il influence
régulation de l’ARN
peut se faire au niveau de la traduction et de la transcription et elle est très rapide car il n’y a pas besoin de fabriquer des protéines de régulation (répresseurs) en effet on utilise des ARN régulateur appartenant au ARNnc — ARN non codant
il y a trois type de régulation:
sRNA (small RNA)
un petit brin d’ARN (→ ARNs) se fixe sur l’ARNm codant car ils sont homologue et très souvent ça inhibe sa traduction mais il y a plusieurs mécanismes:
inhibition de la traduction
stimulation de la traduction
dégradation de l’ARNm
protection de l’ARNm
riboswitches
très important chez les bactéries mais aussi présent dans d’autres organismes typiquement les plantes ou les champignons. Ce sont des ARN riboswitches en amont (avant) la séquence codante de l’ARNm et elle va former des structures 3D car elles possèdes des régions homologues. Lorsque des petites molécules, souvent des produits finaux de voies de biosynthèse sont intégré dans la molécules d’ARNm les structures 3D se déplacent et répriment l’expression du gène. C’est un peu comme la régulation avec des protéines mais c’est plus simple et donc probablement plus ancien.
atténuation
c’est un phénomène qui se passe uniquement chez les procaryotes, ça ne fonctionne pas chez les eucaryotes à cause de la présence d’intron et la traduction n’a pas lieu au même endroit que la transcription
il s’agit d’interrompre la traduction après son initiation car la première partie de l’ARNm va former des structures 3D et une de ces structures vas stopper le ribosome
il y a plusieurs régions dans le ARNm:
régions codantes
promoteur → site de liaison de la polymérase
opérateur → site de régulation
leader peptide
exemple du tryptophane
régions sur le gène de synthèse du tryptophane: 1-2(régions codantes) - 3-4 (régions non codantes)
nous pouvons hybrider les régions 1:2, 2:3, 3:4
si les régions 3:4 s’hybrident ça bloque la transcription et la région riche en uracile favorise la séparation de l’ARNm de l’ADN, provoque fin de la transcription
si les régions 2:3 s’hybrident cela empêche l’hybridation 3:4 et permet la fin de la transcription.
Le type d’hybridation est déterminé par l’abondance du tryptophane (et donc la nécessité ou non d’en synthétiser). Je crois que s’il y a beaucoup de tryptophane, les ARNt tryptophane sont abondant, la traduction a lieu rapidement et ça ne laisse pas le temps à la région 2:3 de se lier mais à la région 3:4 oui → stop la traduction et la translation
régulateur globaux et détection de l’environnement
parfois un changement extérieur nécessite la régulation de plusieurs gènes or il faut que ce soit coordonné, c’est à ce moment qu’intervient le système de contrôle global, il se superpose au contrôle propre de chaque opéron
exemple : répression catabolique
lorsqu’on donne du sucre à une bactérie, elle utilise toujours le glucose en premier et réprime toutes les voies métaboliques des autres sucres. Seulement une fois la réserve de glucose utilisée, les autres sources de carbone sont utilisées. Ce contrôle global réprime aussi la formation du flagelle et la synthèse d’antibiotique.
pourquoi lier ces caractères: quand il y a assez de ressources, il est inutile de se déplacer et de produire des antibiotiques qui sont énergivore. Il y a assez de place pour tout le monde à ce moment là.
la répression catabolitique est causée par une protéine (un activateur appélé: récepteur de l’AMP cyclique—CRP). Elle se lie au promoteur des gènes soumis à la répression catabolitique mais cette liaison n’a lieu qu’en présence d’AMPc. C’est un nucléotide régulateur important dans les cellules procaryotes et eucaryotes. Le glucose stimule l’export d’AMPc et donc la cellule peut se fier au niveau d’AMPc interne pour connaître le taux de glucose.
intégration de plusieurs niveaux de régulation
exemple du lactose:
afin de produire les enzymes de dégradation du lactose, il faut deux conditions:
la présence de lactose, la répresseur lacl est désactivé par le lactose
l’absence de glucose, forte [AMPc], la CRP se lie au promoteur et active la transcription
→ il y a une intégration des deux signaux par combinaison de régulation contrôle positif et contrôle négatif
Comment les bactéries perçoivent-elles leur environnement?
il y a un sytème à deux composantes: senseur et régulateur et une phosphatase qui permet de revenir à l’état initial
le senseur —histidine kinase— est situé à l’extérieur et percoit l’environnement → il est alors autophosphorylé et donne le phosphate au régulateur, le régulateur lui est dans le cytoplasme, il agit comme inducteur ou répresseur de l’expression des gènes
quelques exemples de paramètres détéctés: nutriments, lumière, accepteur/donneur d’électrons, température, voisin bactériens
exemple: adaptation aux changements osmotiques
il y a différent types des porins, typiquement la OmpF et OmpC qui différent par la taille de leur pores. Lorsque la pression osmotique est basse la membrane externe contient plus de OmpF et si elle est haute, elle contient plus de OmpC.
Lors de changement osmotiques, c’est le senseur histidine kinase (EnvZ) qui s’autophosphoryle et donne le phosphate au régulateur OmpR. Le régulateur a une action différente si la pression est
élévée → répression OmpF et induction OmpC
faible → induction OmpF
de plus OmpF est régulé par des ARN régulateurs
Régulation du métabolisme secondaire chez les Pseudomonas
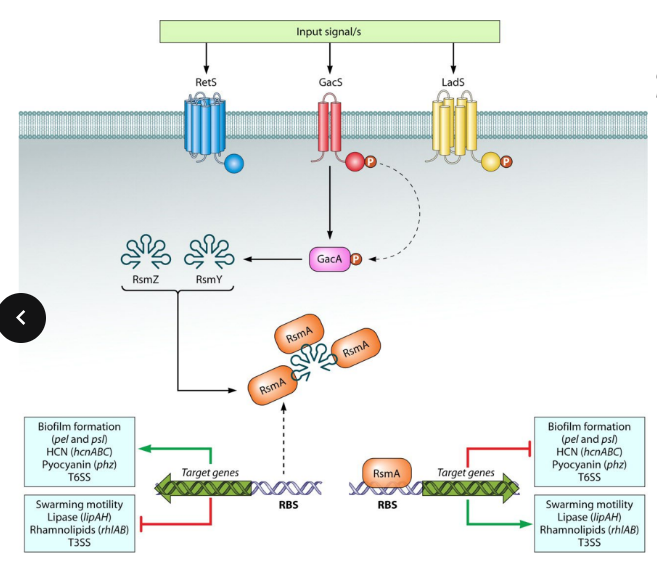
La régulation a lieu grâce aux GacA/GacS (Gac=global activator of secondary metabolites)
L’histidine kinase GacS régule un nombre important de gènes chez les Pseudomonas,
Une fois phosphorylé le régulateur GacA induit la production d’ARN régulateurs (RsmZ et RsmY— Rsm= repressor of secondary metabolites) qui séquestrent le répresseur RsmA ce qui conduit à l’expression des gènes impliqués dans:
les infections chroniques pour des pathogènes comme P. aeruginosa
la synthèse de composés antifongiques pour des souches associées aux plantes comme P. fluorescens
régulation par «quorum-sensing»
notion de quorum: assez pour décider du sort commun
les molécules résponsables de la signalisation dans les gram négatif sont basées sut les AHL (acyl-homoserine lactones) il y a ensuite un groupe reste qui varie
les molécules de signalisation des gram positif sont des oligopeptides autoinducers, il existe aussi une classe spéciale les streptomyces γ-butyrolactones
→ nous pouvons donc nous demander s’il y a une communication entre les espèces car les molécules ne se ressemblent pas tellement
découverte du quorum sensing
il y a une symbiose mutualiste entre une bactérie luminescente et un céphalopode, les bactéries colonisent le photophore de celui ci et c’est seulement lorsqu’elles sont en colonies qu’elles peuvent être luminescentes
mécanimse
les bactéries commencent à produire et à sécréter une petite molécule signal appelée autoinducteur. Lorsque la concentration d'autoinducteur devient suffisamment élevée, le complexe autoinducteur-récepteur active la transcription de gènes spécifiques, dont ceux responsables de la bioluminescence.
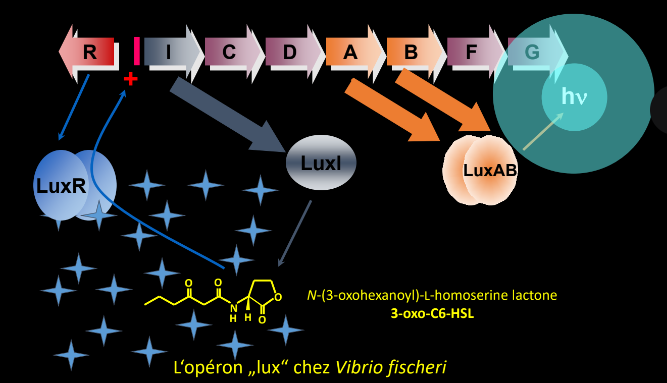
Les acteurs clés de la régulation de la luminescence :
LuxI: C'est une enzyme qui synthétise l'autoinducteur, une molécule de type acyl-homosérine lactone (AHL).
LuxR: C'est un facteur de transcription qui se lie à l'AHL. Le complexe LuxR-AHL se fixe sur une séquence spécifique de l'ADN en amont de l'opéron lux, activant ainsi la transcription des gènes de la bioluminescence.
Opéron lux: Cet opéron regroupe les gènes codant pour les enzymes nécessaires à la production de lumière (luciférase, substrat, etc.).
exemples de phénotype régulés par quorum sensing
réguler la production de facteurs de virulence, c'est-à-dire des molécules qui leur permettent d'infecter et de nuire à leur hôte. Par exemple, Pseudomonas aeruginosa utilise ce mécanisme pour former des biofilms dans les poumons des patients atteints de mucoviscidose.
synchroniser la conjugaison, un processus de transfert de matériel génétique d'une bactérie à une autre.
réguler la production de molécules antimicrobiennes et de sidérophores
coordonner leur mouvement et former des colonies + structure complexe dans les biofilms
La pourriture de l'oignon: La bactérie sauvage provoque une pourriture plus importante que le mutant déficient en quorum sensing.
La production de protéases plus élévé chez les sauvages
motilité augmenté chez les sauvages
formation de biofilms, biofilms plus denses chez les bactéries sauvages
production de fongicides et antibiotiques
invasion des nématodes, les bactéries sauvages peuvent infecter et tuer des nématodes
→diversité de réponses:
production de molécules, la formation de biofilms, la motilité, la virulence, la symbiose, etc.
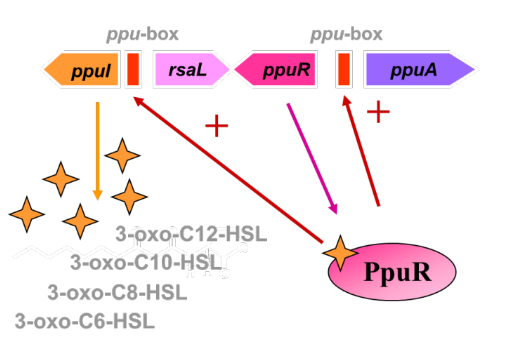
Régulation de la motilité par QS chez Pseudomonas putida IsoF
QS: quorum sensing
un frament d’ADN régulé par QS à plusieur région. il porte des gènes qui sont justement régulé par le QS (ppul et ppuA) et des gènes impliqué dans la régulation (rsal et ppuR)
PpuR actrive la transciption de ppuI et ppuA et ppuI produit des AHL, molécules signales
Le système de QS chez les Burkholderia
éléments clés:
cepI — Ce gène code pour une enzyme qui synthétise une molécule signal spécifique,
cepR — Ce gène code pour un régulateur transcriptionnel qui se lie à des séquences spécifiques d'ADN appelées "cep box".
cep box — Ce sont des séquences d'ADN situées en amont des gènes régulés par CepR.
→ quand la concentration en molécule signal augmente le CepR se lie aux cep box et active ou réprime l’expression de nombreux gènes impliqués dans différents processus biologiques
exemple d’effet de QS chez B. cenocepacia
La pourriture de l'oignon: La bactérie sauvage provoque une pourriture plus importante que le mutant déficient en quorum sensing.
La production de protéases plus élévé chez les sauvages
motilité augmenté chez les sauvages
formation de biofilms, biofilms plus denses chez les bactéries sauvages
production de fongicides et antibiotiques
invasion des nématodes, les bactéries sauvages peuvent infecter et tuer des nématodes
Régulation de la production de violacéine et de cyanure chez Chromobacterium violaceum
c’est une bactérie qui produit plusieurs antibiotiques dont la violacéine (potentiellement anti-cancéreuse) et le cyanure (HCN), la production de ces molécules est régulé par le QS →activation
les bactéries mutantes no sont pas capable de créer leur propre AHL mais elles y réagissent, on peut donc les utiliser pour tester la présence de AHL. (devient des biosenseur)
construction de biosenseur
Construction d’un plasmide où un promoteur régulé par les AHLs est fusionné à la GFP
Insertion de ce plasmide dans une souche sans QS (mutant spontané
ou construit)
Systèmes de QS chez P. aeruginosa
Plusieurs systèmes de QS: P. aeruginosa possède plusieurs systèmes de QS, chacun utilisant des molécules signal différentes (AHL) et régulant différents ensembles de gènes. Les principaux systèmes sont LasI/LasR et RhlI/RhlR.
+Régulateur orphelin: QscR est un régulateur transcriptionnel qui n'est pas directement lié à un système de synthèse d'AHL, mais qui interagit avec les autres systèmes de QS.
Synthèse de signaux non-AHL: La bactérie produit également des molécules de signalisation non-AHL, comme les PQS, qui jouent un rôle important dans la régulation de la virulence
Intégration avec d'autres systèmes de régulation globale: Le système de QS est étroitement lié à d'autres systèmes de régulation globale, comme le système GacA/GacS, la répression catabolique (Vfr) et la régulation en fonction de l'état énergétique (AlgR)
Hiérarchie et complexité: Les différents systèmes de QS et les autres systèmes de régulation interagissent de manière complexe, créant une hiérarchie de régulation. Par exemple, le système GacA/GacS active les systèmes LasI/LasR et RhlI/RhlR, tandis que LasR active la synthèse de PQS et réprime RhlI. → permet l’adaptation à l’environnement