REPLICATION DE L"ADN
1/68
Earn XP
Description and Tags
fini
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
69 Terms
Qu’est-ce que la phase G0 ? définitif ?
c’est la phase de veille qui peut soit etre
définitive, comme pour les neurones
ou transitoire jusqu’à ce qu’un autre stimuli fasse repartir la ç dans le cycle G1
Qu’est-ce que la phase G1 ? durée ?
phase de préparation qui dure en moyenne 10h
Qu’est-ce que la phase S ?
phase de réplication de l’ADN
Qu’est-ce que la phase G2 ?
phase d’attente d’un stimuli pour passer en mitose
qu’estc-e que le réplisome
=>permet la réplication de l’ADN pendant la phase S
un complexe protéique multienzymatique volumineux qui permet de transcrire extrêmement précisément et très rapidement =
Comment est cette réplication? comment est copié l’ADN ? (adjectifs)
elle est semi-conservative et basée sur la complémentarité
L’ADN est copié de manière complémentaire et antiparallèle, donc le néo-brin est dans le sens inverse de l’ancien brin
comment se déroule l’ouverture et la séparation en 2 brins de l’ADN ?
la structure de l’ADN étant très stable, il est nécessaire d’avoir une température très élevée → proche des 100° !!!!!
Les l° Hydrogène qui constituent l’hélice sont faibles quand on les regarde une à une donc il suffit de les séparer et cella se fait par des protéines spécialisées
comment s’appelle la séquence où l’ouverture va commencer ? il y en a combien dans le génome ?
c’est la séquence OR(=Origine de Réplication) sur un couple AT car il ne possède que 2 l°H alors que CG en possède 3 donc c’est plus facile d’ouvrir par là
Il en existe 10 000distribuées dans le génome, car l’ADN est très long donc s’il n’y avait qu’une seule ouverture la transcription sera super longue
mémo : OPEN THE DOR !!!
à quoi servent les séquences OR ?
à commencer à séparer l’ADN en plusieurs points simultanément → plus rapide
comment se passe le recrutement de l’hélicase ? quel est la condition à l’ouverture?
des protéines initiatrices s’accroche à un OR, puis recrute d’autres prot dont la 1ère est l’hélicase, qui ouvre/ dénature l’hélice en présence d’ATP
Au final, comment appelle on le site d’ouverture et de réplication de l’ADN ?
la fourche de réplication qu’on peut observer au ME. Il y en a deux qui sont de part et d’autre de l’OR
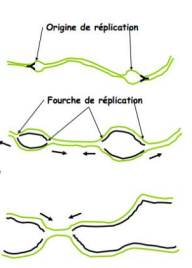
en quoi ce mécanisme est bidirectionnel ?
parce qu’il y a deux fourches qui avancent dans des sens opposés en répliquant l’ADN au fur et à mesure jusqu’au bout du K, en ajoutant les nouveaux brins complémentaires
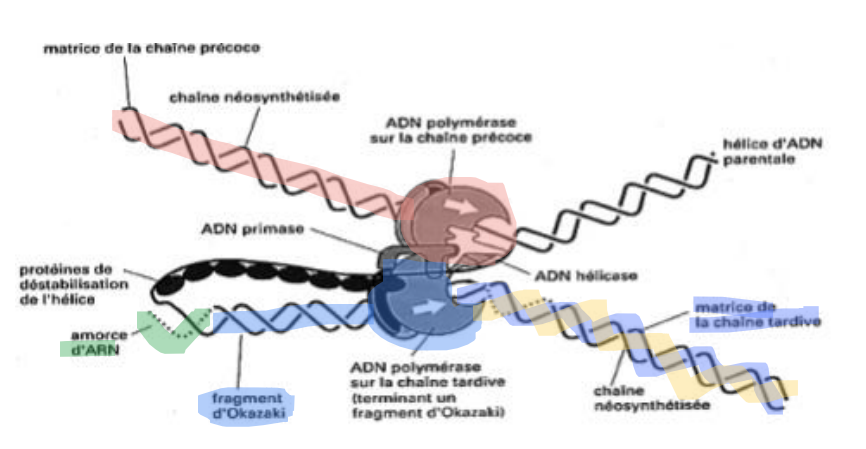
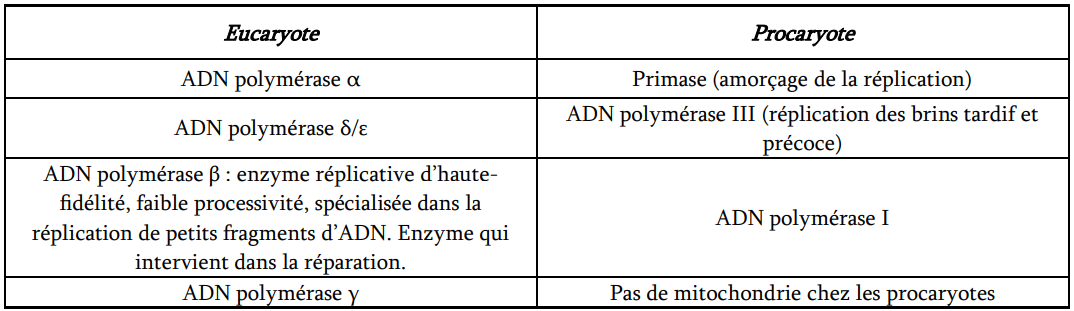
qu’est-ce qui catalyse la synthèse d’un nouveau brin chez les procaryotes? où est elle ?
c’est l’ADN polymérase III qui est ADN-dépendante pusiqu’elle catalyse la polymérisation d’ADN à partir d’une trame d’ADN ancien
elle se trouve à l’intérieur de la fourche de réplication
!!!!!L’ADN polymérase III n’existe que chez les procaryotes, donc essentiellement chez les bactéries.
Pol α, δ, ε = eucaryotes
que fait l’adn polymerase ? '(2choses)
elle synthétise le nouveau brin, et crée une l° phosphodiester entre le 5’P du nouveau nucléotide et le 3’OH de l’extrémité d’un brin en court de synthèse
→ ajout d’un nouveau nucléotide
comme si le brin père était les rails d’un train et qu’on ajoutait des wagons en les reliant ensemble
qu’est-ce qui est la seule direction de polymérisation possible
5’P → 3’OH
comment marche l’hydrolyse du nouveau nucléotide et quel est son but ? équation
l’hélicase a besoin d’NRJ =>
dNTP = désoxyribonucléotide + 3 phosphates (= pont triphosphate) =>il en existe 4 types : dATP, dTTP, dGTP, dCTP
Au moment de l’intégration :
HYDROLYSE : il ne reste qu’1 seul phosphate (Pα) → il forme la liaison phosphodiester
libération d’un pyrophosphate (=diphosphate)
HYDROLYSE : du pyrophosphate → hydrolysés en Pi = 2 phosphates inorganiques (HPO42-)
→ c’est cette hydrolyse qui fournit l’énergie (réaction irréversible)
ADN(n) + dNTP → ADN(n+1) + 2Pi
qu’est-ce que la processivité et pourquoi elle est élevée ?
c’est la capacité de l’ENZ à rester accrochée à la matrice (ancien brin) tout en polymérisant le nouveau. Elle est élevée sinon ça ne marcherait pas
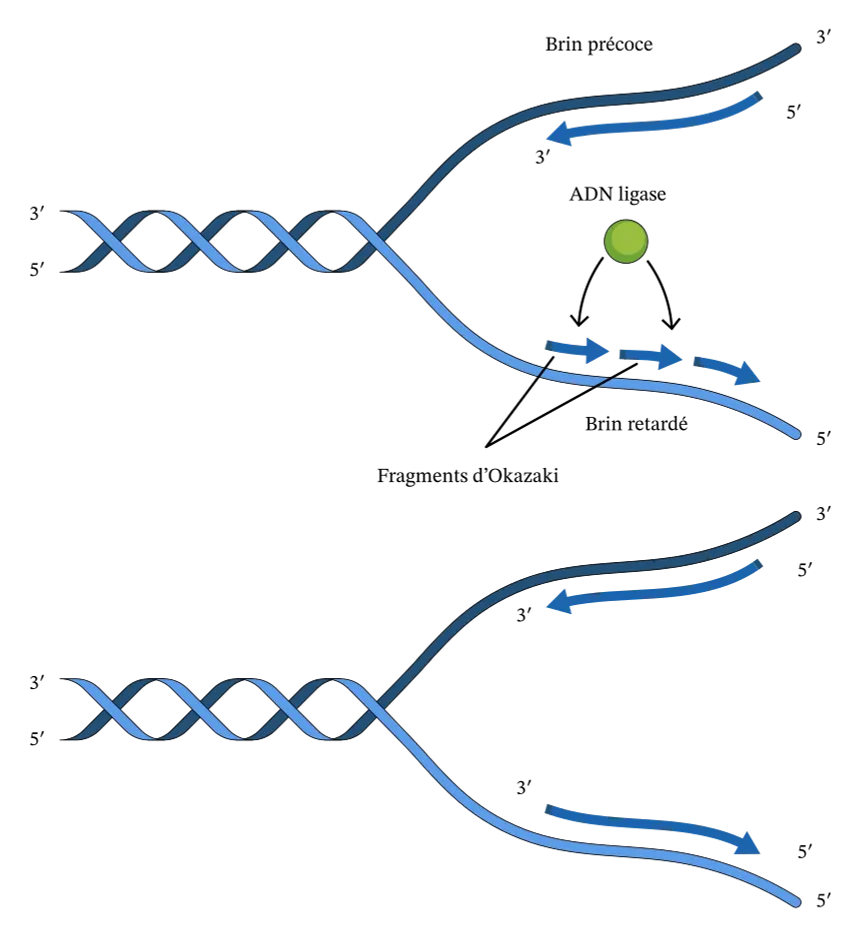
quelle est la solution pour la partie de la fourche qui n’est pas polymérisée dans le sens 5’P →3’OH ?
c’est portion est synthétisée par petits fragments = fragments d’Okazaki qui font environ 300 nucléotides et sont donc bien synthétisé dans le sens 3’→5’
à regarder à partir de 4min
où est le nucléotide le plus nouvellement synthétisé et celui le plus anciennement synthétisé ?
le plus nouvellement synthétisé est le premier nucléotide à l’origine de la fourche et le plus anciennement synthétisé est le nucléotide synthétisé juste après la fourche → le plus près de l’origine de réplication
quel est le brin continu et le brin discontinu ?
continu = précoce : sens de déplacement de la fourche
discontinu = tardif : fragments d’Okazaki
qu’est ce qui se passe quand il y a un mésappariement de base ?quelle est sa prévalence ?
environ une erreur tous les 105 nucléotides répliqués, ce qui est trop élevée pour la vie de la ç
donc l’ADN polymérase va passer sur le brin et vérifier l’appariement des nucléotides = activité 3’→5’ exonucléasique (hydrolysation)
comment ça se passe ? on aura une erreur tous les combien ?
elle passe , détecte une erreur, revient en arrière(3’→5’), hydrolyse le nucléotide pour en mettre un nouveau, et cela en même temps que de créer le brin fils
une erreur tous les 107 nucléotides répliqués
comment se fait l’amorce de la synthèse de l’ADN ? Comment peut on l’appeler ?
Grace à une primase, il va y avoir la formation d’environ 10 ribonucléotides (ARN) chez les bactéries, qui se fait dans le sens 5’→3’.
On peut l’appeler une ARN polymérase ADN dépendante !
quelle est la différence de la primase avec l’ADN polymérase ?
elle ne s’autocorrige pas donc elle peut commettre des erreurs, mais comme ce sont des séquence d’ARN elles pourront être lues et corrigées
Car l’amorce d’ARN est temporaire elle sera remplacée ensuite par de l’ADN correct
Et elle possède une faible processivité = capacité à synthétiser longtemps sans coder le brin (ce qui est l'ogique car elle ne va pas beaucoup synthétiser)
L’ADN polymérase III vient se fixer sur l’amorce nouvellement créée et qu’est-ce qui se passe pour le brin continu et le brin discontinu ?
continu : capable de polymériser au complet le brin fils
discontinu : il y a autant d’amorce en ARN que de fragment d’Okazaki donc 1 okazki = 1 amorce = 1 adn polymérase qui doit y passer
qu’est-ce que la “finition” ? attention knowt change le mot
c’est le fait de remplacer les amorces d’ARN par l’ADN fidèle puis soudure des brins discontinus, notamment les frang d’Okazaki
Qu’est-ce que la RNAse H ? quel est son rôle ?
c’est une enzyme qui va éliminer les amorces d’ARN en se fixant sur l’hétéro complexe ARN/ADN (un brin avec de ladn et un avec de larn) et laisse un trou, elle élimine donc potentiellement une erreur de l’ADN
qui resynthèse a partir des amorphes ? quelles sont ses caractéristiques ?
l’ADN polymérase I !!!!! 1
Elle est très efficace pour la réplication, mais possède une faible processivité = s’accroche pas longtemps au brin
Elle fait l’autocorrection et la réparation de l’ADN →allonge les frag d’Okazaki
(chatgpt)Elle produit de courts fragments d’ADN appelés fragments d’Okazaki, chacun débutant par une amorçe d’ARN (produite par l’ARN primase).
qui fait la soudure ? qu’est-ce que ça crée et où ?
c’est l’ADN ligase qui crée des l° phosphodiester 5’P→3’OH entre les frag d’ADN contigus
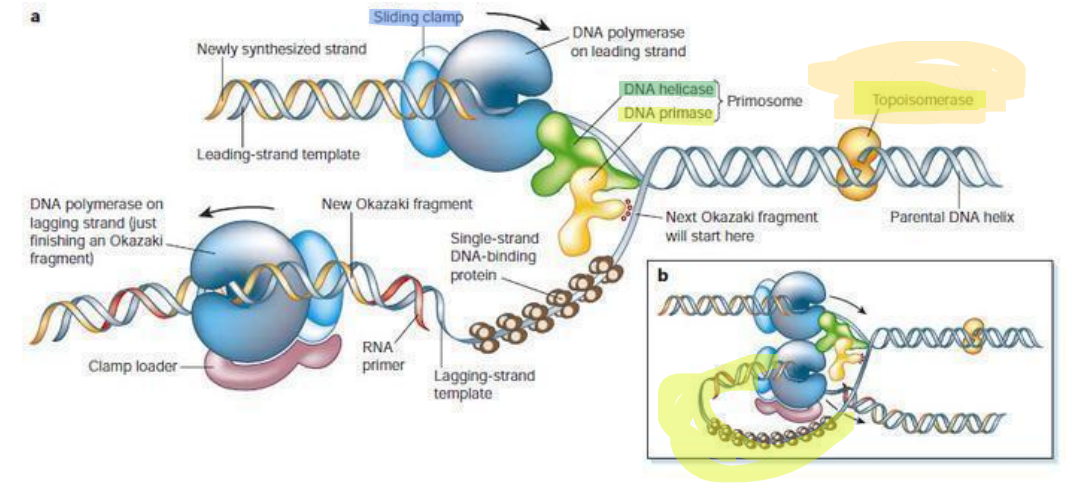
qu’est-ce que le réplisome ?
c’est un complexe multienzymatique multiprotéique volumineux localisé au niveau de la fourche.
il réplique l’ADN → synthése de deux brins dils complémentaire en sens inverse de manière simultanée
déplace la fourche
hélicase = + où elle est par rapport au réplisome
= celle qui ouvre la fourche
elle est à la tête du réplisome
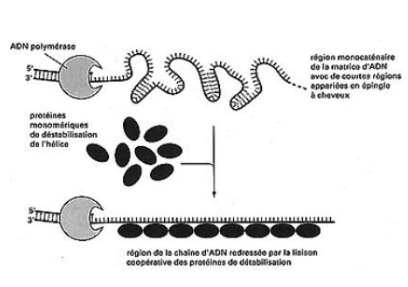
qu’est-ce que les protéine SSB (single String Blinding protéine)?
c’est une protéine de l° stabilisatrices de l’ADN simple brin des zones d’appariements ou structures doubles hélices
t’auras la fonction à la suivante tqt
En quoi l’SSB aide l’ADN après le passage de l’hélicase ?
il maintient l’ADN linéaire pour permettre le réappariements du brin ancien pour éviter les appariements intramoléculaire/intra-caténaires
+ elle favorise l’avancée de la l’ADN polymérase
c’est une réelle aide !
→évite que les deux brins se réapparient, et mal en plus de ça
mémo : sting qui fait une ligne bien droite de la ré des fesses
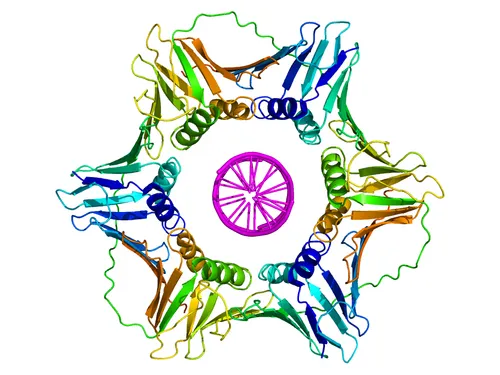
qu’est-ce que la protéine Clamp ? forme et constitution + rôle et lieu
clamp = collier
c’est la ssu B de l’ADN polymérase III qui est elle même constituée de deux sous unités qui s’associent pour former un collier qui fixe l’ADN polymérase au brin ancien.
Cela permet le bon déplacement de l’ADN polymérase tout du long de la traduction, lui donnant une vitesse et une processivité plus élevées
il existe un système de clampage et décalmpage pour les frag d’Okazaki
qu’est-ce que le clamptage/ déclamptage
Clampage (installation du β-clamp)
→ Le clamp loader place l’anneau β autour de l’ADN → la Pol III s’y accroche → réplication efficace.
Déclampage (retrait ou ouverture)
→ La Pol III se libère de l’anneau pour passer au fragment suivant.
✔ Brin avancé : 1 clampage
✔ Brin retardé : cycle clampage/déclampage à répétition → un par fragment d’Okazaki
comment doivent avancer les deux adn polymérase sur la fourche ?
il faut que les 2 ADN polymérases avancent dans le même sens, côte à côte, dans le sens du déplacement de la fourche de réplication
comment c’est possible mdr ?
le brin parental correspondant au futur brin tardif se retourne. L’ADN polymérase se trouve à côté de la primase. La polymérase ne se déplace pas, c’est l’ADN.
en quoi ça permet une réplication plus simple des fragments d’Okazaki par un système de clampage/déclampage de la sous-unité β
L’ADN polymérase III passe de l’extrémité du fragment d’Okazaki qu’elle vient de synthétiser à l’extrémité de l’amorce qu’elle va synthétiser
à quoi servent les topoisomérases ?
réalisent des coupures transitoires mono- ou bicaténaires. Elles réduisent ainsi les tensions que l’on trouve au sein de la double hélice, enlèvent les superenroulements / super tours de l’ADN et résolvent les problèmes d’enchevêtrement des chromosomes
→démêle le double brin qui coince
Topoisomérase de type 1 ? NRJ nécessaire ?
fait une coupure monocaténaire transitoire, réversible, elle ne nécessite pas d’ATP, ni d’énergie.
→Ceci permet la diminution des tensions à l’intérieur même de la double hélice en amont de la fourche de réplication. L’hélice tourne sur elle-même : Coupure monocaténaire ATP indépendante.
Lorsque la tension disparaît, les 2 brins se ressoudent entre eux (réaction réversible).
Topoisomérase de type 2 ? nrj?
fait une coupure bicaténaire, elle nécessite l’hydrolyse d’ATP. Cette action a pour objectif de résoudre les problèmes d’enchevêtrement de molécules dépendantes. L’ADN se reconstruit ensuite.
tout ce qu’on vient de voir c’est pour les PROCARYOTES
bilan de l’adn polymérase 3 chez les pro
Chez les procaryotes : l’ADN polymérase III est l’enzyme réplicative par excellence de la bactérie car elle a une haute processivité. Elle réalise la synthèse des brins précoce et tardif. Elle acquiert sa processivité par sa sous-unité β. La vitesse est élevée : 1000 nu/seconde. Elle a une activité d’autocorrection sur épreuve.
Bilan adn poly 1 chez les pro
L’ADN polymérase I réalise la synthèse de fragments d’environ 10 nucléotides :
- Remplace les fragments ribonucléotidiques par une courte séquence d’ADN d’environ 10 nucléotides.
- 3’→5’ : autocorrection de l’épreuve
- Comble les courtes brèches dans la réparation de l’ADN.
- Action d’autocorrection (donc activité exonucléasique).
- Intervient aussi dans les processus de réparation de l’ADN.
- Faible processivité : elle n’est pas capable de synthétiser un long brin d’ADN sans se détacher.
maintenant chez les EUCARYOTES
ADN polymérase α
4 sous-unités protéiques Elle est constituée de 2 sous-unités d’une primase et 2 sous-unités d’une polymérase → multimérique.
Elle présente une activité ADN et ARN polymérase donc 2 activités polymérasique, mais elle est dépourvue d’autocorrection sur épreuve (proof-reading), donc peut commettre des erreurs à un taux plus élevé. Elle est impliquée dans l’initiation de la synthèse du brin précoce et des nombreux fragments d’Okazaki du brin tardif, en effectuant d’abord la synthèse de l’amorce mixte ARN/ADN : ARN 10 ribonucléotides (ARN), puis de 20 à 30 désoxyribonucléotides (ADN). L’amorce est donc quelque peu différente des procaryotes.
quelle est l’activité de cette plymérase alpha ? autocorrection?
Elle présente une activité ADN et ARN polymérase donc 2 activités polymérasique, mais elle est dépourvue d’autocorrection sur épreuve (proof-reading), donc peut commettre des erreurs à un taux plus élevé.
quel est son rôle ? qu’est-ce qu’elle synthétise et ou
Elle est impliquée dans l’initiation de la synthèse du brin précoce et des nombreux fragments d’Okazaki du brin tardif, en effectuant d’abord la synthèse de l’amorce mixte ARN/ADN :
→ARN 10 ribonucléotides (ARN),
→puis de 20 à 30 désoxyribonucléotides (ADN).
L’amorce est donc quelque peu différente des procaryotes.
ADN polymérase δ et ε
Elles sont toutes les 2 de haute processivité, elles sont impliquées dans la synthèse des 2 brins tardif (ε) et précoce (δ) et ont comme rôle de polymériser la séquence de l’ADN à partir de l’amorce synthétisée par l’ADN polymérase α (→intervient après). Elles ont une activité d’autocorrection sur épreuve. Elles utilisent l’extrémité OH à nue pour polymériser.
L’ADN polymérase ε a la même fonction que l’ADN polymérase δ, c’est pour cela qu’on parle de l’ADN polymérase δ/ε.
ADN polymérase γ
Elle est transloquée dans la mitochondrie où elle est fonctionnelle : utilisée pour la réplication de l’ADN mitochondrial (ADN polymérase à activité réplicative).
tableau récap des diff entre PRO et EU
RP-A (Replicating Protein A)
Elle permet la correction des mésappariements faits par l’ADN polymérase α (erreurs de copies lors de l’amorçage pour la partie ADN de l’amorce).
mémo : Pasquier fait des fautes d’AuRtographe
RF-C (Replicating Factor C)
Elle est recrutée par le complexe amorce/matrice et intervient juste après RP-A. Complexe multimérique, activité ATPasique, qui recrute : PCNA.
PCNA (Proliferating Cell Nuclear Antigen)
Elle correspond à l’anneau de processivité (collier coulissant) de l’ADN polymérase 3 mais protéine indépendante, pas associée directement à une polymérase. Elle est recrutée par RF-C, se clampe sur l’ADN matrice. Elle recrute l’ADN polymérase δ/ε réplicative – facteur de processivité = confère une haute processivité à cette enzyme.
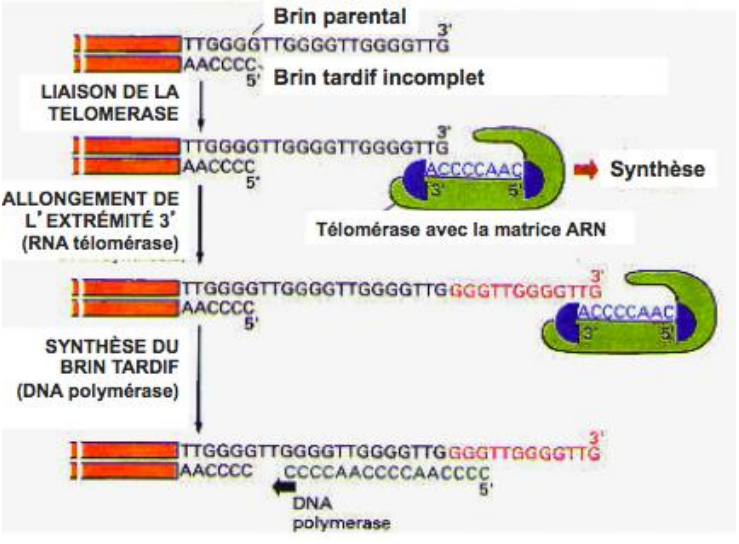
à quel problème sont associés les télomères
C’est le problème posé par la réplication de chromosomes linéaires(→eucaryotes).
Le brin continu est synthétisé jusqu’au bout mais cela n’est pas le cas du brin tardif.
Cela entraîne un raccourcissement des extrémités télomériques du brin tardif à chaque réplication car il n’y a pas d’extrémité 3’OH en amont sur laquelle peut se faire l’élongation qui remplace la séquence de l’amorce
quelle est la séquence des Télomères
séquence 5'-TTAGGG-3' (riche en G) aux extrémités des chromosomes
les chiffres sont pas à connaitre enfin bon c’est sympa quand même
que fait la télomérase ?
= 1 protéine +un ARN intégré =>ribonucléoprotéine
reconnaît ces fragments riches en G dont l’extrémité est 3’ sortante et se fixe au niveau de l’extrémité 3’ du brin parental.
→allonge le brin parental du côté 3’
comment est la matrice de la télomérase ?
Elle est dotée d’une matrice ARN intrinsèque (ribonucléoprotéine) complémentaire du motif hexamérique.
explication : (chat gpt)
L’ARN de la télomérase contient une petite séquence complémentaire au motif TTAGGG →AAUCCC
Cet ARN sert de modèle pour rallonger le brin parental en ajoutant plusieurs TTAGGG à la suite
=>ADN polymérase ARN dépendant →rétrotranscrit une séquence ARN en ADN
comment elle se déplace et pour faire quoi?
Elle se déplace d’une manière discontinue pour effectuer la copie d’environ 60 hexamères.
pourquoi l’extrémité des chromosomes est un minisatellite?
Le rallongement du brin parental 3’ forme un équilibre avec le raccourcissement du brin tardif.
qu’est-ce qu’un microsatellite ?
est une séquence d’ADN composée de motifs répétés en tandem
les télomères sont ils exprimés ? qu’ets-ce que ça implique ?
Est une protéine peu ou non exprimée dans les cellules somatiques adultes → raccourcissement progressif des télomères s à chaque réplication
qu’ests-ce qui se passe au bout de 40 div ?
dégénérescence et mort cellulaire
en quoi ça crée une horloge biologique ?
détermine le temps de survie d’une cellule : la survie des cellules somatiques dépend de leur passé réplicatif.
Permet aux cellules de ne pas vivre trop longtemps.
Mais les cellules à fort potentiel prolifératif (comme les lymphocytes en activité, les cellules souches et germinales) conservent une activité télomérase importante.
du coup, quelle est la particularité des cellules cancéreuses ?
elles ont toutes une activité élevée de télomérase donc elles expriment de manière importante cette télomérase
qu’estc-e qu’un role oncogène et pourquoi ?
Si la télomérase a une action trop élevée =rôle oncogène == un rôle qui pousse une cellule normale à devenir cancéreuse
et s’il n’y a pas de télomérase ?
il n’y a pas de développement de cancer car la cellule dégénère
Cela représente une stratégie thérapeutique :
Inhibition de l’activité télomérase.
Mais activité nécessaire à certaines cellules normales (souches, sexuelles, sanguines…), donc toxicité potentielle.
Molécules anti-télomérases à l’étude.
Cependant, il y a un mécanisme alternatif à la maintenance des télomères, différent de celui la donc inhiber la télomérase ne résoudrait pas tout.
Approches en cours d’évaluation clinique depuis 20 ans.