CM3 stabilité des ARN et transcription bactérienne
1/35
Earn XP
Description and Tags
stop diapo 46 (moi) arret diapo 56
Name | Mastery | Learn | Test | Matching | Spaced |
|---|
No study sessions yet.
36 Terms
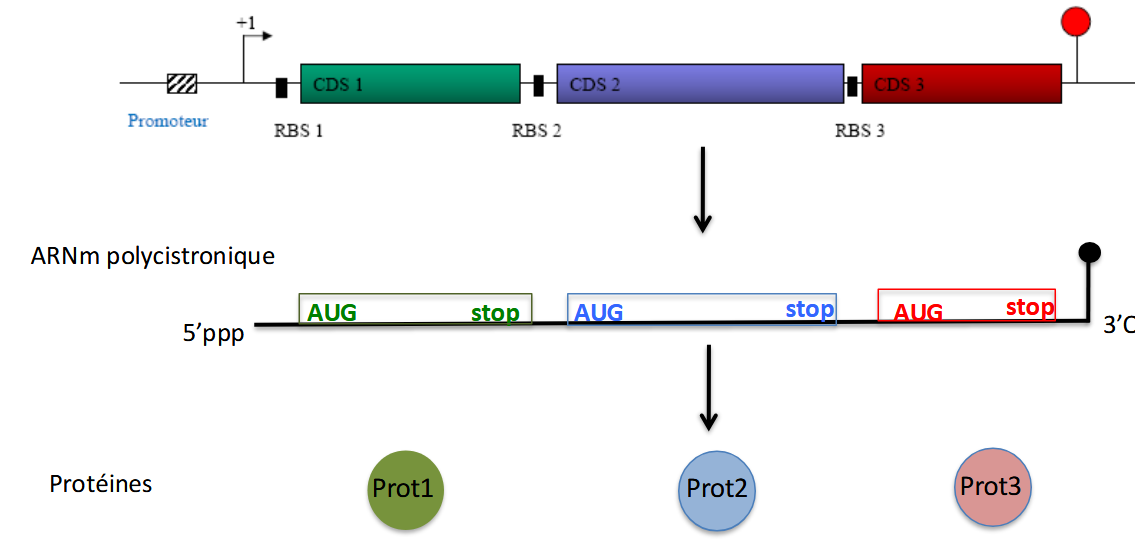
ARN polycistronique
ARN messager qui contient plusieurs cadres de lecture (cistrons) → donc plusieurs gènes.
👉 Un seul ARNm permet de produire plusieurs protéines différentes.
stabilité des ARNm polycistroniques
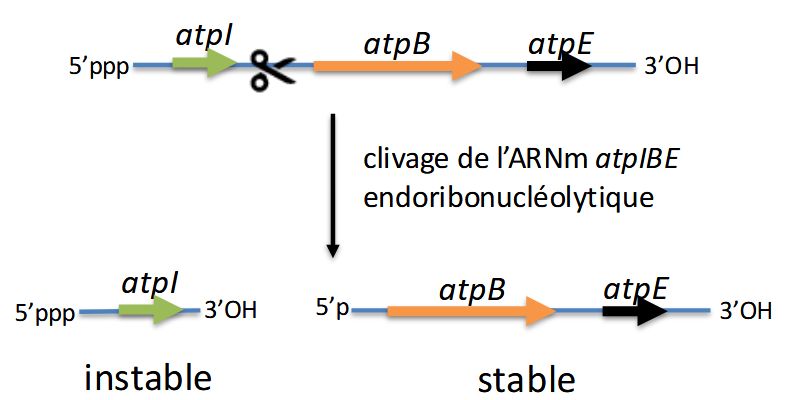
Dans un ARNm polycistronique, tous les gènes ne sont pas exprimés de manière équivalente.
Le clivage interne de l’ARNm peut :
désactiver une partie (ici atpI → instable),
mais stabiliser une autre partie (ici atpB-atpE → stable).
👉 Cela permet une régulation fine de l’expression des différents gènes d’un même opéron.
expression différentielle des cistrons
Chaque gène de l’ARNm polycistronique possède son propre site Shine-Dalgarno (RBS, ribosome binding site).
C’est la séquence où le ribosome s’aligne pour commencer la traduction.
Sans site Shine-Dalgarno → pas de traduction efficace.
L’efficacité de traduction dépend de :
La force du site Shine-Dalgarno (plus ou moins complémentaire à l’ARN 16S).
La structure secondaire de l’ARNm (si le RBS est masqué par une tige-boucle, le ribosome ne peut pas s’y fixer).
La position du cistron (souvent les gènes en 5’ sont plus traduits que ceux en 3’).
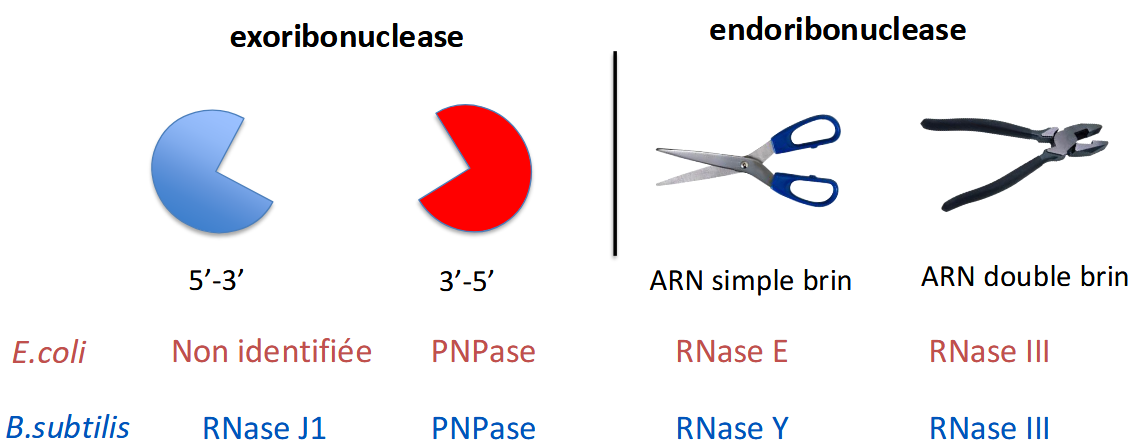
2 types de RNase bactériennes
Exoribonucléases
elles “mangent” l’ARN à partir d’une extrémité.
Elles avancent nucléotide par nucléotide.
Sens possibles :
5’ → 3’ (ex. RNase J chez certaines bactéries),
3’ → 5’ (ex. PNPase, RNase II).
Endoribonucléase
Fonctionnent comme des ciseaux ou pinces ✂ → elles coupent à l’intérieur de la molécule d’ARN.
Peuvent agir sur :
ARN simple brin (ex. RNase E),
ARN double brin (ex. RNase III).

Maturation des ARNt
À une extrémité (3’), il porte l’acide aminé correspondant (liaison ester).
À l’autre extrémité, il possède un anticodon qui reconnaît le codon de l’ARNm.
2. Chargement en acide aminé
Réalisé par les aminoacyl-ARNt synthétases (enzymes très spécifiques).
Chaque enzyme associe un ARNt donné à son acide aminé correct, grâce à l’ATP.
👉 On obtient un ARNt chargé (prêt pour la traduction).
3. Maturation par la RNase P
Les ARNt sont produits initialement sous forme pré-ARNt.
La RNase P coupe l’extrémité 5’ de ce précurseur pour générer un ARNt mature.
La RNase P est une ribonucléoprotéine :
rnpB (ARN) : activité catalytique.
RnpA (protéine) : rôle d’aide/structuration.
👉 Sans maturation correcte, l’ARNt ne peut pas être utilisé efficacement dans la traduction.

RNase P
riboenzyme très conservée (bactéries archées et eucaryotes)
Mature l’ARNt
La RNase P coupe l’extrémité 5’ de ce précurseur pour générer un ARNt mature.
La RNase P est une ribonucléoprotéine :
rnpB (ARN) : activité catalytique.
RnpA (protéine) : rôle d’aide/structuration.
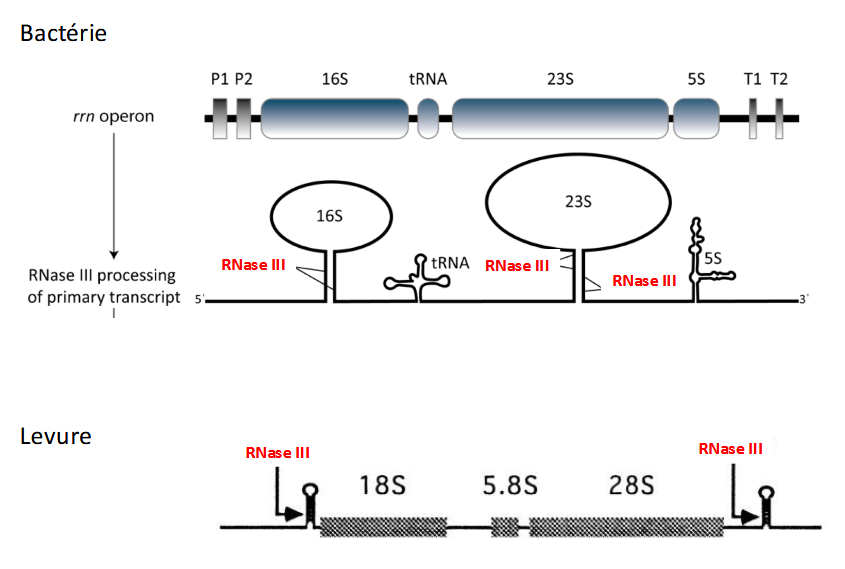
Maturation de l’ARNr
1. Organisation dans les bactéries
Les gènes codant pour les ARNr sont regroupés en opérons ribosomiques (rrn).
Chaque opéron contient des ARNr
2. Transcrit primaire
Produit par l’ARN polymérase → un long précurseur polycistronique contenant 16S, 23S, 5S et ARNt.
3. Maturation
Ce précurseur doit être clivé et modifié pour devenir fonctionnel.
Endoribonucléases (cMaturation des ARNs ribosomiques comme RNase III) clivent le précurseur.
Exoribonucléases affinent les extrémités 5’ et 3’.
Modifications chimiques (méthylations, pseudouridines) stabilisent la structure et facilitent l’assemblage.
👉 La maturation est donc essentielle pour la formation des ribosomes actifs, qui assurent la traduction.
Maturation des ARNm
Contrairement aux eucaryotes (où les ARNm subissent coiffe, épissage et queue poly-A), les ARNm bactériens sont déjà utilisables juste après transcription.
➡ Mais ils peuvent être clivés → Clivage endoribonucléolytique et stabilisés pour contrôler leur demi-vie et donc l’expression des gènes.

RNases majeures impliquées ds la dégradation des ARNm
(Ecoli et B subtilis)
dégradosome (bactérien)
complexe enzymatique central dans la dégradation des ARNm chez les bactéries.
👉 Fonction : couper et dégrader les ARNm de manière contrôlée, souvent du 5’ vers le 3’.

exosome (eucaryote)
complexe multiprotéique de dégradation de l’ARN que l’on trouve surtout chez les archées et les eucaryotes.
Il est fonctionnellement analogue au dégradosome bactérien, mais avec une organisation différente.
Les activités catalytiques sont assurées par des sous-unités ayant une activité exoribonucléase 3’ → 5’.

de quoi dépend l’accès des ARNm aux RNases
- La phosphorylation de l’extrémités 5’
→ protège de la dégradation
- La formation de structures secondaires
→ Des épingles à cheveux (structures en tige-boucle) peuvent bloquer ou ralentir l’accès des ribonucléases.
- La présence des ribosomes
→ Quand des ribosomes traduisent l’ARNm, ils protègent les régions codantes en recouvrant le brin. Les zones libres (UTR, après le STOP, ou non traduites) sont plus vulnérables.
Déphosphorylation de l’extrémité 5’ des ARNm et dégradation
La pyrophosphohydrolase déphosphoryle l’extrémité 5’ des ARNs en monoP et donnent accès aux RNases
(enlève 2 P sur les 3)

lien entre traduction et dégradation des ARNm
Quand les ribosomes traduisent l’ARNm efficacement:
Les ribosomes couvrent l’ARNm → cela le protège des RNases.
L’ARNm est donc stable.
Quand la traduction est moins efficace:
Moins de ribosomes se fixent.
Certaines régions de l’ARNm deviennent accessibles aux endoribonucléases (C2, C3, C4…).
L’ARNm est clivé et devient instable → dégradation plus rapide.
2 voix de dégradation des ARNm bactériens
en 5’ par dephosphorylation
par coupure endoribonucléique
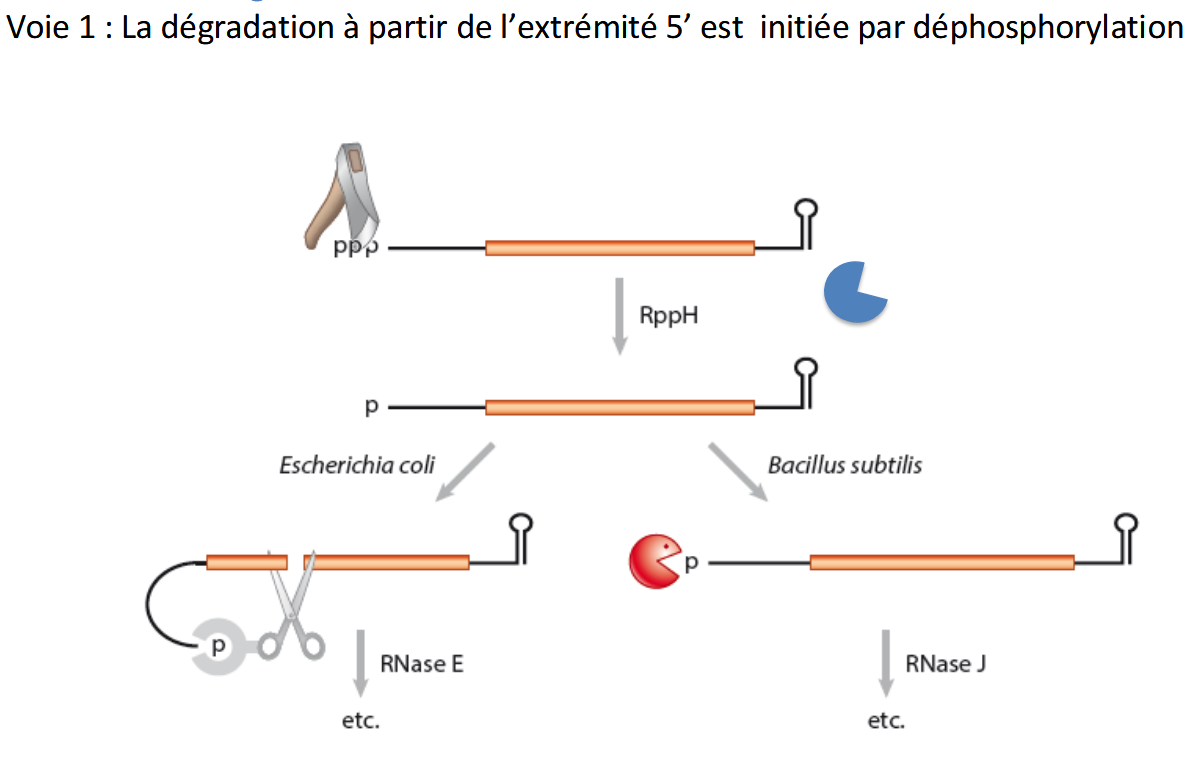
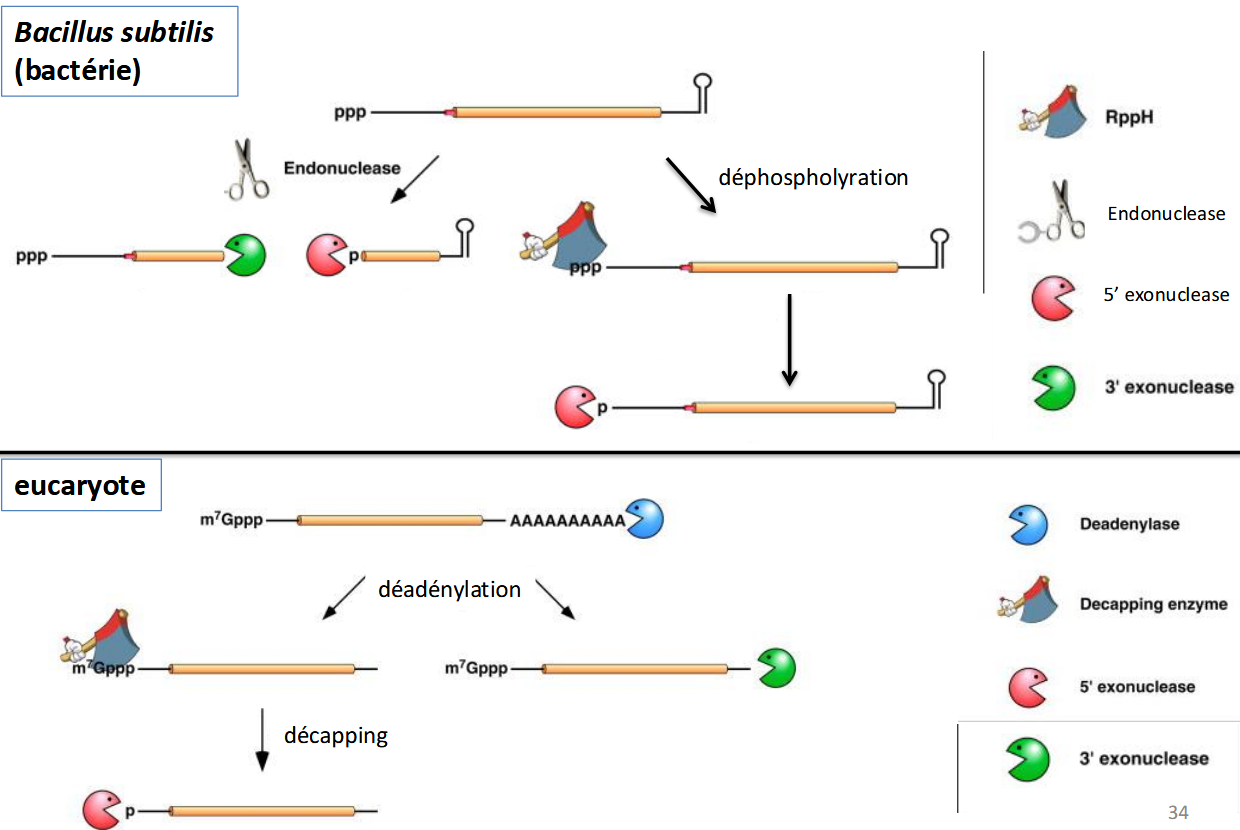
dégradation des ARNm par déphosphorylation
L’ARNm bactérien sort de la transcription avec une extrémité 5’ triphosphate (ppp).
Cette structure protège l’ARNm des RNases.
Déphosphorylation par RppH :
L’enzyme RppH enlève deux phosphates → l’extrémité devient un 5’ monophosphate (p).
À ce stade, l’ARNm devient vulnérable aux RNases.
Différences selon l’espèce :
Chez E. coli : le 5’ monophosphate est reconnu par RNase E, une endoribonucléase qui coupe à l’intérieur de l’ARNm → début de la dégradation.
Chez Bacillus subtilis : la dégradation se fait par une exoribonucléase 5’→3’, comme RNase J, qui ronge l’ARNm à partir du 5’.
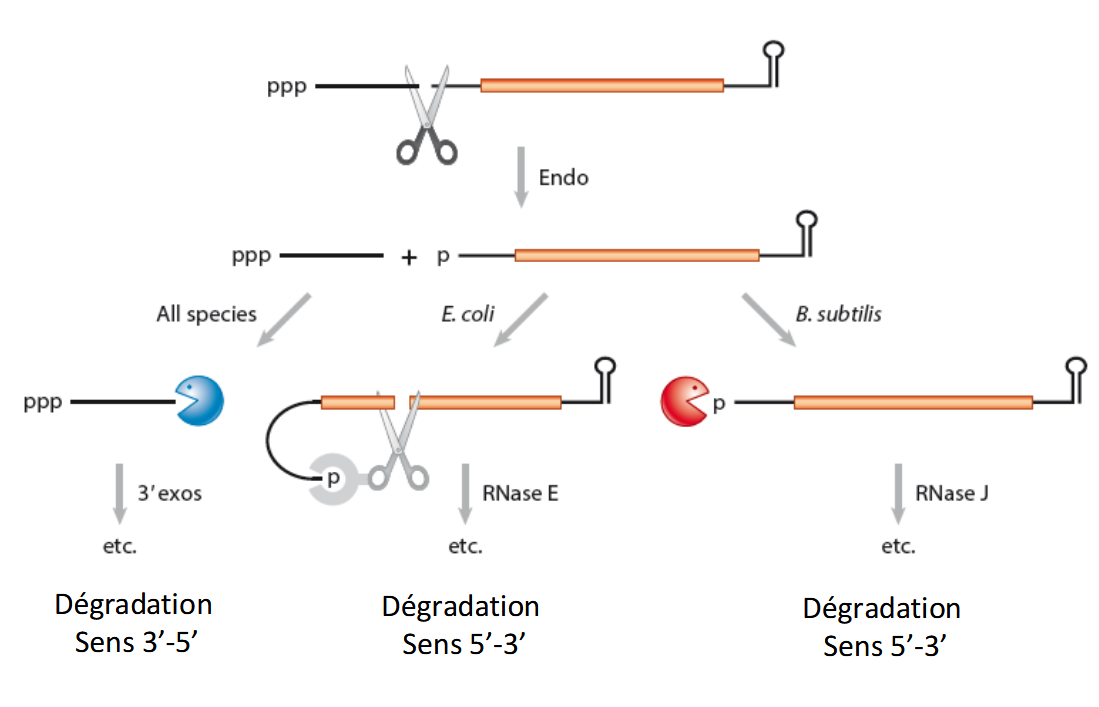
dégradation ARNm par courbure endoribonucléique
Une endoribonucléase (par ex. RNase E chez E. coli) coupe l’ARNm à l’intérieur de la séquence.
Cela crée deux fragments :
un fragment avec une extrémité 5’ monophosphate (p)
un fragment avec une extrémité 3’OH
Ces fragments deviennent instables et sensibles aux exoribonucléases.
2. Dégradation 3’ → 5’ (toutes espèces)
Les fragments terminés en 3’OH sont pris en charge par des exoribonucléases 3’ → 5’
3. Dégradation 5’ → 3’ (après déphosphorylation)
Après l’action de la RppH, l’ARNm passe de 5’ppp à 5’p.
Ensuite :
Chez E. coli → coupure interne par RNase E.
Chez B. subtilis → dégradation directe par RNase J (5’ → 3’ exo).
dégradation ARNm bactériens VS eucaryotes
Bactérie:
Départ : ARNm avec une extrémité 5’ triphosphate (ppp) → relativement stable.
Déphosphorylation (RppH) → conversion en 5’ monophosphate (p).
→ Cela rend l’ARNm sensible aux RNases.Clivage endoribonucléolytique (ciseaux, ex. RNase Y) → coupure interne de l’ARNm.
Dégradation par exoribonucléases :
5’ → 3’ (ex. RNase J)
3’ → 5’ (PNPase, RNase II, etc.).
👉 Ici, la déphosphorylation de l’extrémité 5’ est l’étape clé qui déclenche la dégradation
Eucaryote:
ARNm protégé par :
une coiffe m⁷Gppp à l’extrémité 5’
une queue polyA en 3’.
Ces structures protègent l’ARNm de la dégradation.
Déadénylation : raccourcissement progressif de la queue polyA.
→ l’ARNm devient instable.Deux voies possibles :
Décapping (retrait de la coiffe 5’ m⁷Gppp) → permet la dégradation 5’ → 3’ par Xrn1 (exonucléase).
Dégradation 3’ → 5’ par l’exosome après disparition de la queue polyA.
👉 Chez les eucaryotes, ce sont la coiffe 5’ et la queue polyA qui contrôlent la stabilité et la durée de vie des ARNm.
structure du ribosome
Site A (Aminoacyl) :
→ C’est là que l’ARNt chargé (avec son acide aminé) arrive.
→ Il reconnaît le codon correspondant de l’ARNm (par appariement anticodon-codon).Site P (Peptidyl) :
→ L’ARNt qui porte la chaîne peptidique en cours d’élongation s’y trouve.
→ C’est ici que se fait la formation de la nouvelle liaison peptidique grâce à l’activité peptidyl-transférase.Site E (Exit) :
→ L’ARNt qui vient de donner son acide aminé se déplace vers ce site.
→ Puis il quitte le ribosome, libre pour être rechargé par une aminoacyl-ARNt synthétase.Sous-unité petite (Small subunit) :
→ Elle s’occupe de la lecture de l’ARNm (décodage).Sous-unité grande (Large subunit) :
→ Elle catalyse la formation des liaisons peptidiques.

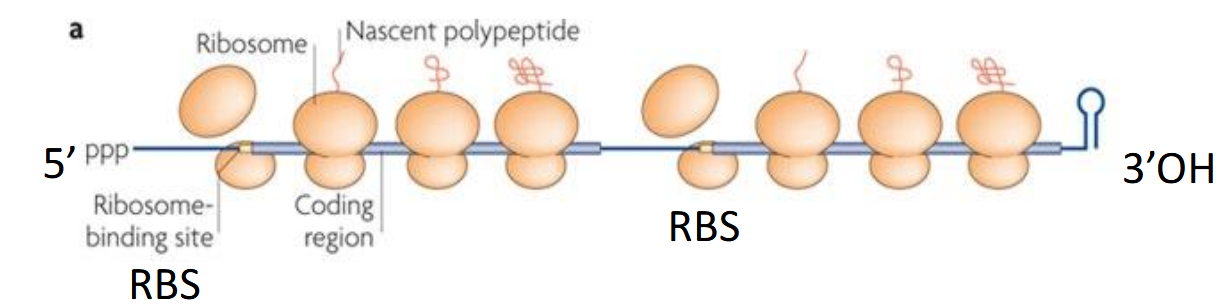
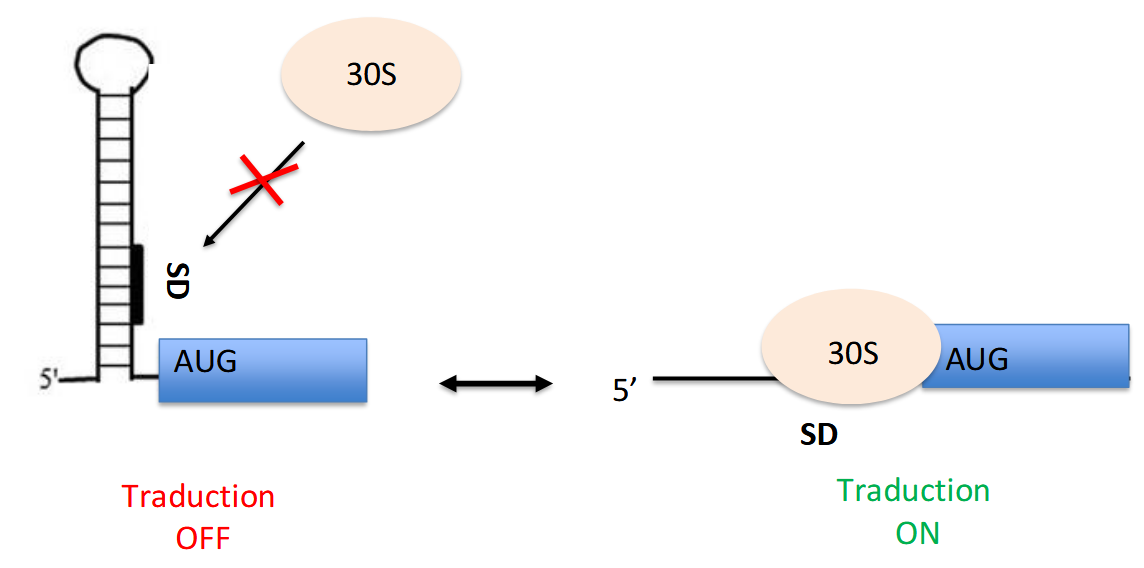
séquence Shine Dalgarno (RBS)
là ou se fixe le ribosome (30S) pour effectuer la traduction (+ codon initiateur)
si non accessible = ribosome se fixe pas = pas de traduction
facteurds protéiques de l’initiation de la traduction (bactéries)
IF1, IF2 et IF3
ARNt met
Chez les bactéries, le premier acide aminé incorporé est une méthionine modifiée : la formyl-méthionine (fMet).
Elle est apportée par un ARNt spécifique : ARNt^fMet.
Ce groupement formyl permet de distinguer la méthionine initiatrice des méthionines internes de la protéine.
Initiation de la traduction
La petite sous-unité 30S + facteurs d’initiation IF1, IF2 et IF3 se fixent d’abord sur l’ARNm au niveau de la séquence Shine-Dalgarno, proche du codon AUG.
L’ARNt^fMet s’apparie au codon AUG dans le site P.
Ensuite, la grande sous-unité 50S vient s’associer → formation du ribosome 70S actif
Après assemblage du complexe 70S, l’hydrolyse du GTP libère les IF → la traduction peut commencer.
élongation de la traduction
Reconnaissance du codon (site A)
Un ARNt chargé (avec son acide aminé) entre dans le site A du ribosome.
Son anticodon s’apparie avec le codon complémentaire de l’ARNm.
Formation de la liaison peptidique (site P → site A)
L’enzyme peptidyl-transférase (activité du 23S rRNA dans la grande sous-unité 50S) catalyse la formation de la liaison peptidique.
Le polypeptide en croissance passe de l’ARNt du site P à l’ARNt du site A.
Translocation
Le ribosome se déplace de 3 nucléotides vers l’extrémité 3’ de l’ARNm.
L’ARNt vide du site P glisse vers le site E (Exit) et sort.
L’ARNt porteur du peptide passe du site A → site P.
Le site A est à nouveau libre pour accueillir un nouvel ARNt.
👉 Ce cycle recommence jusqu’à ce qu’un codon stop arrive dans le site A, ce qui déclenche la terminaison.

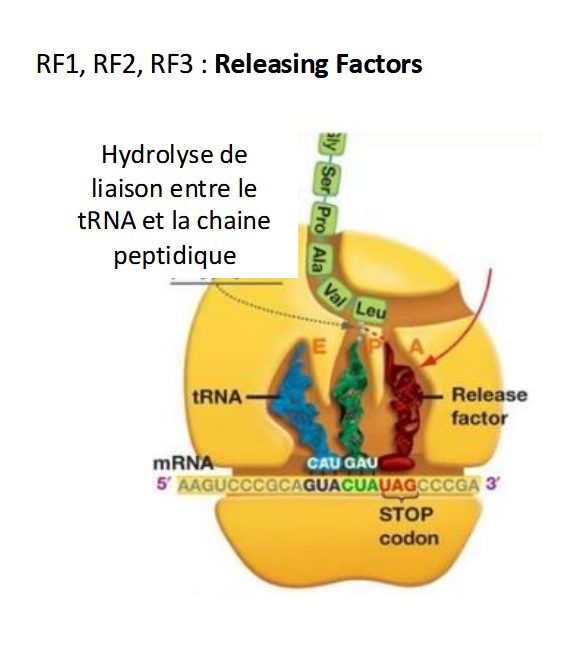
terminaison de la traduction
Arrivée du codon stop (UAA, UAG ou UGA)
→ Aucun ARNt ne correspond à ces codons.
→ À la place, un facteur de terminaison (Release Factor, RF) vient se fixer sur le site A du ribosome.
hydrolisent la liaison ARNt - chaine peptidique
libération du peptide

Realeasing factor
RF1, RF2 et RF3 hydrolisent la liaison ARNt - chaine peptidique
blocage de la traduction
pause ou arret du ribosome
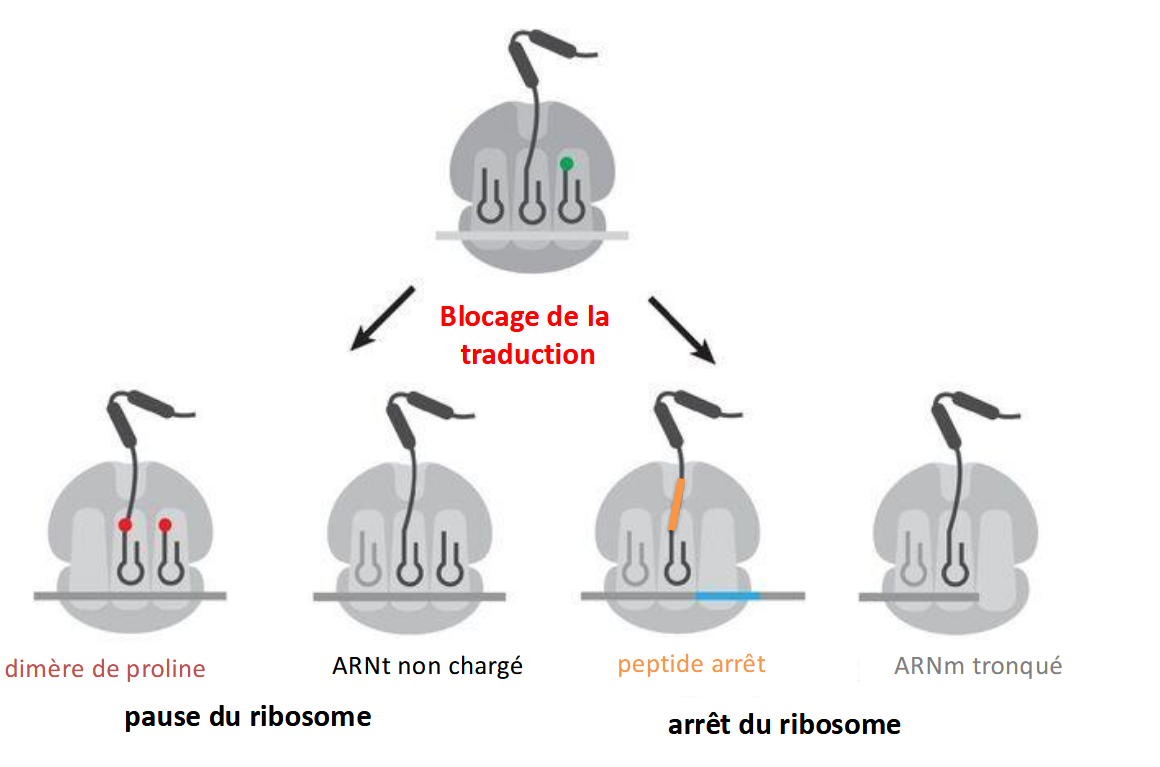
Pause de la traduction
dimère de proline
Arnt non chargé
arret du ribosome
peptide arret
ARNm tronqué
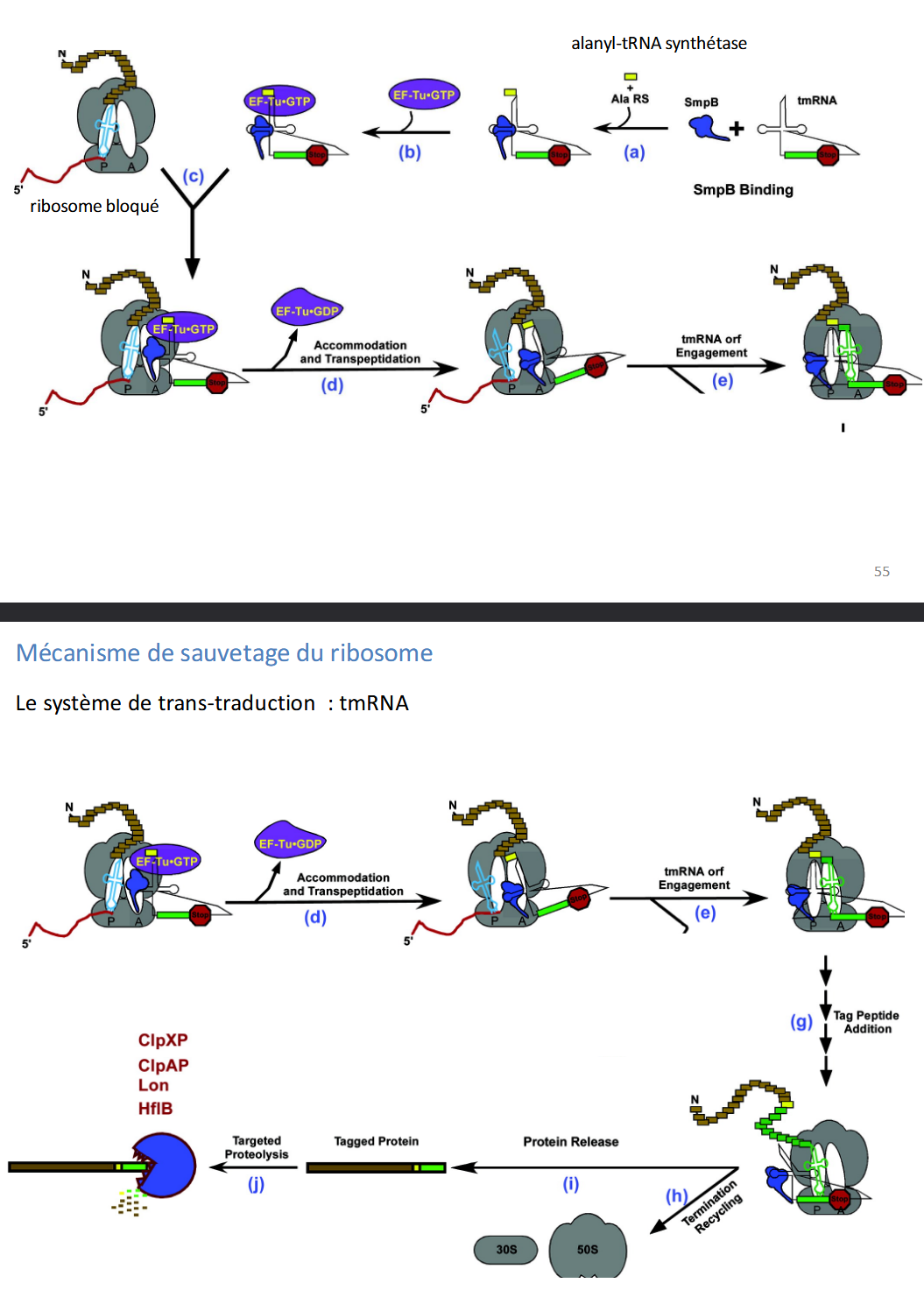
mécanisme sauvant les ribosomes bloqués
système de trans traduction
→ Parfois, la traduction s’arrête prématurément :
l’ARNm est tronqué (sans codon stop),
ou bien le ribosome reste bloqué à la fin de l’ARNm sans pouvoir se détacher.
👉 Résultat : le ribosome est bloqué avec un ARNt dans le site P, une chaîne peptidique incomplète, et aucun codon stop à traduire.
→ Le système fait intervenir :
tmRNA (ARN de transfert-messager) : hybride entre un ARNt et un petit ARNm,
SmpB : une protéine qui se lie au tmRNA pour le stabiliser et aider à sa reconnaissance par le ribosome.
système de trans traduction
(a) → (c)
Le tmRNA est chargé avec une alanine
Ce complexe s’associe à SmpB puis à EF-Tu-GTP pour former un complexe actif.
(d)
Ce complexe se fixe sur le ribosome bloqué, et le tmRNA entre dans le site A
→ La chaîne peptidique en cours est transférée sur l’alanine du tmRNA.
→ L’ARNm défectueux est rejeté.
(e) → (g)
Le tmRNA fournit alors
quelques codons codant un “peptide étiquette” (tag peptide),
suivi d’un codon stop.
👉 La traduction redémarre temporairement sur le tmRNA, jusqu’à ce codon stop.
(h) → (i)
La terminaison a lieu normalement
Le ribosome est recyclé, et la chaîne peptidique complète est libérée, désormais munie d’un “tag” à son extrémité C-terminale.
(j)
Cette étiquette (tag) sert de signal de dégradation :
les protéases ClpXP, ClpAP, Lon, HflB (FtsH) reconnaissent ce tag
→ la protéine incomplète est dégradée.
2 types de régulation post transcriptionelle
dégradation des ARNm
traduction
région 5’ UTR et régulation post transcriptionnelles
séquence située en amont du codon start (AUG), donc non traduite mais essentielle pour réguler l’expression du gène.
Elle contient :
la séquence Shine-Dalgarno (SD) : site de fixation du ribosome (sous-unité 30S)
des structures secondaires (tiges-boucles) capables de masquer ou révéler ce site
des sites de liaison pour des prot régulatrices
régulations post transcriptionnelles via la région 5’UTR (4)
1⃣ Thermosenseurs
Ce sont des structures d’ARN sensibles à la température.
À basse température, la région Shine-Dalgarno est bloquée dans une tige-boucle → traduction OFF.
Quand la température augmente, la structure se dénature, libérant le site SD → traduction ON.
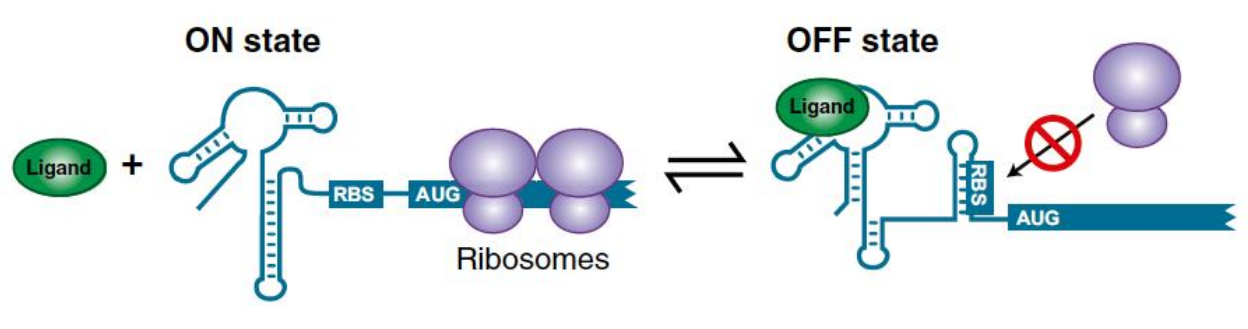
2⃣ Riboswitch
Ce sont des structures d’ARN capables de lier un métabolite spécifique (vitamine, cofacteur, etc.).
La liaison du ligand provoque un changement de conformation :
soit elle masque la SD → traduction inhibée,
soit elle la libère → traduction activée.
3⃣ Sites de liaison des ARN régulateurs (sRNA)
Des petits ARN non codants peuvent s’hybrider à la 5′ UTR :
Ils peuvent bloquer la SD (répression),
ou ouvrir la structure (activation).
4⃣ Sites de liaison des protéines régulatrices
Certaines protéines se fixent directement sur la 5′ UTR :
pour empêcher la fixation du ribosome (inhibition),
ou stabiliser l’ARNm (activation).
Riboswitch
Ce sont des structures d’ARN capables de lier un métabolite spécifique (vitamine, cofacteur, etc.).
La liaison du ligand provoque un changement de conformation :
soit elle masque la SD → traduction inhibée,
soit elle la libère → traduction activée.
👉 Exemple : riboswitch à la thiamine-pyrophosphate (TPP).
ex: thjim regulation chez E coli
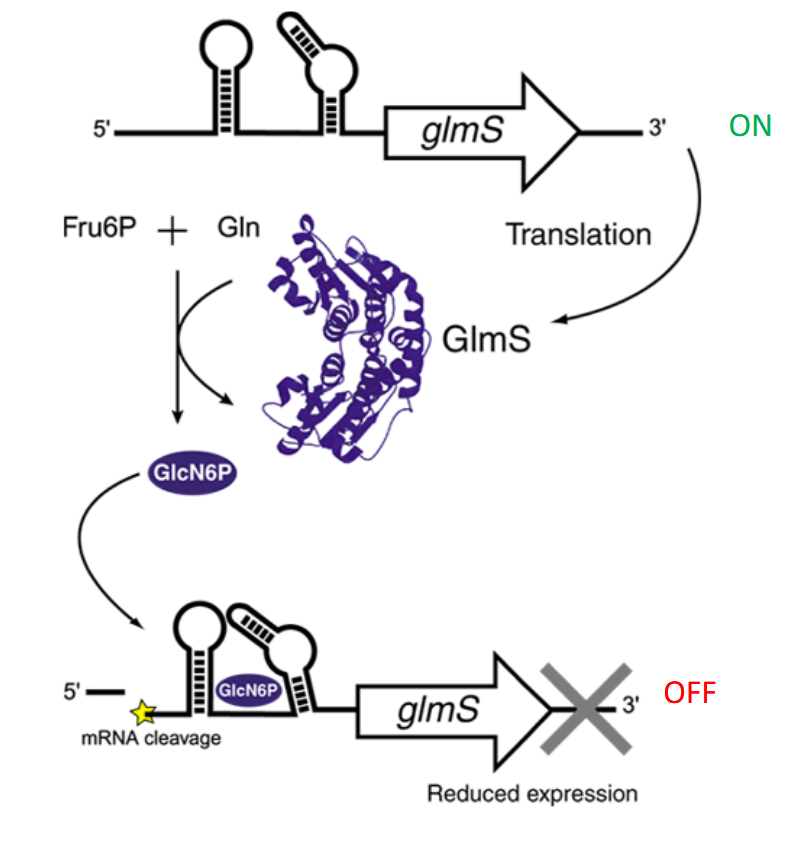
régulation en cis par le ribozyme de B.subtilis
Faible concentration de GlcN6P → gène ON
L’ARNm du gène glmS est transcrit normalement.
Il contient dans sa région 5′ UTR une structure ribozyme (un riboswitch catalytique).
En absence de GlcN6P, cette structure reste inactive.
➡ L’ARNm est stable → la traduction de GlmS a lieu → production de GlcN6P.
Forte concentration de GlcN6P → gène OFF
Quand le niveau intracellulaire de GlcN6P augmente, ce métabolite se lie directement au ribozyme présent dans l’extrémité 5′ de son propre ARNm.
Cette liaison active la fonction catalytique du ribozyme.
Le ribozyme clive l’ARNm (auto-coupure).
➡ L’ARNm devient instable → il est dégradé par les RNases → la traduction s’arrête.
C’est donc un mécanisme d’auto-rétro-contrôle négatif :